dhs <- read_csv("data/dhs.csv")Solucionario de ejercicios
Pipelines
Ejercicio 1
A) Escribir en lenguaje natural, que procedimientos se estan llevando a cabo en el siguiente codigo.
De la base de datos
dat, seleccionar las variables de interes (wealth_ind,literacy,educ_yrs,death_1m), luego filtrar aquellas observaciones con datos completos, luego agrupar los datos por categorias de nivel de ingreso y alfabetismo, luego calcular la mediana de años de educacion y el numero total de muertes neonatales en cada grupo y luego (finalmente) filtrar los grupos que tengan mas de 10 muertes neonatales.
B) ¿Que conclusiones podria sacar de este pipeline?
Los grupos de educacion e ingresos que tienen mas de 10 muertes neo-natales son los grupos desde el mas pobre hasta rico (1-4) que pueden leer almenos una oracion.
Ejercicio 2
A) Escribir el pipeline para la siguiente tarea de manejo de datos.
De la base de datos dhs, filtrar las observaciones de las madres menores de 20 años, luego remover todos las observaciones con valores ausentes (NA) en las variables edad, edad de pareja y seguro, luego calcular la diferencia entre la edad de la participante y la de su pareja, luego agrupar de acuerdo a si tienen seguro y acceso a agua potable segura, y luego calcular el promedio de hijos de las madres en cada una de estas categorias y la diferencia promedio de edad con sus parejas.
B) ¿Que conclusiones podria sacar de este pipeline?
El grupo de mujeres sin seguro de salud y sin agua potable segura, son aquellas con la menor diferencia de años con su pareja. El promedio de hijos es muy similar en las categorias de seguro de salud y acceso a agua potable segura.
ggplot
Ejercicio 1
A) Usando la base de datos who explorar la relación entre la inteisdad de emisiones de CO2 debido a actividades económicas (co2_economic_output) y malnutrición infantil (malnutrition_weight_age). Diferenciar los paises de acuerdo al continente (continent) donde se encuentran localizado
who <- read_csv("data/who.csv")
who %>%
ggplot(aes(x = co2_economic_output, y = malnutrition_weight_age,
col = continent)) +
geom_point()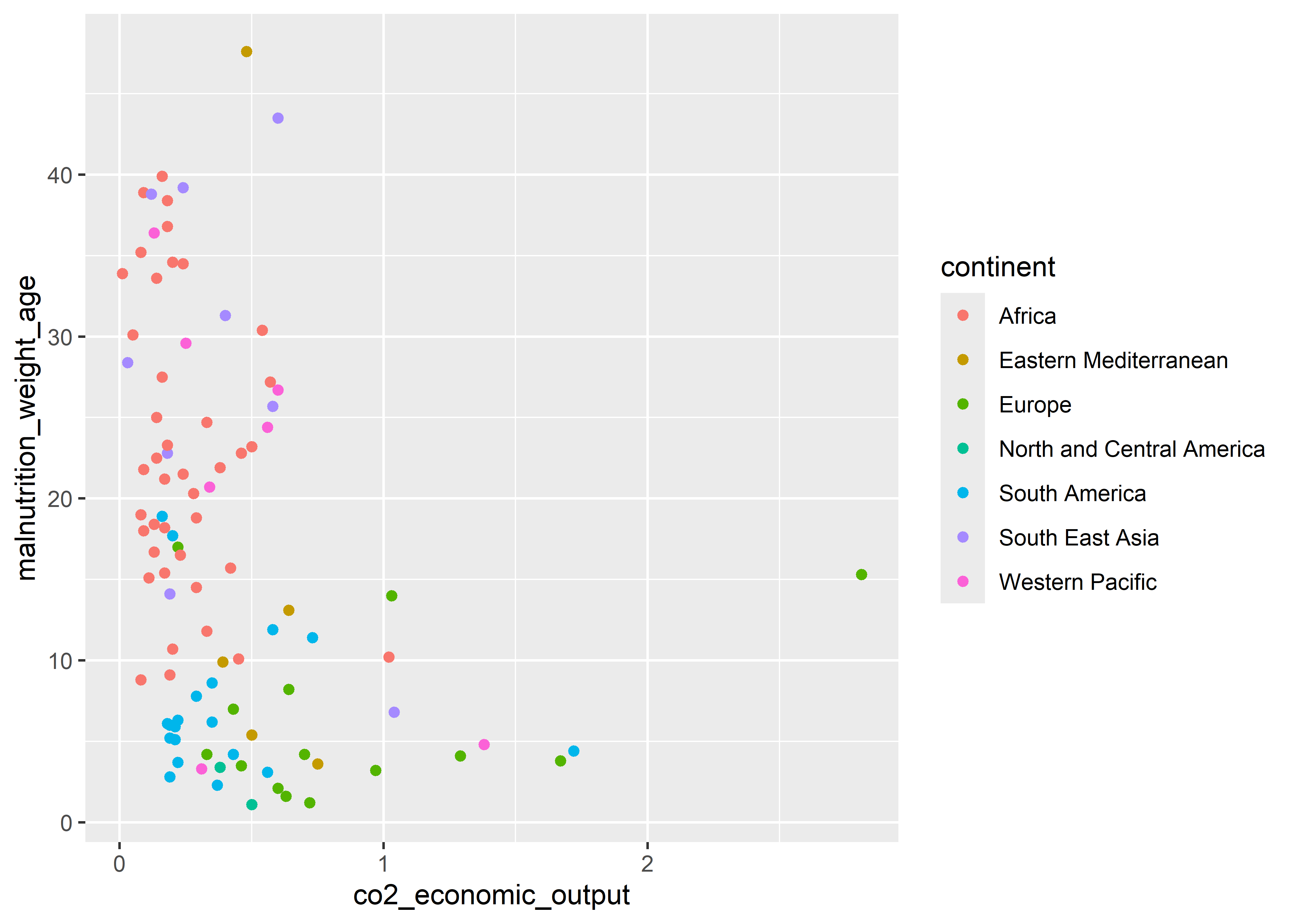
B) Que conclusiones podría sacar de este gráfico
Los paises del continente Africano tiene un perfil con alta malnutrición infantil y baja inteisdad de emisiones de CO2 debido a actuvidades económicas. Por lo contrario, los países Europeos muestran un perfil con baja malnutrición infantil y alta inteisdad de emisiones de CO2 debido a actividades económicas. Existe una tendencia de que aquellos paises donde hay mayor inteisdad de emisiones de CO2 debido a actividades económicas tiene una menor tasa de malnutrición infantil. No hay una estructura causal detrás de este análisis por lo que no se puede sugerir que la “manipulación” de alguna de estas variables generará cambios en la otra. En caso no exita suficiente información de alguna de malnutrición infantil, la inteisdad de emisiones de CO2 debido a actividades económicas podría ser un buen proxy.
Ejercicio 2
A) Reproducir el siguiente gráfico que muestra la diferencia en la distribución de camas hospitalarias (hospital_beds) en los diferentes quintiles de gasto en salud (contruir en base a la variable health_expenditure_gdp).
who %>%
drop_na(health_expenditure_gdp) %>%
mutate(cat_exp = cut_number(health_expenditure_gdp, 5)) %>%
ggplot(aes(x = hospital_beds, y = cat_exp)) +
geom_violin(fill = "gray") +
geom_point(aes(color = cat_exp)) +
theme_bw() +
scale_color_brewer(palette = "Reds") +
labs(x = "Camas Hospitalarias", y = "Quintiles de Gasto en Salud",
color = "Quintiles de Gasto en Salud")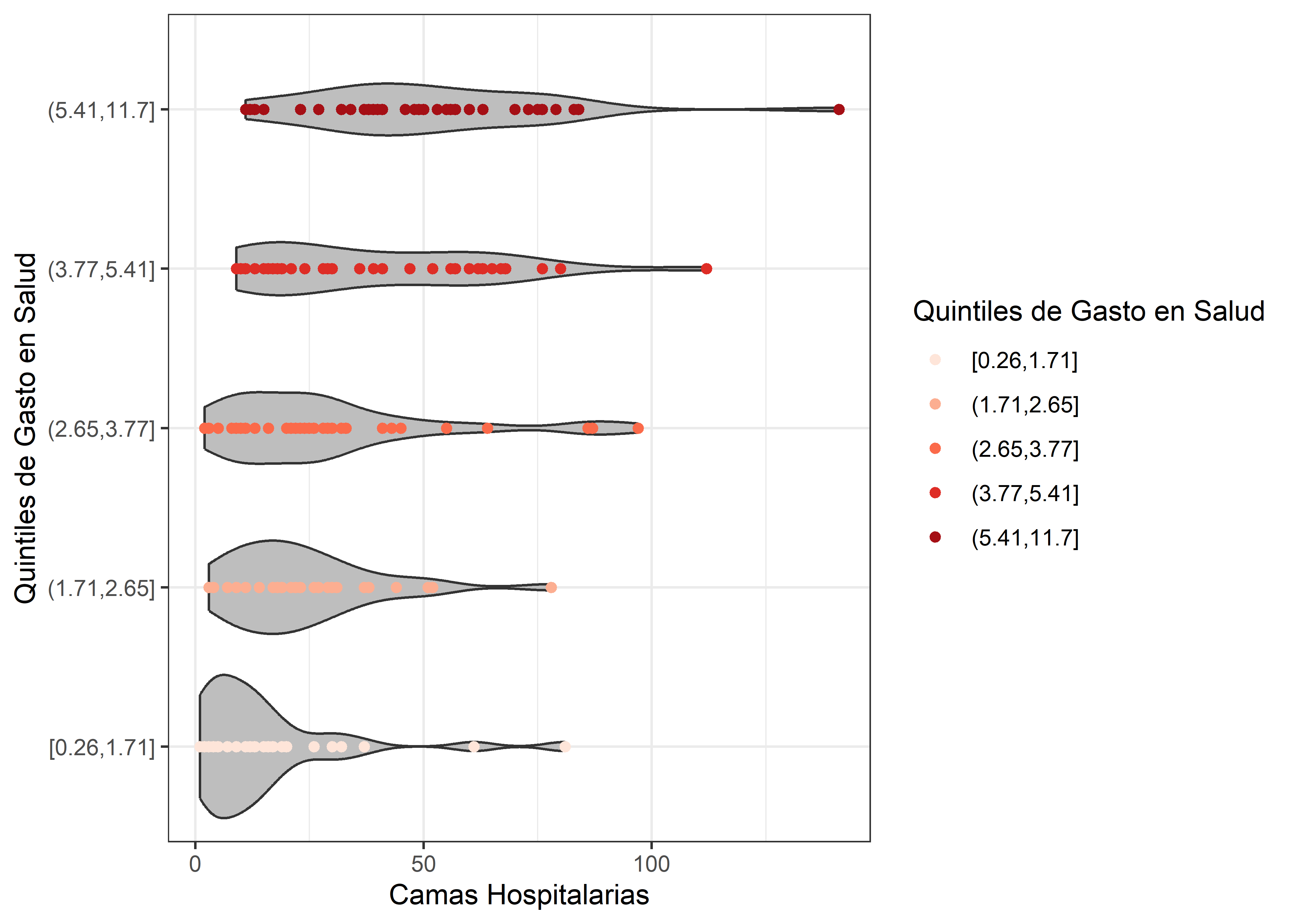
Analisis Exploratorio de Datos (AED)
Ejercicio 1
Realizar la exploración gráfica sobre la distribución de las variables consumo de energía (en_intake) y tiempo en dias (time). Además, calcule los valores de la media, mediana, simetría y curtosis. Utiliza la geometría geom_vline() para complementar los gráficos. Finalmente, sustente su conclusión sobre la forma de las distribuciones.
Para en_intake:
ratones %>%
ggplot(aes(x = en_intake)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(en_intake)), col = "red") +
geom_vline(aes(xintercept = median(en_intake)), col = "blue") +
geom_vline(aes(xintercept = Mode(en_intake)), col = "green")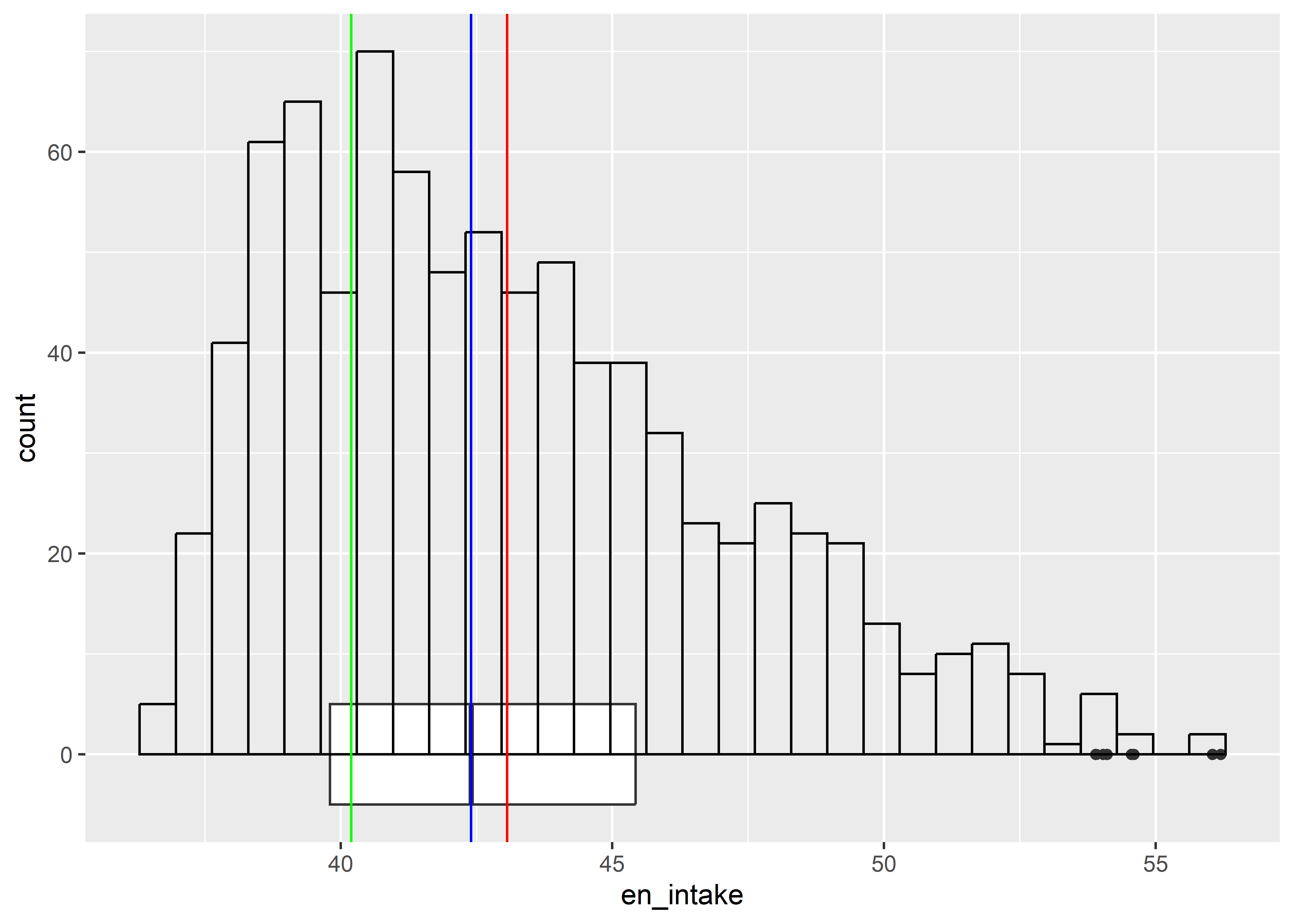
Para time:
ratones %>%
ggplot(aes(x = time)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(time)), col = "red") +
geom_vline(aes(xintercept = median(time)), col = "blue") +
geom_vline(aes(xintercept = Mode(time)), col = "green")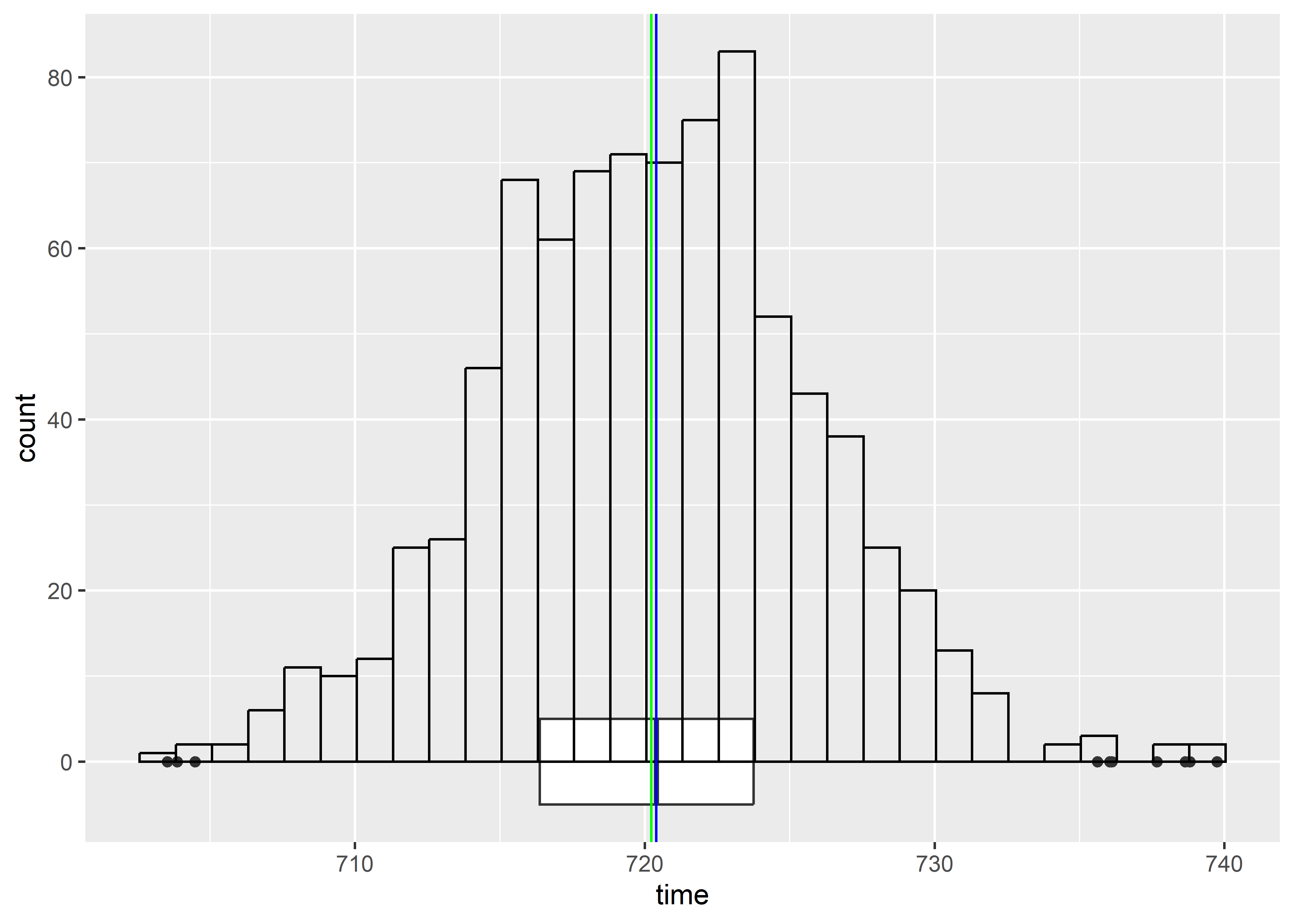
De acuerdo a la exploración gráfica y las diferentes medidas de tendencia central la variable
timetiene distribucion nomal y la variableen_intaketiene un sesgo a la derecha (positivo).
Ejercicio 2
Comparar la dispersión de las variables eficiencia metabólica (kJ/día por gramo de peso corporal) (meta_eff) y respuesta temprana a la insulina (pmol/l) (ein_resp) utilizando la(s) medida(s) de dispersión más adecuada(s).
Para meta_eff:
ratones %>%
ggplot(aes(x = meta_eff)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(meta_eff)), col = "red") +
geom_vline(aes(xintercept = median(meta_eff)), col = "blue") +
geom_vline(aes(xintercept = Mode(meta_eff)), col = "green")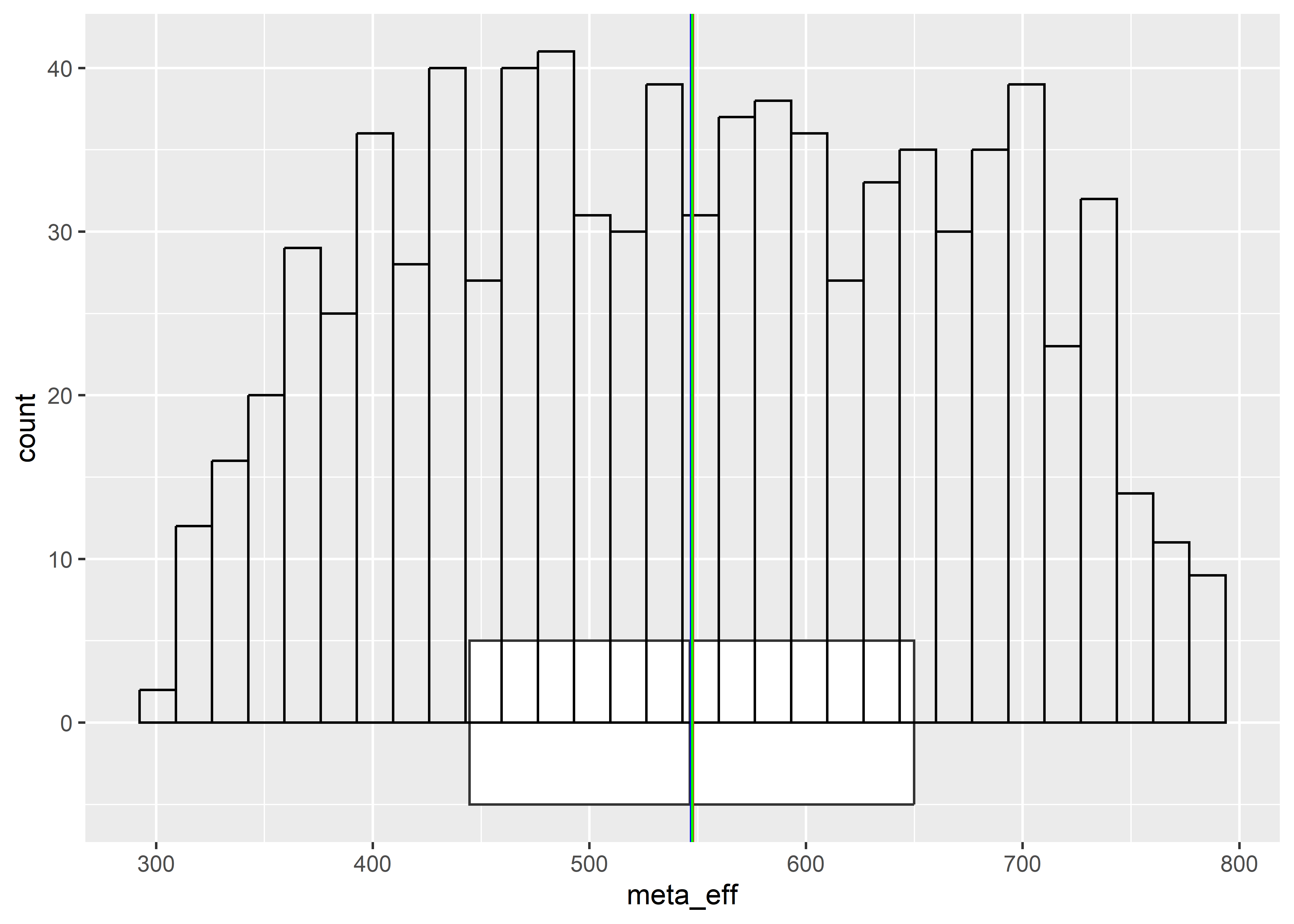
ratones %>%
estat(~ meta_eff) N Min Max Mean Median SD CV
1 meta_eff 846 307.74 792.26 547.78 546.88 123.09 0.22Para ein_resp:
ratones %>%
ggplot(aes(x = ein_resp)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(ein_resp)), col = "red") +
geom_vline(aes(xintercept = median(ein_resp)), col = "blue") +
geom_vline(aes(xintercept = Mode(ein_resp)), col = "green")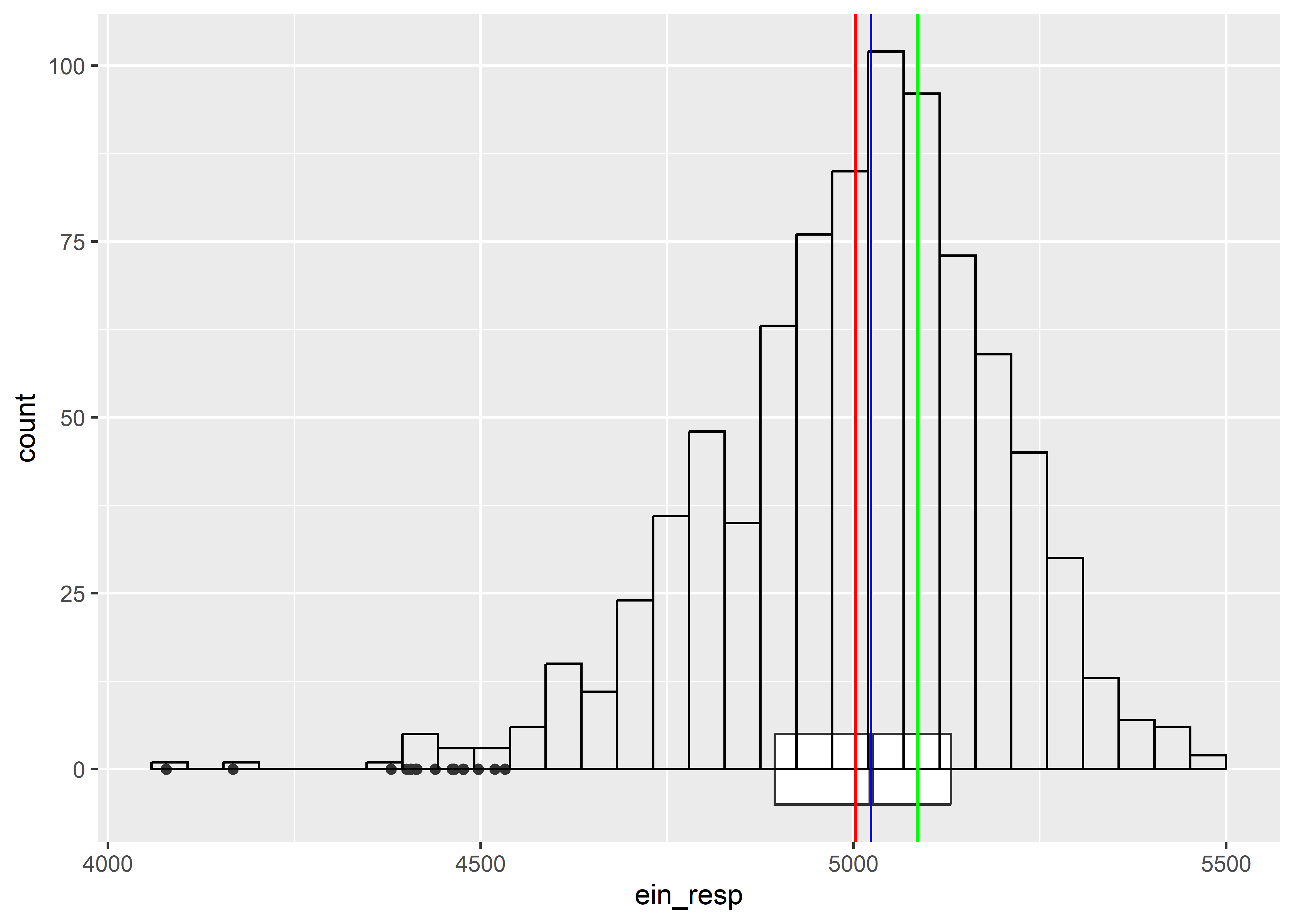
ratones %>%
estat(~ ein_resp) N Min Max Mean Median SD CV
1 ein_resp 846 4078.18 5471.18 5002.99 5023.54 191.3 0.04De acuerdo a la exploración gráfica la variable
meta_efftiene una mayor dispersion que la variableein_resp. Sin embargo, las medidas de dispersion tradicionales muestran queein_resptiene una mayordesviacion standard (sd)yvarianza (var)quemeta_eff. Estas mediciones estan influenciadas por la escala de ambas variables. Al calcular elcoeficiente de variacion (cv)se observa que el cvmeta_eff = 0.2247065>ein_resp = 0.03823714.
Ejercicio 3
A) Explorar y comparar en_intake y ein_respen términos de asimetría.
Para en_intake:
ratones %>%
ggplot(aes(x = en_intake)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(en_intake)), col = "red") +
geom_vline(aes(xintercept = median(en_intake)), col = "blue") +
geom_vline(aes(xintercept = Mode(en_intake)), col = "green")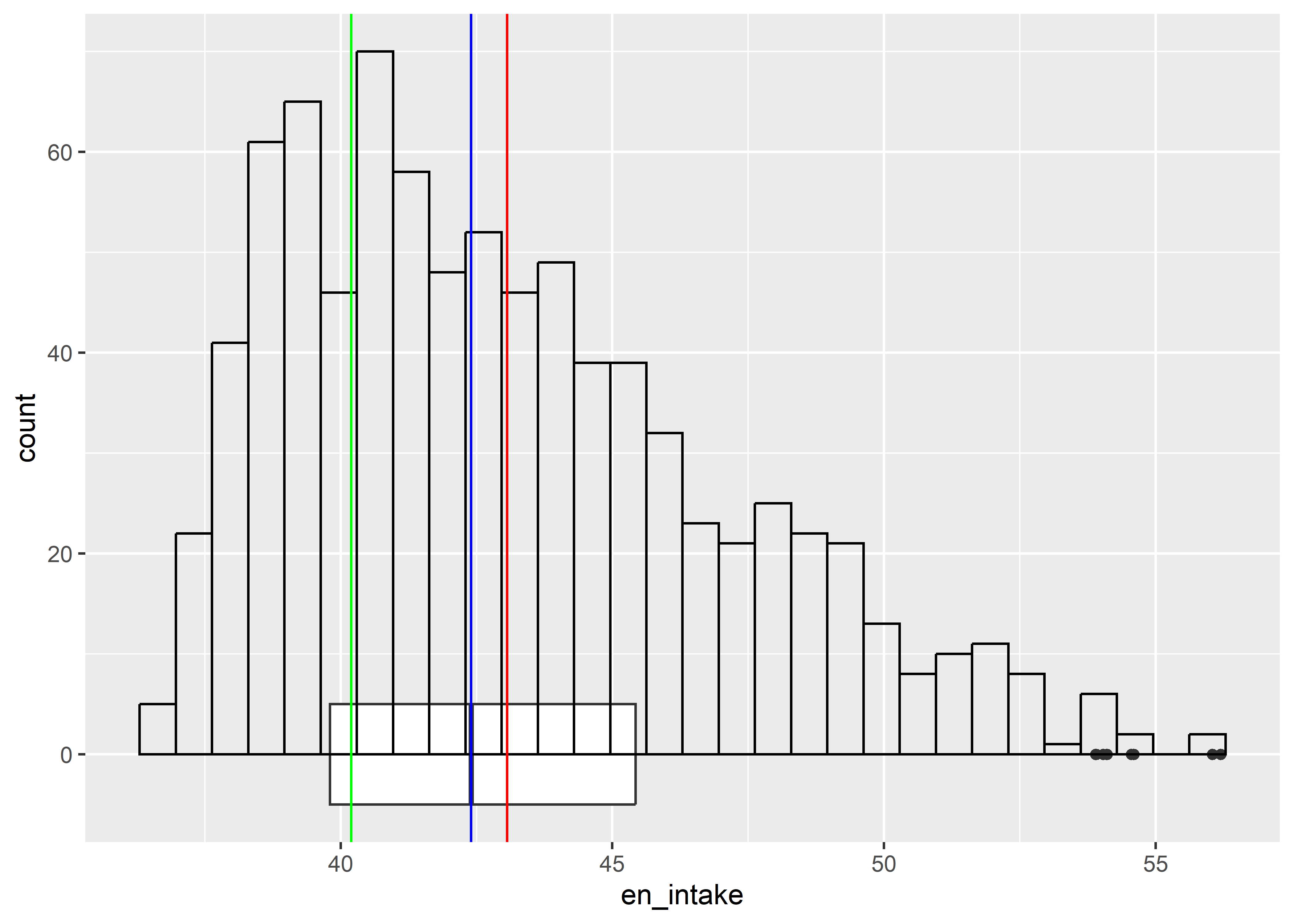
skewness(ratones$en_intake)[1] 0.7060313Para ein_resp:
ratones %>%
ggplot(aes(x = ein_resp)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(ein_resp)), col = "red") +
geom_vline(aes(xintercept = median(ein_resp)), col = "blue") +
geom_vline(aes(xintercept = Mode(ein_resp)), col = "green")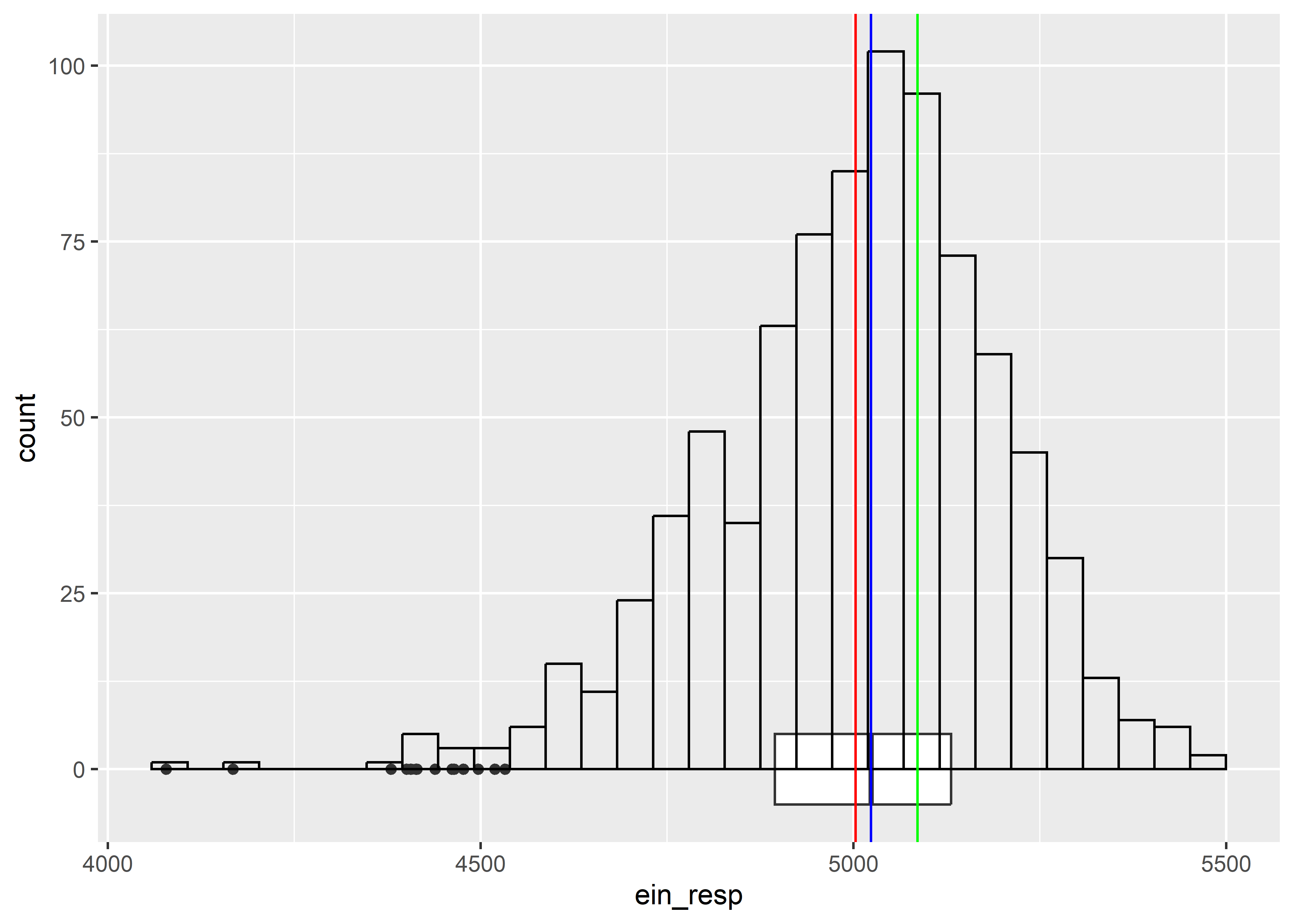
skewness(ratones$ein_resp)[1] -0.6105134De acuerdo a la inspeccion grafica la variable
en_intaketiene un sesgo a la derecha (positivo) [Moda<Mediana<Media]. Por otro lado, la variableein_resptiene un sesgo a la izquierda (negativo) [Moda>Mediana>Media].
B) Explorar y comparar insulin y meta_effen términos de curtosis.
Para insulin:
ratones %>%
ggplot(aes(x = insulin)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(insulin)), col = "red") +
geom_vline(aes(xintercept = median(insulin)), col = "blue") +
geom_vline(aes(xintercept = Mode(insulin)), col = "green")
kurtosis(ratones$insulin)[1] 4.090441Para meta_eff:
ratones %>%
ggplot(aes(x = meta_eff)) +
geom_boxplot(width = 10) +
geom_histogram(fill="transparent", col = "black") +
geom_vline(aes(xintercept = mean(meta_eff)), col = "red") +
geom_vline(aes(xintercept = median(meta_eff)), col = "blue") +
geom_vline(aes(xintercept = Mode(meta_eff)), col = "green")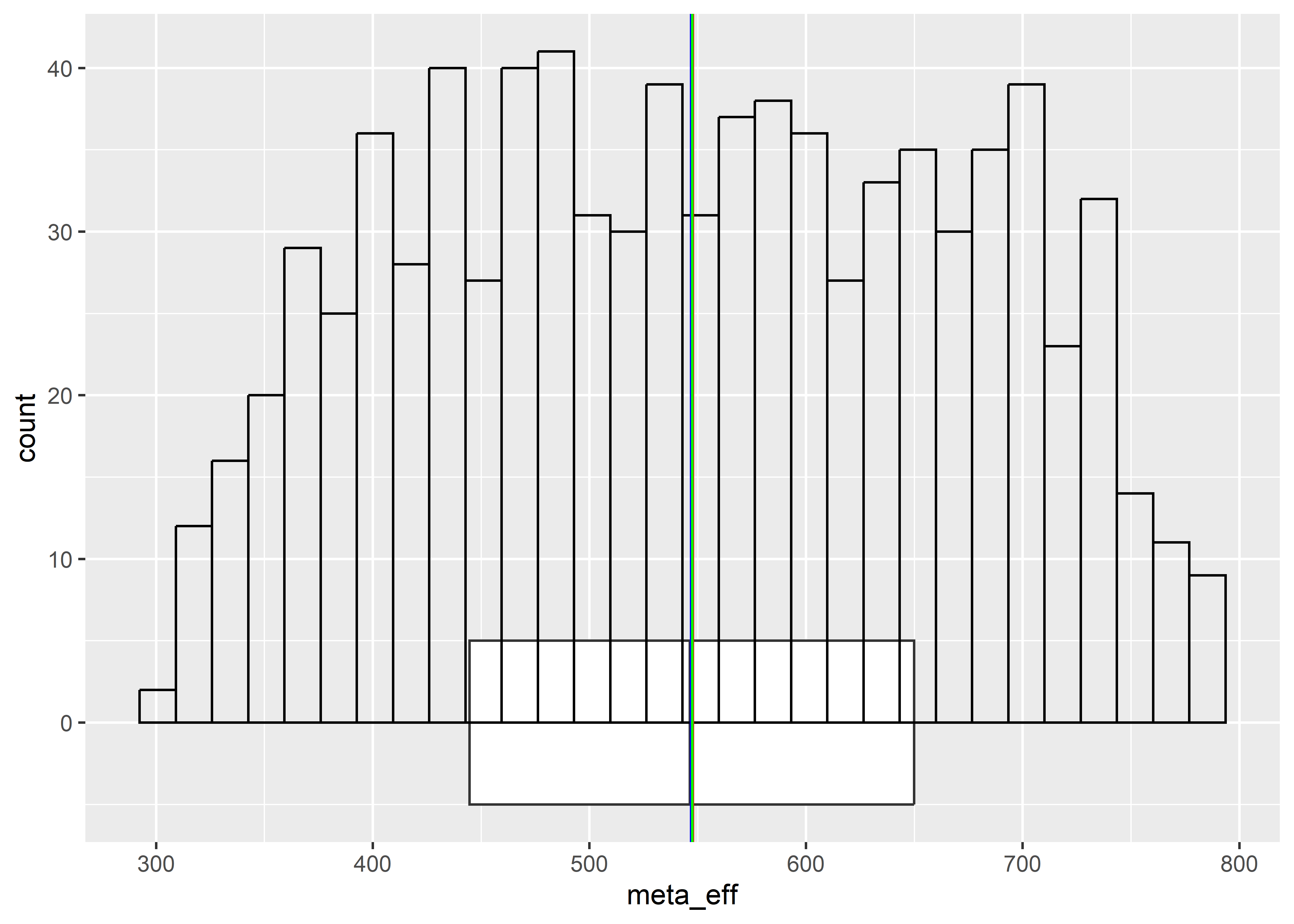
kurtosis(ratones$ein_resp)[1] 4.027426De acuerdo a las medidas de tendencia central, ambas variables
insulinymeta_efftienen la Media, Moda y Mediana muy cercanas, sugiriendo que ambas tienen distribucion normal. Sin embargo, al realizar la inspeccion gráfica y medición de lakurtosisobservamos queinsulines leptocurtica ymeta_effes platicurtica.
Resumen de variables
| var | mean | median | mode | sd | cv | skew | kurt | type |
|---|---|---|---|---|---|---|---|---|
| Bodyweight | 28.847038 | 28.19000 | 26.070000 | 6.260434 | 0.2170217 | 0.5567657 | 3.138633 | S. derecha |
| glucose | 7.006728 | 7.00021 | 7.029131 | 0.383118 | 0.0546786 | 0.0012147 | 3.355597 | Normal |
| en_intake | 43.066220 | 42.39931 | 40.193526 | 3.971329 | 0.0922145 | 0.7060313 | 2.880872 | S. derecha |
| insulin | 497.116404 | 494.06867 | 496.967784 | 110.452824 | 0.2221870 | 0.0586389 | 4.090441 | Normal, Lepto |
| meta_eff | 547.776213 | 546.87788 | 547.531554 | 123.088855 | 0.2247065 | 0.0053068 | 1.940977 | Normal, Plat |
| ein_resp | 5002.985187 | 5023.54299 | 5086.226871 | 191.299835 | 0.0382371 | -0.6105134 | 4.027426 | S. izquierda |
| time | 720.225261 | 720.39538 | 720.218237 | 5.588546 | 0.0077594 | 0.0396712 | 3.247685 | Normal |
Exactitud y precisión
Ejercicio 1
Carguemos el conjunto de datos:
data("ChickWeight")View(ChickWeight)Llamemos a tidyverse para poder hacer el procesamiento que se indica en el ejercicio:
Ahora, debemos calcular el promedio de las observaciones por cada pollito dada la dieta que recibió:
`summarise()` has grouped output by 'Chick'. You can override using the
`.groups` argument.View(chick)Procedemos a calcular las estimaciones la media del peso promedio de los pollitos durante el experimento para cada tipo de dieta y sus errores estándar:
chick_mean <- chick %>%
group_by(Diet) %>%
summarise(
n = n(),
sd = sd(mean_weight),
sample_mean = mean(mean_weight)
) |>
mutate(se = sd / sqrt(n))
chick_mean# A tibble: 4 × 5
Diet n sd sample_mean se
<fct> <int> <dbl> <dbl> <dbl>
1 1 20 31.1 98.1 6.95
2 2 10 31.4 123. 9.94
3 3 10 27.3 143. 8.63
4 4 10 16.2 135. 5.11Grafiquemos las estimaciones y sus errores estándar:
chick_mean %>%
ggplot(aes(x = Diet, y = sample_mean, col = Diet)) +
geom_point(size = 4) +
geom_errorbar(
aes(ymin = sample_mean - se, ymax = sample_mean + se), width = 0.1
)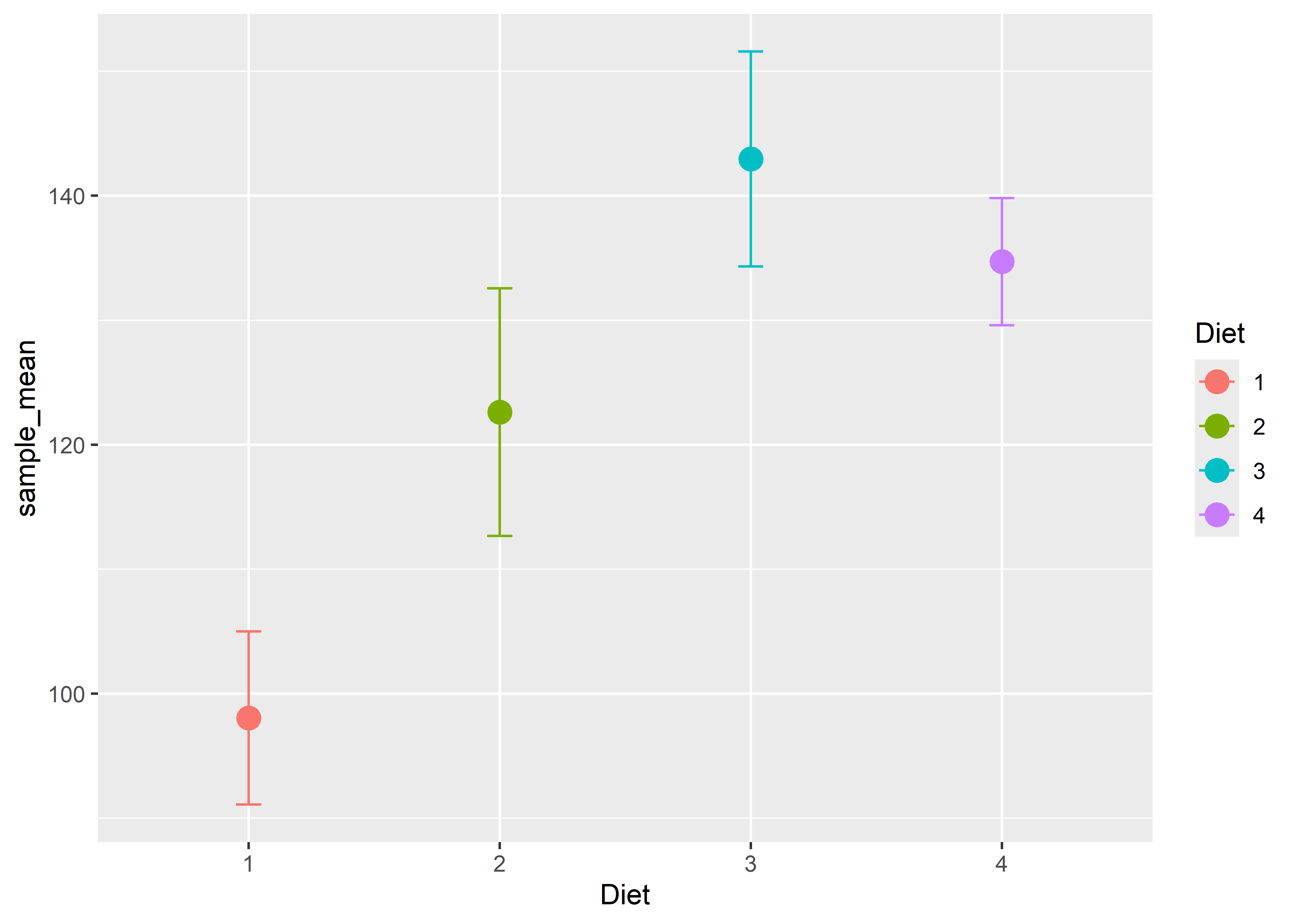
Se observa que la media estimada del peso promedio de los pollitos durante el experimento fue mayor en el grupo que recibió la dieta 3 (142.95$
Ahora, para calcular los intervalos de confianza para cada grupo, podemos usar la función t_test() del paquete rstatix. Cargamos el paquete y procedemos a realizar los cálculos:
Attaching package: 'rstatix'The following object is masked from 'package:stats':
filterchick_ci <- chick %>%
group_by(Diet) %>%
t_test(mean_weight ~ 1, detailed = TRUE) %>%
select(Diet, estimate, conf.low, conf.high)
chick_ci# A tibble: 4 × 4
Diet estimate conf.low conf.high
<fct> <dbl> <dbl> <dbl>
1 1 98.1 83.5 113.
2 2 123. 100. 145.
3 3 143. 123. 162.
4 4 135. 123. 146.Graficamos los intervalos de confianza para las estimaciones de la media del peso promedio de los pollitos durante el experimento:
chick_ci %>%
ggplot(aes(x = Diet, y = estimate, color = Diet)) +
geom_point(size = 4) +
geom_pointrange(aes(ymin = conf.low, ymax = conf.high))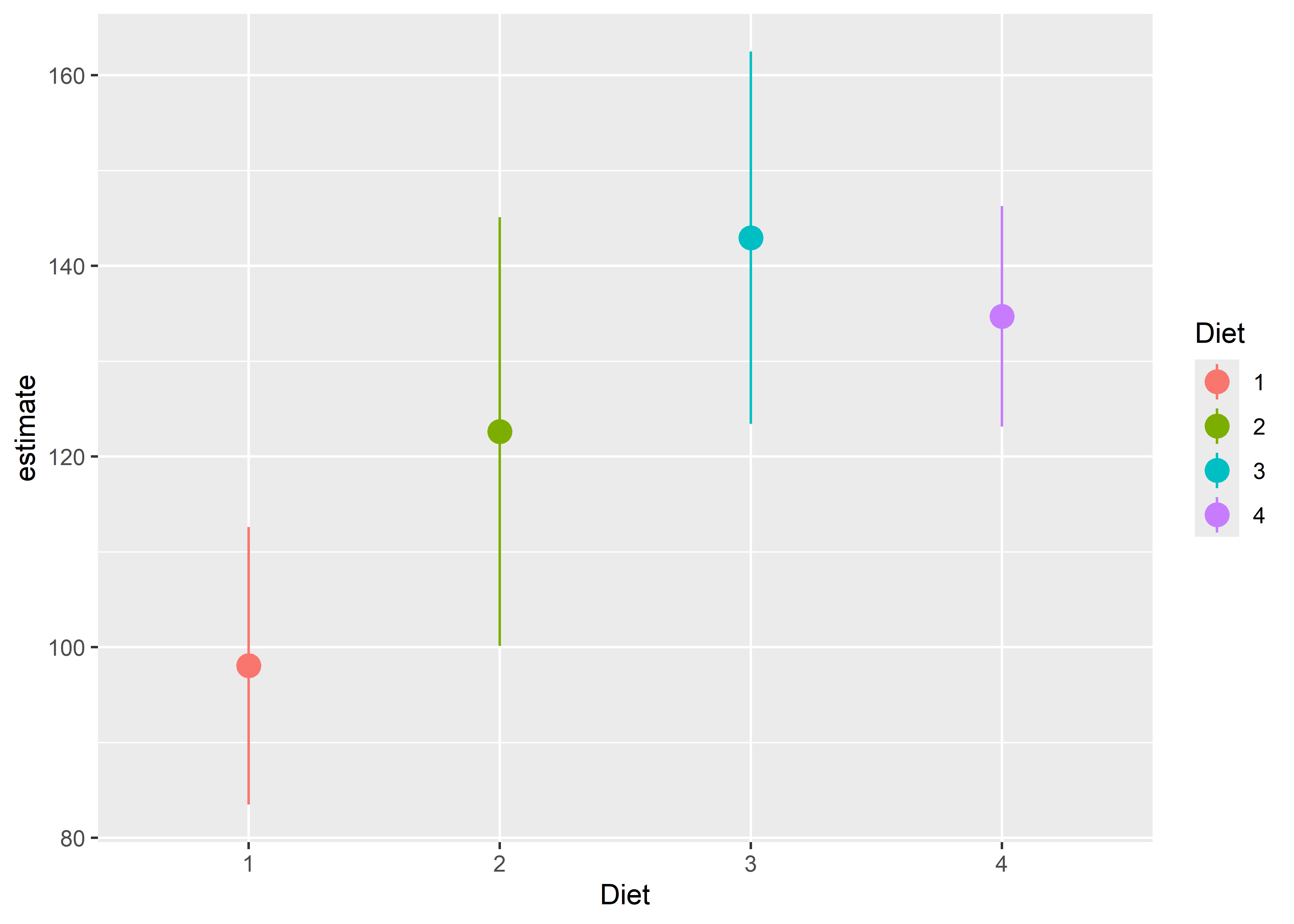
De este gráfico podemos llegar a las mismas conclusiones con respecto a la precisión de las estimaciones que con el gráfico anterior usando los errores estándar. Sin embargo, la interpretación de los intervalos de confianza es diferente. Aquí podemos decir, por ejemplo, que con un 95% de confianza, se estima que la media del peso promedio de los pollitos que reciben la dieta 3 se encuentra entre los 123.42 y 162.47 gramos.
Ejercicio 2
Carguemos los datos:
fev <- read_csv("data/fev.csv")Rows: 654 Columns: 6
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
dbl (6): id, age, fev, hgt, sex, smoke
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Ejecutemos los modelos:
regre_model_fit_1 <- lm(fev ~ age + hgt, data = fev)
regre_model_fit_2 <- lm(fev ~ age + poly(hgt, 2) + sex, data = fev)
library(mgcv)Loading required package: nlme
Attaching package: 'nlme'The following object is masked from 'package:dplyr':
collapseThis is mgcv 1.9-1. For overview type 'help("mgcv-package")'.
Attaching package: 'mgcv'The following object is masked from 'package:pracma':
magicLas mismas funciones vistas para los modelos de regresión lineal se pueden usar para los GAM.
Llamemos al paquete broom para poder usar la función augment() y calcular los valores predichos:
regre_model_pred_1 <- regre_model_fit_1 %>%
augment() %>%
select(obs = fev, pred = .fitted)
regre_model_pred_2 <- regre_model_fit_2 %>%
augment() %>%
select(obs = fev, pred = .fitted)
regre_model_pred_3 <- regre_model_fit_3 %>%
augment() %>%
select(obs = fev, pred = .fitted)
regre_models_predictions <- bind_rows(
list(
"model_1" = regre_model_pred_1, "model_2" = regre_model_pred_2,
"model_3" = regre_model_pred_3
),
.id = "model"
)Ahora, caguemos el paquete yardstick que contiene las funciones para el cálculo de diversas medidas para expresar el error de predicción:
Attaching package: 'yardstick'The following object is masked from 'package:readr':
specCon las medidas solicitadas en el ejercicio, definamos un conjunto de medidas:
metrics_regression <- metric_set(rmse, mae, mape)Por último, realicemos el cálculo de las medidas para cada uno de los modelos:
# A tibble: 9 × 4
model .metric .estimator .estimate
<chr> <chr> <chr> <dbl>
1 model_1 rmse standard 0.419
2 model_2 rmse standard 0.395
3 model_3 rmse standard 0.393
4 model_1 mae standard 0.319
5 model_2 mae standard 0.297
6 model_3 mae standard 0.294
7 model_1 mape standard 12.7
8 model_2 mape standard 11.7
9 model_3 mape standard 11.6 Observamos que, según todas las medidas, el modelo aditivo generalizado (model_3) tiene un menor error de predicción, seguido muy cercanamente por el modelo con efecto polinomial para la altura (model_2), mientras que el modelo con solo efectos lineales para ambos predictores (model_1) es claramente inferior que los dos anteriores. Dados estos resultados, por el principio de parsimonia (escoger el modelo más simple que tenga un buen ajuste o poder predictivo) podríamos preferir el model_2 al model_3, ya que el último es más complejo de interpretar.
Ejercicio 3
Cargamos los datos:
data("Bernard", package = "pubh")View(Bernard)Realicemos las operaciones que se indican en el ejercicio:
class_model_pred_1 <- class_model_fit_1 %>%
augment(type.predict = "response") %>%
mutate(pred_class = ifelse(.fitted > 0.5, "Dead", "Alive")) %>%
select(obs = fate, pred = pred_class)
class_model_pred_2 <- class_model_fit_2 %>%
augment(type.predict = "response") %>%
mutate(pred_class = ifelse(.fitted > 0.5, "Dead", "Alive")) %>%
select(obs = fate, pred = pred_class)
class_model_pred_3 <- class_model_fit_3 %>%
augment(type.predict = "response") %>%
mutate(pred_class = ifelse(.fitted > 0.5, "Dead", "Alive")) %>%
select(obs = fate, pred = pred_class)Al igual que en el ejercicio anterior, lo que se tendría que hacer una vez que se tienen las predicciones de todos los modelos es juntarlas en una sola tabla:
Como se nos dice en la pista del ejercicio, es necesario antes convertir a las columnas de las clases reales u observadas (obs) y las clases predichas (pred) en factores con el primer nivel como el evento de interés. Dado que nuestro evento de interés es “Alive”, ponemos a esta clase como el primer nivel:
Ahora, definimos un conjunto de medidas con aquellas solicitadas en el ejercicio y calculamos para cada modelo:
metrics_classification <- metric_set(accuracy, mcc, kap)
class_models_predictions %>%
group_by(model) %>%
metrics_classification(truth = obs, estimate = pred, event_level = "second")# A tibble: 9 × 4
model .metric .estimator .estimate
<chr> <chr> <chr> <dbl>
1 model_1 accuracy binary 0.685
2 model_2 accuracy binary 0.692
3 model_3 accuracy binary 0.692
4 model_1 mcc binary 0.190
5 model_2 mcc binary 0.227
6 model_3 mcc binary 0.220
7 model_1 kap binary 0.164
8 model_2 kap binary 0.208
9 model_3 kap binary 0.198Observamos que el primer modelo (model_1) tiene el peor performance de clasificación según las medidas de evaluación usadas. El segundo (model_2) y tercer (model_3) modelo tienen un performance similar, aunque model_2 tiene un mayor coeficiente de correlación de Matthews y coeficiente kappa e igual exactitud (accuracy) que model_3.
Regresión lineal: Conceptos básicos
Ejercicio 1
Leamos y demos un vistazo a los datos:
iq <- read_csv("data/iq.csv")Rows: 434 Columns: 5
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
dbl (5): kid_score, mom_hs, mom_iq, mom_work, mom_age
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.View(iq)Los puntajes de los test de IQ para los niños se encuentran en la variable kid_score, mientras que los puntajes de las madres en mom_iq. Para construir el modelo de regresión lineal, usamos la función lm():
Call:
lm(formula = kid_score ~ mom_iq, data = iq)
Residuals:
Min 1Q Median 3Q Max
-56.753 -12.074 2.217 11.710 47.691
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 25.79978 5.91741 4.36 1.63e-05 ***
mom_iq 0.60997 0.05852 10.42 < 2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 18.27 on 432 degrees of freedom
Multiple R-squared: 0.201, Adjusted R-squared: 0.1991
F-statistic: 108.6 on 1 and 432 DF, p-value: < 2.2e-16Podemos obtener los intervalos del confianza de los parámetros del modelo usando la función confint(). Para un 97% de confianza, agregamos el argumento level = 0.97:
confint(regre_simple_iq, level = 0.97) 1.5 % 98.5 %
(Intercept) 12.9158953 38.6836604
mom_iq 0.4825579 0.7373912Para obtener los resultados en formato tidy, podemos usar la función tidy() del paquete broom con los argumentos conf.int = TRUE y conf.level = 0.97:
tidy(regre_simple_iq, conf.int = TRUE, conf.level = 0.97)# A tibble: 2 × 7
term estimate std.error statistic p.value conf.low conf.high
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 25.8 5.92 4.36 1.63e- 5 12.9 38.7
2 mom_iq 0.610 0.0585 10.4 7.66e-23 0.483 0.737Interpretación: Tenemos un 97% de confianza de que, en promedio, el puntaje del test de IQ de un niño aumentaría entre 0.48 y 0.74 unidades por un aumento de una unidad en el puntaje de la madre.
Ejercicio 2
La variable mom_hs contiene la variable indicadora de si la madre se graduó (1) o no (0) de la secundaria. Primero, convertimos esta variable en factor:
Ahora, construimos el modelo de regresión lineal múltiple:
Call:
lm(formula = kid_score ~ mom_iq + mom_hs, data = iq_v2)
Residuals:
Min 1Q Median 3Q Max
-52.873 -12.663 2.404 11.356 49.545
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 25.73154 5.87521 4.380 1.49e-05 ***
mom_iq 0.56391 0.06057 9.309 < 2e-16 ***
mom_hsSí 5.95012 2.21181 2.690 0.00742 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 18.14 on 431 degrees of freedom
Multiple R-squared: 0.2141, Adjusted R-squared: 0.2105
F-statistic: 58.72 on 2 and 431 DF, p-value: < 2.2e-16Por último, obtenemos los intervalos de confianza:
tidy(regre_mult_iq, conf.int = TRUE, conf.level = 0.97)# A tibble: 3 × 7
term estimate std.error statistic p.value conf.low conf.high
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 25.7 5.88 4.38 1.49e- 5 12.9 38.5
2 mom_iq 0.564 0.0606 9.31 6.61e-19 0.432 0.696
3 mom_hsSí 5.95 2.21 2.69 7.42e- 3 1.13 10.8 Intrepretación:
Tenemos un 97% de confianza de que, en promedio, el puntaje del test de IQ de un niño aumenta entre 0.43 y 0.70 unidades por un aumento de una unidad en el puntaje de la madre, dado que la madre no cambia su condición de graduada o no graduada de la secundaria.
Tenemos un 97% de confianza de que si la madre pasara de no estar graduada de la secundaria a graduada, el puntaje del test de IQ del niño aumentaría, en promedio, entre 1.1343381 y 10.765896 unidades, dado que la madre mantiene su puntaje de IQ.
Ejercicio 3
regre_mult_inter_iq <- lm(kid_score ~ mom_iq + mom_hs + mom_iq*mom_hs, data = iq_v2)
summary(regre_mult_inter_iq)
Call:
lm(formula = kid_score ~ mom_iq + mom_hs + mom_iq * mom_hs, data = iq_v2)
Residuals:
Min 1Q Median 3Q Max
-52.092 -11.332 2.066 11.663 43.880
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -11.4820 13.7580 -0.835 0.404422
mom_iq 0.9689 0.1483 6.531 1.84e-10 ***
mom_hsSí 51.2682 15.3376 3.343 0.000902 ***
mom_iq:mom_hsSí -0.4843 0.1622 -2.985 0.002994 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 17.97 on 430 degrees of freedom
Multiple R-squared: 0.2301, Adjusted R-squared: 0.2247
F-statistic: 42.84 on 3 and 430 DF, p-value: < 2.2e-16Para obtener los efectos marginales, podemos usar la función estimate_slopes() del paquete modelbased:
Attaching package: 'modelbased'The following object is masked from 'package:rstatix':
get_emmeansThe following object is masked from 'package:huxtable':
print_mdestimate_slopes(regre_mult_inter_iq, trend = "mom_iq", at = "mom_hs")Estimated Marginal Effects
mom_hs | Coefficient | SE | 95% CI | t(430) | p
------------------------------------------------------------
No | 0.97 | 0.15 | [0.68, 1.26] | 6.53 | < .001
Sí | 0.48 | 0.07 | [0.36, 0.61] | 7.38 | < .001
Marginal effects estimated for mom_iqIntrepretación: El aumento promedio en el puntaje del test de IQ de un niño asociado a un aumento en el puntaje del test de IQ de la madre es de 0.97 unidades en el grupo de las madres que no se graduaron de la secundaria, mientras que en el grupo de las madres que sí se graduaron el aumento promedio es de 0.48 unidades.
Regresión lineal: Evaluación y extensiones
Ejercicio 1
Leamos y demos un vistazo a los datos:
iq <- read_csv("data/iq.csv")Rows: 434 Columns: 5
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
dbl (5): kid_score, mom_hs, mom_iq, mom_work, mom_age
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.View(iq)Los puntajes de los test de IQ para los niños se encuentran en la variable kid_score, los puntajes de las madres en mom_iq, y la variable indicadora de si la madre se graduó o no de la secundaria está dada por mom_hs. Primero, convertimos la variable mom_hs en factor:
Ahora, construimos el modelo de regresión múltiple:
Call:
lm(formula = kid_score ~ mom_iq + mom_hs, data = iq_v2)
Residuals:
Min 1Q Median 3Q Max
-52.873 -12.663 2.404 11.356 49.545
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 25.73154 5.87521 4.380 1.49e-05 ***
mom_iq 0.56391 0.06057 9.309 < 2e-16 ***
mom_hsSí 5.95012 2.21181 2.690 0.00742 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 18.14 on 431 degrees of freedom
Multiple R-squared: 0.2141, Adjusted R-squared: 0.2105
F-statistic: 58.72 on 2 and 431 DF, p-value: < 2.2e-16Para generar los gráficos de residuales, podemos usar la función autoplot() del paquete ggfortify:
autoplot(regre_mult_iq)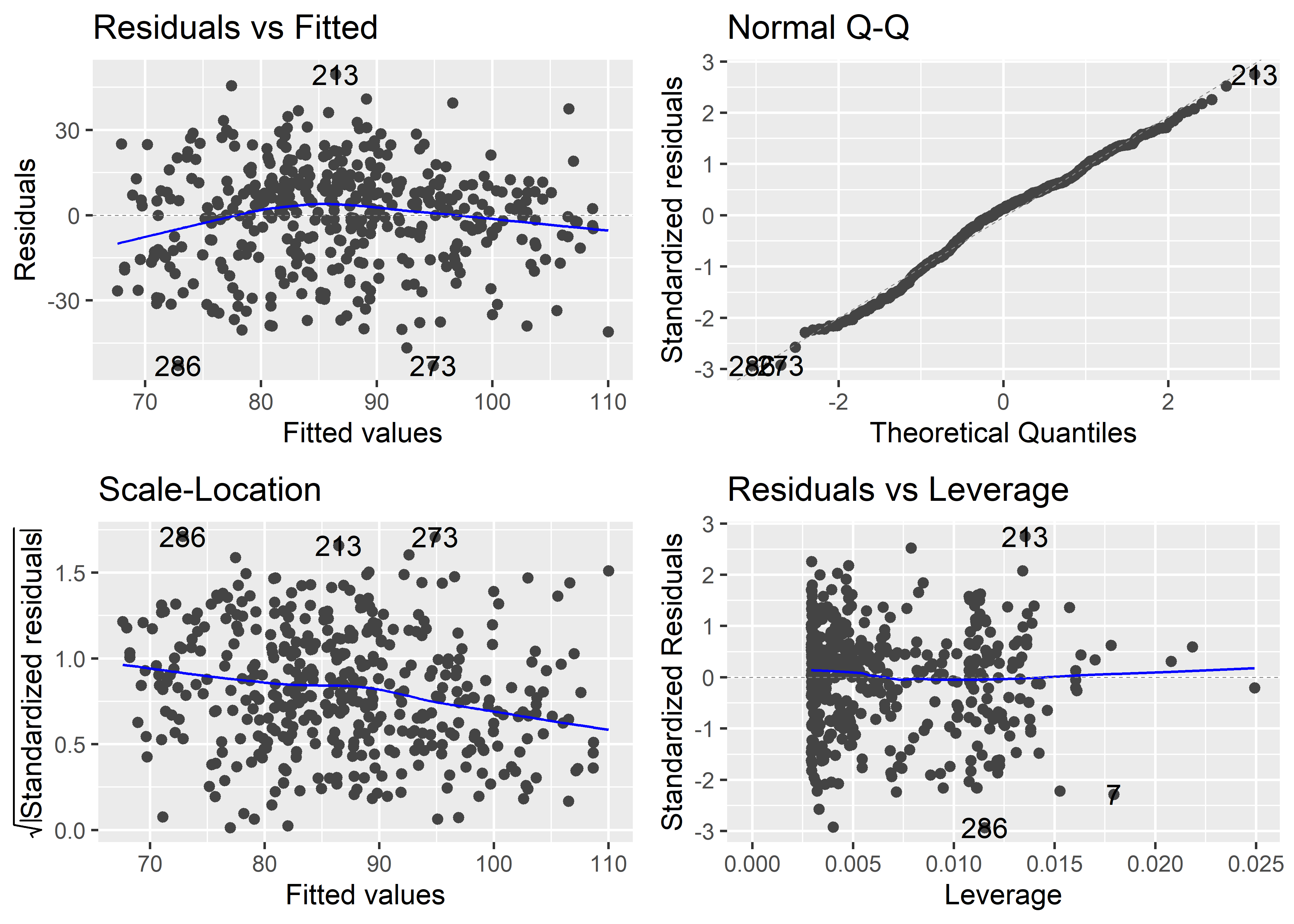
Vemos que, en general, no hay patrones que podrían indicar una falta grave de adecuación del modelo. Las líneas de tendencia en los gráficos “Residuals vs Fitted” y “Scale-location” no son exactamente horizontales, pero tampoco tienen un patrón curvilíneo acentuado. Sin embargo, podría ser buena idea probar añadiendo más predictores o alguna transformación de los predictores actuales para ver si se logra eliminar estos ligeros patrones.
Ejercicio 2
Para añadir una transformación polinómica de grado 2 sobre un predictor usamos la función poly() y pasamos al parámetro degree el valor 2:
regre_mult_poly_iq <- lm(kid_score ~ poly(mom_iq, degree = 2) + mom_hs, data = iq_v2)
summary(regre_mult_poly_iq)
Call:
lm(formula = kid_score ~ poly(mom_iq, degree = 2) + mom_hs, data = iq_v2)
Residuals:
Min 1Q Median 3Q Max
-54.553 -10.940 2.502 11.095 48.710
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 82.784 1.937 42.749 <2e-16 ***
poly(mom_iq, degree = 2)1 178.049 18.733 9.504 <2e-16 ***
poly(mom_iq, degree = 2)2 -55.989 18.096 -3.094 0.0021 **
mom_hsSí 5.107 2.207 2.314 0.0211 *
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 17.96 on 430 degrees of freedom
Multiple R-squared: 0.2313, Adjusted R-squared: 0.2259
F-statistic: 43.12 on 3 and 430 DF, p-value: < 2.2e-16Ahora veamos los gráficos de residuales:
autoplot(regre_mult_poly_iq)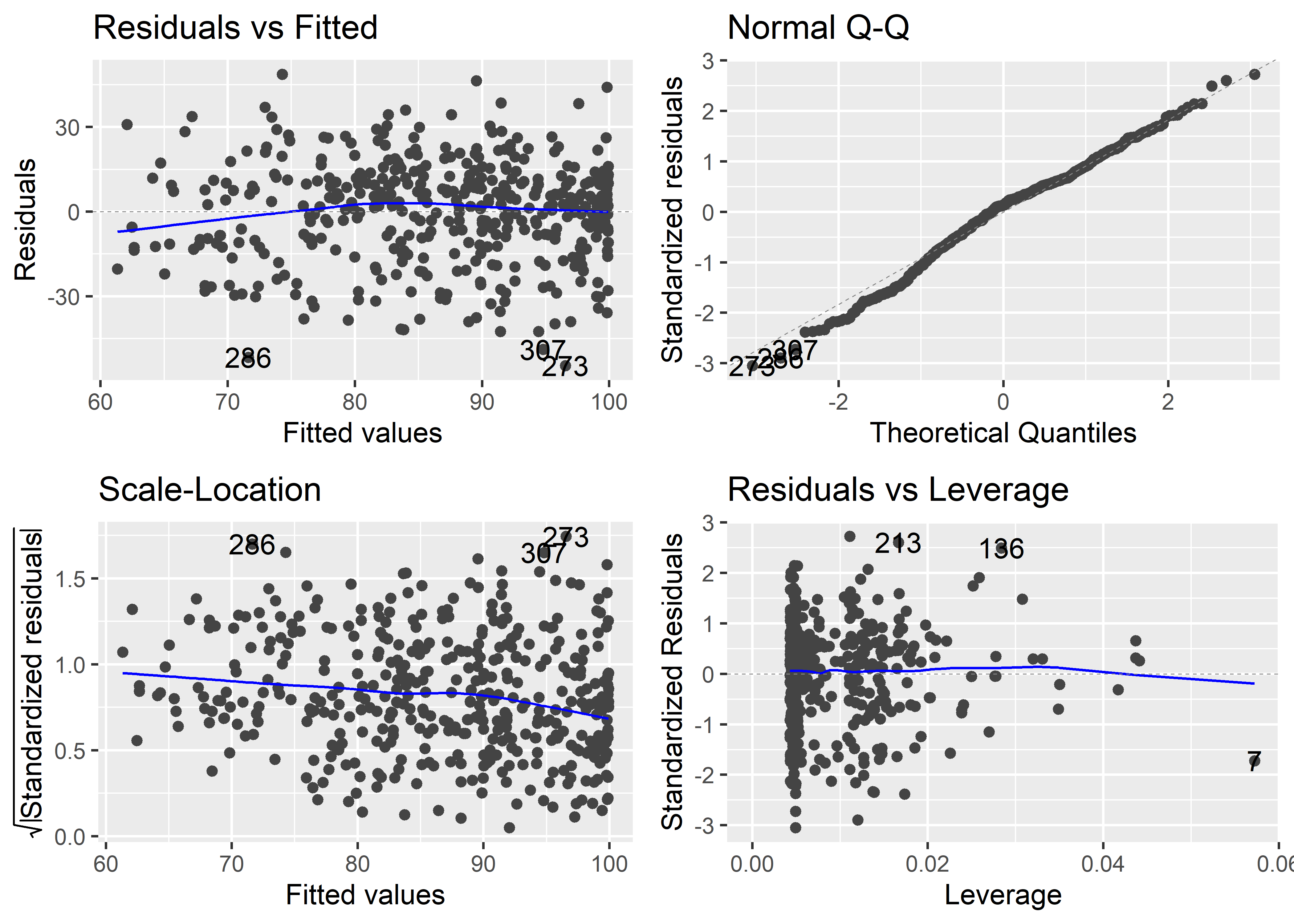
Vemos que con la transformación polinómica sobre mom_iq, las líneas de tendencia en los gráficos “Residuals vs Fitted” y “Scale-location” se hacen más horizontales y los patrones curvilíneos se atenúan. Por lo tanto, este modelo parece ser adecuado para explicar los puntajes de las pruebas de IQ de los niños.
Ejercicio 3
Ajustemos los modelos que se piden:
Podemos usar la función compare_performance() del paquete performance para comparar el ajuste de los modelos:
Attaching package: 'performance'The following objects are masked from 'package:yardstick':
mae, rmseThe following objects are masked from 'package:huxtable':
print_html, print_mdcompare_performance(
regre_simple_iq_iq, regre_simple_hs_iq, regre_multi_iq, regre_multi_inter_iq,
rank = TRUE
)# Comparison of Model Performance Indices
Name | Model | R2 | R2 (adj.) | RMSE | Sigma | AIC weights | AICc weights | BIC weights | Performance-Score
---------------------------------------------------------------------------------------------------------------------------------
regre_multi_inter_iq | lm | 0.230 | 0.225 | 17.888 | 17.971 | 0.967 | 0.966 | 0.725 | 100.00%
regre_multi_iq | lm | 0.214 | 0.210 | 18.073 | 18.136 | 0.031 | 0.031 | 0.176 | 56.41%
regre_simple_iq_iq | lm | 0.201 | 0.199 | 18.224 | 18.266 | 0.002 | 0.002 | 0.099 | 49.89%
regre_simple_hs_iq | lm | 0.056 | 0.054 | 19.807 | 19.853 | 4.51e-19 | 4.70e-19 | 1.98e-17 | 0.00%Considerando todos los indicadores de ajuste, el modelo que incluye la interacción entre el puntaje del test de IQ de la madre y la condición de si la madre se graduó o no de secundaria tiene un ajuste a los datos.
Medidas de frecuencia
Ejercicio 1
- Prevalencia puntual al incio de estudio de enfermedad coronaria estratificado por diabetes
angina_0_diabetes <- framingham %>%
filter(TIME == 0) %>%
mutate(PREVMI = factor(PREVMI,
levels = c(0,1),
labels = c("no", "yes")),
DIABETES = factor(DIABETES,
levels = c(0,1),
labels = c("No diabetes", "Diabetes"))) %>%
group_by(DIABETES, PREVMI) %>%
summarise(n=n())`summarise()` has grouped output by 'DIABETES'. You can override using the
`.groups` argument.angina_0_diabetes# A tibble: 4 × 3
# Groups: DIABETES [2]
DIABETES PREVMI n
<fct> <fct> <int>
1 No diabetes no 4234
2 No diabetes yes 79
3 Diabetes no 114
4 Diabetes yes 7Adding missing grouping variables: `DIABETES`
Storing counts in `nn`, as `n` already present in input# A tibble: 4 × 6
# Groups: DIABETES [2]
DIABETES n midpoint me lower upper
<fct> <int> <dbl> <dbl> <dbl> <dbl>
1 No diabetes 79 1.83 0.400 1.43 2.23
2 No diabetes 4234 98.2 0.400 97.8 98.6
3 Diabetes 7 5.79 4.18 1.61 9.96
4 Diabetes 114 94.2 4.18 90.0 98.4 En la población de Framingham, la prevalencia puntual al incio del estudio de infarto al miocardio en población diabética es 4.18 (IC95%: 90.04 - 98.39).
Ejercicio 2
- Prevalencia de hipertensión en el periodo 2
#Seleccionando el periodo 2
pp_hipertension <- framingham %>%
mutate(hipertension = PREVHYP + HYPERTEN) %>%
mutate(hipertension = case_when(hipertension >= 1 ~ 1,
TRUE ~ 0)) %>%
mutate(hipertension = factor(hipertension,
levels = c(0,1),
labels = c("No hipertensión", "Hipertensión")))
hipertension_p2 <- pp_hipertension %>%
filter(PERIOD == 2) %>%
group_by(hipertension) %>%
summarise(n = n())
hipertension_p2 %>%
rpro(n, ci = 95)Storing counts in `nn`, as `n` already present in input
ℹ Use `name = "new_name"` to pick a new name.# A tibble: 2 × 5
n midpoint me lower upper
<int> <dbl> <dbl> <dbl> <dbl>
1 995 25.3 1.36 24.0 26.7
2 2936 74.7 1.36 73.3 76.0En la población de Framingham, la prevalencia de hipertensión en el segundo periodo de estudio 1.36 (IC95%: 73.33 - 76.05).
Ejercicio 3
- Incidencia de stroke en el periodo 3
free_prev_stroke <- framingham %>%
filter(PREVSTRK == 0 & PERIOD == 3) %>%
mutate(STROKE = factor(STROKE,
levels = c(0,1),
labels = c("No stroke ", "Stroke")))
stroke <- free_prev_stroke %>%
group_by(STROKE) %>%
count()
stroke %>%
ungroup() %>%
rpro(n, ci = 95)Storing counts in `nn`, as `n` already present in input
ℹ Use `name = "new_name"` to pick a new name.# A tibble: 2 × 5
n midpoint me lower upper
<int> <dbl> <dbl> <dbl> <dbl>
1 214 6.70 0.867 5.83 7.57
2 2980 93.3 0.867 92.4 94.2 Durante el periodo 3 del estudio de Framingham, se observó una incidencia acumulada del 6.7% (IC 95%: 5.83 - 7.57) . Es decir, por cada 1000 personas, hubo aproximadamente 67 casos nuevos de stroke.
Medidas de asociación
Ejercicio 1
Calculando la Razón de prevalencia de PREVCHD al basal respecto al factor de exposición de sexo:
prevalence_chd <- framingham %>%
filter(TIME==0) %>%
select(PREVCHD, SEX) %>%
mutate(PREVCHD = factor(PREVCHD,
levels = c(1,0),
labels = c("Enfermedad cardicaca", "No enfermedad cardiaca")),
SEX = factor(SEX, levels = c(1,2),
labels = c("Male", "Female")))
tab_prevalence <- table(prevalence_chd$SEX, prevalence_chd$PREVCHD)
tab_prevalence
Enfermedad cardicaca No enfermedad cardiaca
Male 124 1820
Female 70 2420epi.2by2(tab_prevalence, method = "cross.sectional") Outcome + Outcome - Total Prev risk *
Exposed + 124 1820 1944 6.38 (5.33 to 7.56)
Exposed - 70 2420 2490 2.81 (2.20 to 3.54)
Total 194 4240 4434 4.38 (3.79 to 5.02)
Point estimates and 95% CIs:
-------------------------------------------------------------------
Prev risk ratio 2.27 (1.70, 3.02)
Prev odds ratio 2.36 (1.75, 3.18)
Attrib prev in the exposed * 3.57 (2.30, 4.83)
Attrib fraction in the exposed (%) 55.93 (41.28, 66.92)
Attrib prev in the population * 1.56 (0.68, 2.45)
Attrib fraction in the population (%) 35.75 (22.78, 46.53)
-------------------------------------------------------------------
Uncorrected chi2 test that OR = 1: chi2(1) = 33.206 Pr>chi2 = <0.001
Fisher exact test that OR = 1: Pr>chi2 = <0.001
Wald confidence limits
CI: confidence interval
* Outcomes per 100 population units En la población de Framingham, la prevalencia de cardiopatía coronaria en las mujeres es 2.27 (IC95%: 1.70 - 3.02) veces la prevalencia en el grupo de los hombres.
Calculando la Razón de Odds de PREVCHD al basal respecto al factor de exposición de condición de fumador:
odds_chd <- framingham %>%
filter(TIME==0) %>%
select(PREVCHD, CURSMOKE) %>%
mutate(PREVCHD = factor(PREVCHD,
levels = c(1,0),
labels = c("Enfermedad cardicaca", "No enfermedad cardiaca")),
CURSMOKE = factor(CURSMOKE,
levels = c(1,0),
labels = c("Fumador","No Fumador")))
odds_tab <- table(odds_chd$CURSMOKE, odds_chd$PREVCHD)
odds_tab
Enfermedad cardicaca No enfermedad cardiaca
Fumador 86 2095
No Fumador 108 2145epi.2by2(odds_tab, method = "case.control") Outcome + Outcome - Total Odds
Exposed + 86 2095 2181 0.04 (0.03 to 0.05)
Exposed - 108 2145 2253 0.05 (0.04 to 0.06)
Total 194 4240 4434 0.05 (0.04 to 0.05)
Point estimates and 95% CIs:
-------------------------------------------------------------------
Exposure odds ratio 0.82 (0.61, 1.09)
Attrib fraction (est) in the exposed (%) -22.65 (-65.86, 9.08)
Attrib fraction (est) in the population (%) -10.04 (-25.20, 3.28)
-------------------------------------------------------------------
Uncorrected chi2 test that OR = 1: chi2(1) = 1.916 Pr>chi2 = 0.166
Fisher exact test that OR = 1: Pr>chi2 = 0.186
Wald confidence limits
CI: confidence intervalEn la población de Framingham, el Odds de cardiopatía coronaria en el grupo de fumadores es 0.82 (IC95%: 0.61 - 1.09) veces el Odds en el grupo de no fumadores. Como se puede observar, el intervalo de confianza contiene a la unidad, por tal motivo, este resultado no es estadísticamente significativo.
Calculando la Razón de Odds de PREVCHD en el periodo 2 respecto al factor de exposición de diabetes:
incident_chd <- framingham %>%
filter(PREVCHD == 0 & PERIOD == 2) %>%
select(ANYCHD, DIABETES) %>%
mutate(ANYCHD = factor(ANYCHD,
levels = c(1,0),
labels = c("Enfermedad cardicac", "No enfermedad cardiaca")),
DIABETES = factor(DIABETES,
levels = c(1,0),
labels = c("Diabetico", "No diabetico")))
tab_incience <- table(incident_chd$DIABETES, incident_chd$ANYCHD)
tab_incience
Enfermedad cardicac No enfermedad cardiaca
Diabetico 62 62
No diabetico 721 2797epi.2by2(tab_incience, method = "cohort.count") Outcome + Outcome - Total Inc risk *
Exposed + 62 62 124 50.00 (40.89 to 59.11)
Exposed - 721 2797 3518 20.49 (19.17 to 21.87)
Total 783 2859 3642 21.50 (20.17 to 22.87)
Point estimates and 95% CIs:
-------------------------------------------------------------------
Inc risk ratio 2.44 (2.02, 2.94)
Inc odds ratio 3.88 (2.70, 5.57)
Attrib risk in the exposed * 29.51 (20.60, 38.41)
Attrib fraction in the exposed (%) 59.01 (50.55, 66.02)
Attrib risk in the population * 1.00 (-0.88, 2.89)
Attrib fraction in the population (%) 4.67 (3.05, 6.27)
-------------------------------------------------------------------
Uncorrected chi2 test that OR = 1: chi2(1) = 61.785 Pr>chi2 = <0.001
Fisher exact test that OR = 1: Pr>chi2 = <0.001
Wald confidence limits
CI: confidence interval
* Outcomes per 100 population units En la población de Framingham, la incidencia de cardiopatía coronaria en el grupo de diabéticos es 2.44 (IC95%: 2.02 - 2.94) veces la incidencia en el grupo de no fumadores.
Ejercicio 2
Razón de tasa de incidencias para la variable Enfermedad Coronaria (ANYCHD) en función de la condición de diabetes. Hacer el cálculo para el segundo periodo de estudio.
rti_chd <- framingham %>%
filter(PREVCHD == 0 & PERIOD == 2) %>%
select(ANYCHD, DIABETES, TIMECHD) %>%
mutate(ANYCHD = factor(ANYCHD,
levels = c(1,0),
labels = c("Enfermedad cardicaca", "No enfermedad cardiaca")),
DIABETES = factor(DIABETES,
levels = c(1,0),
labels = c("Diabetico", "No diabetico"))) %>%
filter(ANYCHD == "Enfermedad cardicaca") %>%
group_by(DIABETES, ANYCHD) %>%
summarise(personas_tiempo = sum(TIMECHD),
cases = n()) %>%
select(-ANYCHD) %>%
relocate(cases, .after = DIABETES)`summarise()` has grouped output by 'DIABETES'. You can override using the
`.groups` argument.rti_chd# A tibble: 2 × 3
# Groups: DIABETES [2]
DIABETES cases personas_tiempo
<fct> <int> <int>
1 Diabetico 62 314764
2 No diabetico 721 3988907rti_final <- rateratio(rti_chd$cases[1], rti_chd$cases[2],
rti_chd$personas_tiempo[1], rti_chd$personas_tiempo[2],
conf.level = 0.95) Cases Person-time
Exposed 62 314764
Unexposed 721 3988907
Total 783 4303671str(rti_final)List of 5
$ p.value : num 0.516
$ conf.int : num [1:2] 0.841 1.412
..- attr(*, "conf.level")= num 0.95
$ estimate : num 1.09
$ method : chr "Incidence rate ratio estimate and its significance probability"
$ data.name: chr "rti_chd$cases[1] rti_chd$cases[2] rti_chd$personas_tiempo[1] rti_chd$personas_tiempo[2]"
- attr(*, "class")= chr "htest"print(rti_final)
Incidence rate ratio estimate and its significance probability
data: rti_chd$cases[1] rti_chd$cases[2] rti_chd$personas_tiempo[1] rti_chd$personas_tiempo[2]
p-value = 0.516
95 percent confidence interval:
0.8407569 1.4124728
sample estimates:
[1] 1.089746En la población de Framingham, la tasa de incidencia de enfermedad coronaria en el grupo de diabéticos es 1.09 (IC95%: 0.84 - 1.41) veces la tasa de incidencia en el grupo de no diabéticos.
Estudios Transversales
Ejercicio 1
Elabore un modelo de regresión logístico incluyendo a la condición de UCI (UCI) como outcome y solo a las variables Sexo (SEX) y la condición de fumador (TABAQUISMO) como predictores. Exponencie los coeficientes de la regresión para obtener los OR.
Para ello, factorizaremos las variables que serán utilizadas en el análisis.
covid_mex_clean_1 <- covid_mex %>%
mutate(
SEXO = case_when(
SEXO == 1 ~ 1,
TRUE ~ 0
),
TABAQUISMO = factor(
TABAQUISMO,
levels = c(0, 1),
labels = c("No Fumador", "Fumador")
),
SEXO = factor(
SEXO,
levels = c(0, 1),
labels = c("Femenino", "Masculino")
),
UCI = factor(
UCI,
levels = c(0,1),
labels = c("No UCI", "UCI")
)
)Posteriormente, se ejecutará el modelo de regresión logística
Finalmente, se deben exponenciar los coeficientes para poder obtener el OR.
exponenciado_logistica <- log_reg_eje_1 %>%
tidy(exponentiate = TRUE, conf.int = TRUE)
exponenciado_logistica# A tibble: 3 × 7
term estimate std.error statistic p.value conf.low conf.high
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 0.0722 0.104 -25.3 6.42e-141 0.0585 0.0880
2 SEXOMasculino 0.753 0.177 -1.60 1.09e- 1 0.529 1.06
3 TABAQUISMOFumador 1.30 0.257 1.01 3.15e- 1 0.760 2.09 En la población de pacientes mexicanos con COVID-19, El Odds de ingresar a UCI en el grupo de hombres es 0.75 (IC95%: 0.53 - 1.06) veces el Odds en el grupo de mujeres, luego de ajustar por la condición de fumador. Este resultado no fue estadísticamente significativo (p=0.109).
Ejercicio 2
Elabore un modelo de regresión de Poisson Modificado incluyendo a la condición de UCI (UCI) como outcome y solo a las variables obesidad (OBESIDAD) y la condición de asma (ASMA) como predictores. Exponencie los coeficientes de la regresión para obtener los OR.
Al igual que en el ejercicio anterior, deberemos convertir en factor las variables que serán utilizadas en nuestro análisis.
Posteriormente, se ejecutará el modelo de regresión de Poisson
poisson_eje_2 <- glm(
UCI ~ OBESIDAD + ASMA,
family = poisson(link="log"),
data = covid_mex_clean_2
)
poisson_eje_2
Call: glm(formula = UCI ~ OBESIDAD + ASMA, family = poisson(link = "log"),
data = covid_mex_clean_2)
Coefficients:
(Intercept) OBESIDADObeso ASMANo Asmatico
-2.8826 0.3812 0.4839
Degrees of Freedom: 2599 Total (i.e. Null); 2597 Residual
Null Deviance: 906.4
Residual Deviance: 899.8 AIC: 1234Ahora estimaremos la varianza robusta y exponenciaremos los coeficientes.
model_poisson_mod_exp <- summ(poisson_eje_2, robust = "HC1", confint = T, digits = 3, exp = T)
model_poisson_mod_exp| Observations | 2600 |
| Dependent variable | UCI |
| Type | Generalized linear model |
| Family | poisson |
| Link | log |
| χ²(2) | 6.557 |
| Pseudo-R² (Cragg-Uhler) | 0.007 |
| Pseudo-R² (McFadden) | 0.005 |
| AIC | 1233.839 |
| BIC | 1251.429 |
| exp(Est.) | 2.5% | 97.5% | z val. | p | |
|---|---|---|---|---|---|
| (Intercept) | 0.056 | 0.047 | 0.067 | -31.203 | 0.000 |
| OBESIDADObeso | 1.464 | 1.065 | 2.013 | 2.348 | 0.019 |
| ASMANo Asmatico | 1.622 | 0.795 | 3.310 | 1.330 | 0.184 |
| Standard errors: Robust, type = HC1 |
En la población de pacientes mexicanos con COVID-19, la proporción de personas que ingresan a UCI en el grupo de obesos es 1.46 (IC95%: 1.07 - 2.01) veces la proporción en el grupo de no obesos, luego de ajustar por la condición de asma Este resultado fue estadísticamente significativo con un valor p = 0.019.
Estudios de Casos y Controles
Ejercicio 1
A) Usando la base de datos peru (dhs.csv), realizar un emparejamiento individual en base a la provincia, grupo de riqueza y quintil de edad. Utilizar una proporción fija. Es decir, incluir solo a los casos que se hayan podido emparejar 3 controles.
library(tidyverse)
library(MatchIt)
library(survival)
library(broom)
peru <- read_csv("data/dhs.csv")
peru <- peru %>%
drop_na() %>%
mutate(province = str_sub(village, 1, 2),
safe_water = factor(safe_water, labels = c("No", "Si")),
age_cat = cut_number(age, 5))
peru_match <- matchit(death_1m ~ province + wealth_ind + age_cat,
exact = ~ province + wealth_ind + age_cat,
ratio = 3,
data = peru)
dat_m <- match.data(peru_match)
dat_m <- dat_m %>%
group_by(subclass) %>%
filter(n() > 3) %>%
ungroup()
logit2 <- clogit(death_1m ~ safe_water + strata(subclass), data = dat_m)
tidy(logit2, exponentiate = T, conf.int = T)# A tibble: 1 × 7
term estimate std.error statistic p.value conf.low conf.high
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 safe_waterSi 0.498 0.386 -1.81 0.0707 0.234 1.06B) Comparar los resultados con el emparejamiento individual con proporción fija.
Estudios de cohortes
Ejercicio 1
Realizar un modelo de regresión de Poisson para estimar tasas individuales, considerando como desenlace a la variable casos de hipertensión (HYPERTEN) y como covariables a edad (AGE), número de cigarrillos por día (CIGPDAY) e índice de masa corporal (BMI).
Recordemos que en un estudio de cohortes se iniciará el análisis con casos incidentes (casos nuevos). Para ello, manipularemos la base de datos para obtener los casos nuevos de hipertensión.
Utilizaremos el comando slice_tail() para obtener el último registro de cada participante en la base de datos.
A continuación ejecutaremos el modelo de regresión de Poisson.
poisson_hipertension <- glm(HYPERTEN ~ AGE + CIGPDAY + BMI,
family = poisson(link="log"),
data = casos_hipertension)
poisson_hipertension_tidy <- tidy(poisson_hipertension, conf.int = T, exponentiate = T)
poisson_hipertension_tidy# A tibble: 4 × 7
term estimate std.error statistic p.value conf.low conf.high
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 0.546 0.237 -2.55 0.0107 0.343 0.869
2 AGE 0.987 0.00281 -4.79 0.00000171 0.981 0.992
3 CIGPDAY 0.996 0.00197 -2.11 0.0349 0.992 1.00
4 BMI 1.03 0.00633 5.40 0.0000000659 1.02 1.05 En la población de estudio, la incidencia acumulada de hipertensión arterial es 0.987 (IC95%: 0.981 - 0.992) cuando la edad incrementa en una unidad, ajustando por cigarrillos por día e índice de masa corporal. Siendo este resultado estadísticamente significativo (p < 0.001).
Ejercicio 2
Considerando las variables del modelo del ejercicio 1, calcule las tasas agrupadas por periodo (PERIOD) y sexo (SEX).
casos_hipertension_agrupado <- casos_hipertension %>%
group_by(PERIOD, SEX) %>%
summarise(casos_hipertension = sum(HYPERTEN, na.rm = T),
AGE = mean(AGE, na.rm = T),
BMI = mean(BMI, na.rm = T),
CIGPDAY = mean(CIGPDAY, na.rm = T)
)`summarise()` has grouped output by 'PERIOD'. You can override using the
`.groups` argument.En la población de estudio, la incidencia acumulada de hipertensión arterial es 0.802 (IC95%: 0.655 - 0.981) cuando la edad incrementa en una unidad, ajustando por consumo de cigarillo por día, índice de masa corporal y sexo. Siendo este resultado estadísticamente significativo (p=0.032).
Estudios de supervivencia
Para este ejercicio, procedemos con la creación de la variable edad dicotomizada (<= 70 años y > 70 años), para su posterior factorización.
Con la base de datos preparada, realizaremos el análisis de supervivencia utilizando el método de Kaplan-Meier.
# A tibble: 201 × 9
time n.risk n.event n.censor estimate std.error conf.high conf.low strata
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
1 5 182 1 0 0.995 0.00551 1 0.984 edad_cat…
2 11 181 1 0 0.989 0.00781 1 0.974 edad_cat…
3 13 180 1 0 0.984 0.00960 1 0.965 edad_cat…
4 15 179 1 0 0.978 0.0111 1.00 0.957 edad_cat…
5 53 178 2 0 0.967 0.0137 0.993 0.941 edad_cat…
6 60 176 2 0 0.956 0.0159 0.986 0.927 edad_cat…
7 61 174 1 0 0.951 0.0169 0.983 0.920 edad_cat…
8 62 173 1 0 0.945 0.0179 0.979 0.913 edad_cat…
9 65 172 2 0 0.934 0.0197 0.971 0.899 edad_cat…
10 71 170 1 0 0.929 0.0206 0.967 0.892 edad_cat…
# ℹ 191 more rowsEn el tiempo 15 del estudio, observamos que la supervivencia es de 97.80% (95%CI: 95.70% - 99.96%) en el grupo de personas que tienen 70 años o menos.
En el tiempo 13 del estudio, observamos que la supervivencia es de 91.30% (95%CI: 83.51% - 99.82%) en el grupo de personas que tienen más de 70 años.
Ejercicio 2
Para el gráfico de Kaplan-Meier estratificado utilizaremos la función autoplot().
autoplot(km_ecog_curve, surv.alpha = 0.8, conf.int.alpha = 0.4) +
labs(x = "Tiempo",
y = "Función de Supervivencia",
title = "Supervivencia de pacientes \n con cancer de pulmón avanzado") +
scale_fill_lancet() +
scale_color_lancet() +
theme_minimal()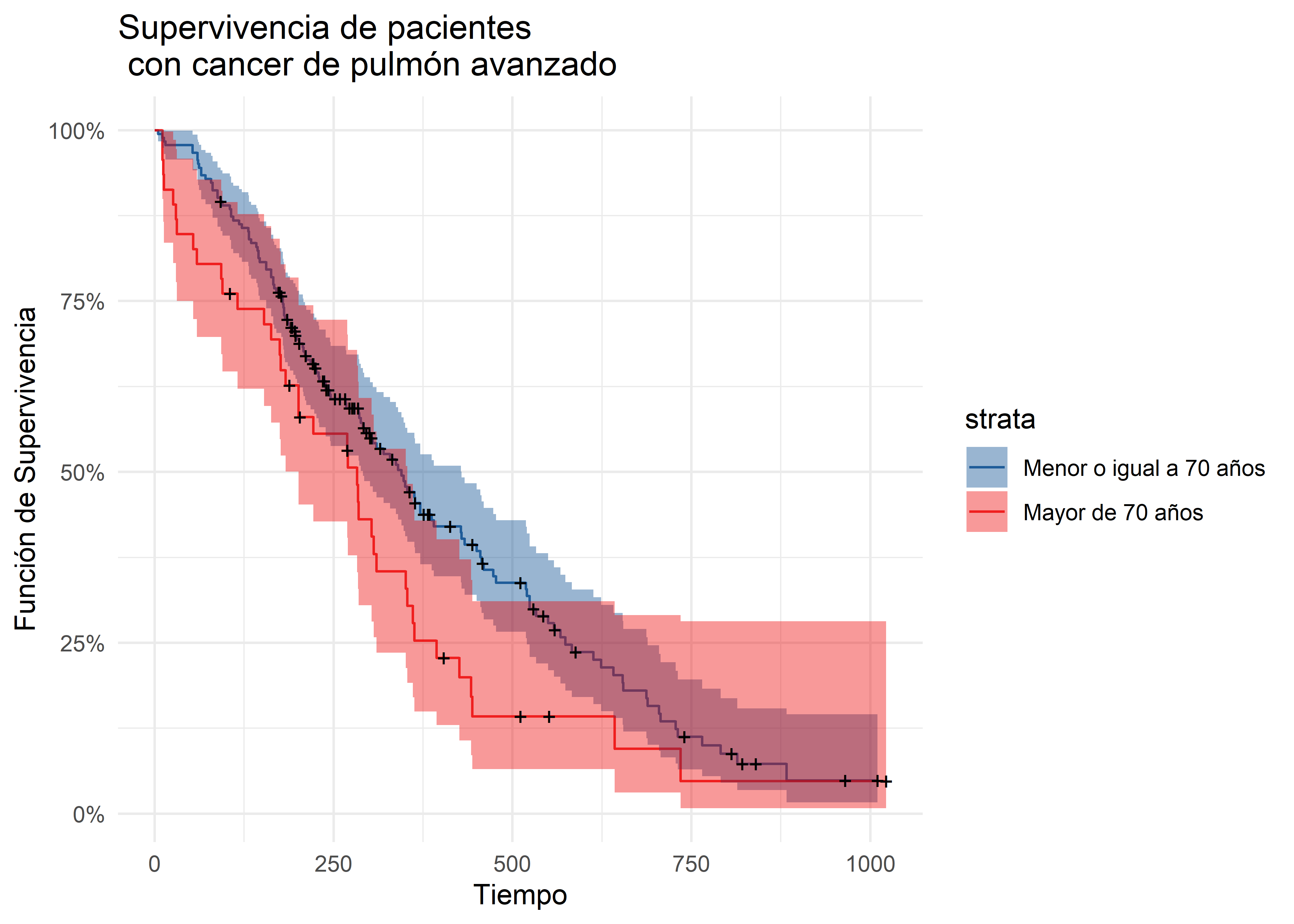
En el gráfico de Kaplan-Meier observamos los pacientes menores o iguales a 70 años llegan al 50% de supervivencia en el día 340, mientras que para el grupo de pacientes mayores a 70 años, el mismo valor de supervivenvia fue observado en el día 270.
Análisis multinivel
Ejercicio 1
Leamos y demos un vistazo a los datos:
nurses <- read_csv("data/nurses.csv")Rows: 1000 Columns: 11
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
dbl (11): hospital, ward, wardid, nurse, age, gender, experien, stress, ward...
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.View(nurses)Primero, transformemos las variables indicadoras en factores:
Para construir los modelos multinivel, carguemos el paquete lme4:
Loading required package: Matrix
Attaching package: 'Matrix'The following objects are masked from 'package:pracma':
expm, lu, tril, triuThe following objects are masked from 'package:tidyr':
expand, pack, unpack
Attaching package: 'lme4'The following object is masked from 'package:nlme':
lmListAjustemos el primer modelo con efectos aleatorios correlacionados para los coeficientes de expcon y wardype:
model_v1 <- lmer(
stress ~ age + gender + experien + expcon + wardtype + (expcon + wardtype|wardid),
data = nurses
)Warning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 1 negative eigenvaluessummary(model_v1)Linear mixed model fit by REML ['lmerMod']
Formula: stress ~ age + gender + experien + expcon + wardtype + (expcon +
wardtype | wardid)
Data: nurses
REML criterion at convergence: 1644.2
Scaled residuals:
Min 1Q Median 3Q Max
-2.75297 -0.70472 -0.00538 0.65276 2.80266
Random effects:
Groups Name Variance Std.Dev. Corr
wardid (Intercept) 0.2544 0.5043
expcon1 0.6708 0.8190 0.14
wardtype1 0.4295 0.6554 -0.72 -0.31
Residual 0.2174 0.4662
Number of obs: 1000, groups: wardid, 100
Fixed effects:
Estimate Std. Error t value
(Intercept) 5.762313 0.115759 49.778
age 0.022162 0.002199 10.078
gender1 -0.453350 0.034987 -12.958
experien -0.061755 0.004474 -13.804
expcon1 -0.695213 0.147271 -4.721
wardtype1 0.040844 0.125188 0.326
Correlation of Fixed Effects:
(Intr) age gendr1 expern expcn1
age -0.276
gender1 -0.225 -0.015
experien 0.000 -0.817 0.029
expcon1 -0.266 0.003 -0.002 0.001
wardtype1 -0.591 -0.002 -0.004 0.002 -0.051
optimizer (nloptwrap) convergence code: 0 (OK)
unable to evaluate scaled gradient
Model failed to converge: degenerate Hessian with 1 negative eigenvaluesVemos que el modelo ha fallado en converger. Usemos la función allFit() para encontrar el optimizador que logre la convergencia y la función glance() del paquete broom.mixed para mostrar los resultados en una tabla:
model_v1_allfit <- allFit(model_v1, verbose = FALSE)Warning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 1 negative eigenvaluesWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 2 negative eigenvaluesWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 1 negative eigenvaluesglance(model_v1_allfit)# A tibble: 5 × 9
optimizer nobs sigma logLik AIC BIC REMLcrit df.residual NLL_rel
<chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <dbl>
1 bobyqa 1000 0.466 -822. 1670. 1734. 1644. 987 0
2 Nelder_Mead 1000 0.466 -822. 1670. 1734. 1644. 987 9.66e-12
3 nlminbwrap 1000 0.466 -822. 1670. 1734. 1644. 987 7.50e-12
4 nloptwrap.NLOPT_… 1000 0.466 -822. 1670. 1734. 1644. 987 6.90e- 9
5 nloptwrap.NLOPT_… 1000 0.466 -822. 1670. 1734. 1644. 987 7.00e- 9Vemos que con el optimizador bobyqa se logra la convergencia (NLL_rel igual a 0). Reajustemos el modelo:
model_v1 <- lmer(
stress ~ age + gender + experien + expcon + wardtype + (expcon + wardtype|wardid),
data = nurses, control = lmerControl(optimizer = "bobyqa")
)
summary(model_v1)Linear mixed model fit by REML ['lmerMod']
Formula: stress ~ age + gender + experien + expcon + wardtype + (expcon +
wardtype | wardid)
Data: nurses
Control: lmerControl(optimizer = "bobyqa")
REML criterion at convergence: 1644.2
Scaled residuals:
Min 1Q Median 3Q Max
-2.75297 -0.70472 -0.00538 0.65276 2.80266
Random effects:
Groups Name Variance Std.Dev. Corr
wardid (Intercept) 0.2544 0.5043
expcon1 0.7575 0.8704 0.03
wardtype1 0.1677 0.4095 -0.52 -0.46
Residual 0.2174 0.4662
Number of obs: 1000, groups: wardid, 100
Fixed effects:
Estimate Std. Error t value
(Intercept) 5.762313 0.115759 49.778
age 0.022161 0.002199 10.078
gender1 -0.453350 0.034987 -12.958
experien -0.061755 0.004474 -13.804
expcon1 -0.695213 0.147271 -4.721
wardtype1 0.040844 0.125189 0.326
Correlation of Fixed Effects:
(Intr) age gendr1 expern expcn1
age -0.276
gender1 -0.225 -0.015
experien 0.000 -0.817 0.029
expcon1 -0.266 0.003 -0.002 0.001
wardtype1 -0.591 -0.002 -0.004 0.002 -0.051Para evaluar la significancia del modelo,
Computing bootstrap confidence intervals ...
4 message(s): boundary (singular) fit: see help('isSingular')
510 warning(s): Hessian is numerically singular: parameters are not uniquely determined (and others)Computing bootstrap confidence intervals ...
6 message(s): boundary (singular) fit: see help('isSingular')
539 warning(s): Hessian is numerically singular: parameters are not uniquely determined (and others)# A tibble: 13 × 8
effect group term estimate std.error statistic conf.low conf.high
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 fixed <NA> (Intercept) 5.76 0.116 49.8 5.57 5.99
2 fixed <NA> age 0.0222 0.00220 10.1 0.0182 0.0269
3 fixed <NA> gender1 -0.453 0.0350 -13.0 -0.520 -0.387
4 fixed <NA> experien -0.0618 0.00447 -13.8 -0.0715 -0.0529
5 fixed <NA> expcon1 -0.695 0.147 -4.72 -0.964 -0.384
6 fixed <NA> wardtype1 0.0408 0.125 0.326 -0.231 0.269
7 ran_pars wardid sd__(Inter… 0.504 NA NA 0.345 0.668
8 ran_pars wardid cor__(Inte… 0.0293 NA NA -0.339 0.349
9 ran_pars wardid cor__(Inte… -0.521 NA NA -0.833 -0.0806
10 ran_pars wardid sd__expcon1 0.870 NA NA 0.644 1.13
11 ran_pars wardid cor__expco… -0.460 NA NA -0.889 0.575
12 ran_pars wardid sd__wardty… 0.410 NA NA 0.281 0.708
13 ran_pars Residual sd__Observ… 0.466 NA NA 0.444 0.486 Utilizando el método Bootstrap, vemos que los efectos aleatorios de expcon y wardtype son significativos, dado que los intervalos de confianza no contienen el 0.
Ajustemos ahora el segundo modelo con efectos aleatorios no correlacionados para expcon y wardtype:
model_v2 <- lmer(
stress ~ age + gender + experien + (expcon|wardid) + (wardtype|wardid),
data = nurses
)Warning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 3 negative eigenvaluessummary(model_v2)Linear mixed model fit by REML ['lmerMod']
Formula: stress ~ age + gender + experien + (expcon | wardid) + (wardtype |
wardid)
Data: nurses
REML criterion at convergence: 1660.4
Scaled residuals:
Min 1Q Median 3Q Max
-2.71832 -0.70404 -0.00175 0.66250 2.83227
Random effects:
Groups Name Variance Std.Dev. Corr
wardid (Intercept) 0.17893 0.4230
expcon1 0.68068 0.8250 0.33
wardid.1 (Intercept) 0.09447 0.3074
wardtype1 0.19977 0.4470 -0.94
Residual 0.21735 0.4662
Number of obs: 1000, groups: wardid, 100
Fixed effects:
Estimate Std. Error t value
(Intercept) 5.655003 0.089447 63.22
age 0.022241 0.002199 10.11
gender1 -0.453405 0.034997 -12.96
experien -0.061849 0.004475 -13.82
Correlation of Fixed Effects:
(Intr) age gendr1
age -0.358
gender1 -0.295 -0.015
experien 0.002 -0.817 0.028
optimizer (nloptwrap) convergence code: 0 (OK)
unable to evaluate scaled gradient
Model failed to converge: degenerate Hessian with 3 negative eigenvaluesEl modelo no convergió. Busquemos el optimizador que logre la convergencia:
model_v2_allfit <- allFit(model_v2, verbose = FALSE)Warning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 3 negative eigenvaluesWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 2 negative eigenvaluesWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 3 negative eigenvaluesWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 3 negative eigenvaluesglance(model_v2_allfit)# A tibble: 5 × 9
optimizer nobs sigma logLik AIC BIC REMLcrit df.residual NLL_rel
<chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <dbl>
1 bobyqa 1000 0.466 -830. 1682. 1736. 1660. 989 0
2 Nelder_Mead 1000 0.466 -830. 1682. 1736. 1660. 989 1.02e-10
3 nlminbwrap 1000 0.466 -830. 1682. 1736. 1660. 989 3.02e-11
4 nloptwrap.NLOPT_… 1000 0.466 -830. 1682. 1736. 1660. 989 2.67e- 9
5 nloptwrap.NLOPT_… 1000 0.466 -830. 1682. 1736. 1660. 989 1.33e-10En este caso, fue el optimziador bobyqa. Reajustemos el modelo:
model_v2 <- lmer(
stress ~ age + gender + experien + (expcon|wardid) + (wardtype|wardid),
data = nurses, control = lmerControl(optimizer = "bobyqa")
)
summary(model_v2)Linear mixed model fit by REML ['lmerMod']
Formula: stress ~ age + gender + experien + (expcon | wardid) + (wardtype |
wardid)
Data: nurses
Control: lmerControl(optimizer = "bobyqa")
REML criterion at convergence: 1660.4
Scaled residuals:
Min 1Q Median 3Q Max
-2.71832 -0.70404 -0.00175 0.66250 2.83227
Random effects:
Groups Name Variance Std.Dev. Corr
wardid (Intercept) 0.19693 0.4438
expcon1 0.78029 0.8833 0.16
wardid.1 (Intercept) 0.07646 0.2765
wardtype1 0.12532 0.3540 -0.94
Residual 0.21735 0.4662
Number of obs: 1000, groups: wardid, 100
Fixed effects:
Estimate Std. Error t value
(Intercept) 5.655003 0.089447 63.22
age 0.022241 0.002199 10.11
gender1 -0.453405 0.034997 -12.96
experien -0.061849 0.004475 -13.82
Correlation of Fixed Effects:
(Intr) age gendr1
age -0.358
gender1 -0.295 -0.015
experien 0.002 -0.817 0.028Evaluemos la significancia de los efectos aleatorios:
Computing bootstrap confidence intervals ...
14 message(s): boundary (singular) fit: see help('isSingular')
554 warning(s): Hessian is numerically singular: parameters are not uniquely determined (and others)Computing bootstrap confidence intervals ...
14 message(s): boundary (singular) fit: see help('isSingular')
574 warning(s): Hessian is numerically singular: parameters are not uniquely determined (and others)# A tibble: 11 × 8
effect group term estimate std.error statistic conf.low conf.high
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 fixed <NA> (Intercept) 5.66 0.0894 63.2 5.48 5.82
2 fixed <NA> age 0.0222 0.00220 10.1 0.0182 0.0269
3 fixed <NA> gender1 -0.453 0.0350 -13.0 -0.520 -0.387
4 fixed <NA> experien -0.0618 0.00448 -13.8 -0.0707 -0.0532
5 ran_pars wardid sd__(Inter… 0.444 NA NA 0.279 0.516
6 ran_pars wardid cor__(Inte… 0.163 NA NA -0.00265 0.372
7 ran_pars wardid sd__expcon1 0.883 NA NA 0.675 1.13
8 ran_pars wardid.1 sd__(Inter… 0.277 NA NA 0.00183 0.552
9 ran_pars wardid.1 cor__(Inte… -0.936 NA NA -1.00 -0.401
10 ran_pars wardid.1 sd__wardty… 0.354 NA NA 0.202 0.515
11 ran_pars Residual sd__Observ… 0.466 NA NA 0.444 0.485 Vemos que han habido algunos problemas de convergencia a la hora de calcular los intervalos de confianza mediante Bootstrap, por lo que no podemos estar totalmente seguros que estos resultados sean confiables. Si obviamos estos problemas, solo para efectos prácticamos, podemos observar que los efectos aleatorios no correlacionados de expcon y wardtype son significativos.
Ejercicio 2
Podemos ajustar el modelo de tres niveles con interceptos aleatorios para los pabellones dentro de cada hospital y para los hospitales usando las siguiente líneas de código:
Linear mixed model fit by REML ['lmerMod']
Formula: stress ~ (1 | hospital/ward)
Data: nurses
REML criterion at convergence: 1945
Scaled residuals:
Min 1Q Median 3Q Max
-2.97604 -0.65739 0.01792 0.67398 3.11229
Random effects:
Groups Name Variance Std.Dev.
ward:hospital (Intercept) 0.4888 0.6992
hospital (Intercept) 0.1743 0.4175
Residual 0.3013 0.5489
Number of obs: 1000, groups: ward:hospital, 100; hospital, 25
Fixed effects:
Estimate Std. Error t value
(Intercept) 5.0010 0.1103 45.34Nótese que se usa el identificador ward y no wardid con en los ejercicios anteriores. La razón es que los valores de wardid no están anidados dentro de los valores de hospital, ya que esta variable identifica a cada pabellón idenpendientemente del hospital al que pertenece.
Por otro lado, en la salida del modelo vemos que el efecto aleatorio (1|hospital/ward) se expande en los efectos aleatorios del hospital ((1|hospital)) y del pabellón relativo a cada hospital ((1|hospital:ward)). Por esta razón, también podemos ajustar este modelo la siguiente manera:
model_ri_3lvl <- lmer(stress ~ (1|hospital) + (1|hospital:ward), data = nurses)
summary(model_ri_3lvl)Linear mixed model fit by REML ['lmerMod']
Formula: stress ~ (1 | hospital) + (1 | hospital:ward)
Data: nurses
REML criterion at convergence: 1945
Scaled residuals:
Min 1Q Median 3Q Max
-2.97604 -0.65739 0.01792 0.67398 3.11229
Random effects:
Groups Name Variance Std.Dev.
hospital:ward (Intercept) 0.4888 0.6992
hospital (Intercept) 0.1743 0.4175
Residual 0.3013 0.5489
Number of obs: 1000, groups: hospital:ward, 100; hospital, 25
Fixed effects:
Estimate Std. Error t value
(Intercept) 5.0010 0.1103 45.34Ahora, ajustemos el modelo de dos niveles con interceptos aleatorios para los pabellones (indistintamente de a qué hospital pertenecen) para hacer la comparación:
Linear mixed model fit by REML ['lmerMod']
Formula: stress ~ (1 | wardid)
Data: nurses
REML criterion at convergence: 1952.4
Scaled residuals:
Min 1Q Median 3Q Max
-2.94699 -0.67691 -0.00005 0.69678 3.12110
Random effects:
Groups Name Variance Std.Dev.
wardid (Intercept) 0.6576 0.8109
Residual 0.3013 0.5489
Number of obs: 1000, groups: wardid, 100
Fixed effects:
Estimate Std. Error t value
(Intercept) 5.00065 0.08295 60.28Para calcular el coeficiente de correlación interclase (ICC) usamos la función icc() del paquete performance. Primero, para el modelo de dos niveles:
library(performance)
icc(model_ri_2lvl)# Intraclass Correlation Coefficient
Adjusted ICC: 0.686
Unadjusted ICC: 0.686Ahora, para el modelo de tres niveles:
icc(model_ri_3lvl)# Intraclass Correlation Coefficient
Adjusted ICC: 0.688
Unadjusted ICC: 0.688Vemos que la proporción de varianza explicada por los factores de agrupación (niveles) en el modelo de tres niveles es mayor que la de los factores de agrupación en el modelo de dos niveles. Podemos también separa el ICC por los factores de agrupación de a partir del segundo nivel:
icc(model_ri_3lvl, by_group = TRUE)# ICC by Group
Group | ICC
---------------------
hospital:ward | 0.507
hospital | 0.181Vemos que la proporción de varianza explicada por pabellones dentro de cada hospital es mayor que la explicada por los hospitales.
Por último, para comparar la calidad de ajuste de los modelos, podemos usar la función anova():
anova(model_ri_2lvl, model_ri_3lvl)refitting model(s) with ML (instead of REML)Data: nurses
Models:
model_ri_2lvl: stress ~ (1 | wardid)
model_ri_3lvl: stress ~ (1 | hospital) + (1 | hospital:ward)
npar AIC BIC logLik deviance Chisq Df Pr(>Chisq)
model_ri_2lvl 3 1955.3 1970 -974.64 1949.3
model_ri_3lvl 4 1950.4 1970 -971.18 1942.4 6.9195 1 0.008526 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Podemos observar que el AIC del modelo de tres niveles es menor que el del modelo de dos niveles. Por tanto, el primero brinda un mejor ajuste. Además, dado que los modelos son anidados, podemos evaluar la significancia del modelo de tres niveles con respecto al de dos niveles. En este caso, vemos que el p-valor de la prueba ji al cuadrado es significativo y, por tanto, el modelo de tres niveles es significativo sobre el de dos niveles.
Ejercicio 3
Para que al función lmer() devuelva las pruebas de significancia para los efectos fijos necesitamos llamar antes al paquete lmerTest:
Attaching package: 'lmerTest'The following object is masked from 'package:lme4':
lmerThe following object is masked from 'package:pracma':
randThe following object is masked from 'package:stats':
stepAhora, ajustamos el modelo con las espeficicaciones indicadas en el ejercicio:
model_full_3lvl <- lmer(
stress ~ age + gender + experien + expcon + wardtype + hospsize + (expcon|hospital) +
(wardtype|hospital) + (hospsize|hospital:ward), data = nurses
)Warning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 1 negative eigenvaluesWarning: Model failed to converge with 1 negative eigenvalue: -3.0e-05summary(model_full_3lvl)Linear mixed model fit by REML. t-tests use Satterthwaite's method [
lmerModLmerTest]
Formula: stress ~ age + gender + experien + expcon + wardtype + hospsize +
(expcon | hospital) + (wardtype | hospital) + (hospsize |
hospital:ward)
Data: nurses
REML criterion at convergence: 1608.4
Scaled residuals:
Min 1Q Median 3Q Max
-2.66405 -0.69679 -0.00233 0.63510 2.76914
Random effects:
Groups Name Variance Std.Dev. Corr
hospital.ward (Intercept) 0.10032 0.3167
hospsize 0.06596 0.2568 -0.56
hospital (Intercept) 0.08239 0.2870
wardtype1 0.03144 0.1773 -0.10
hospital.1 (Intercept) 0.11122 0.3335
expcon1 0.64523 0.8033 -0.62
Residual 0.21733 0.4662
Number of obs: 1000, groups: hospital:ward, 100; hospital, 25
Fixed effects:
Estimate Std. Error df t value Pr(>|t|)
(Intercept) 5.523071 0.158393 29.170334 34.869 < 2e-16 ***
age 0.022235 0.002196 909.104890 10.123 < 2e-16 ***
gender1 -0.453315 0.034894 914.051118 -12.991 < 2e-16 ***
experien -0.061739 0.004464 915.765552 -13.831 < 2e-16 ***
expcon1 -0.711134 0.175172 20.524193 -4.060 0.000586 ***
wardtype1 0.081922 0.077054 13.139514 1.063 0.306852
hospsize 0.277322 0.129052 22.163320 2.149 0.042818 *
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Correlation of Fixed Effects:
(Intr) age gendr1 expern expcn1 wrdty1
age -0.197
gender1 -0.168 -0.013
experien -0.004 -0.817 0.028
expcon1 -0.324 0.003 -0.002 0.000
wardtype1 -0.206 -0.012 -0.001 0.014 0.000
hospsize -0.633 -0.005 0.003 0.004 -0.008 -0.004
optimizer (nloptwrap) convergence code: 0 (OK)
unable to evaluate scaled gradient
Model failed to converge: degenerate Hessian with 1 negative eigenvaluesVemos que el modelo no ha convergido. Evaluemos otros optimizadores:
model_full_3lvl_allfit <- allFit(model_full_3lvl, verbose = FALSE)Warning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
unable to evaluate scaled gradientWarning in checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge: degenerate Hessian with 1 negative eigenvaluesWarning: Model failed to converge with 1 negative eigenvalue: -3.0e-05glance(model_full_3lvl_allfit)# A tibble: 5 × 9
optimizer nobs sigma logLik AIC BIC REMLcrit df.residual NLL_rel
<chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <dbl>
1 bobyqa 1000 0.466 -804. 1642. 1726. 1608. 983 0
2 Nelder_Mead 1000 0.466 -804. 1642. 1726. 1608. 983 4.88e-11
3 nlminbwrap 1000 0.466 -804. 1642. 1726. 1608. 983 7.64e- 9
4 nloptwrap.NLOPT_… 1000 0.466 -804. 1642. 1726. 1608. 983 1.04e- 8
5 nloptwrap.NLOPT_… 1000 0.466 -804. 1642. 1726. 1608. 983 2.78e- 8El optimizador con el que se logra la convergencia es bobyqa. Reajustemos el modelo:
model_full_3lvl <- lmer(
stress ~ age + gender + experien + expcon + wardtype + hospsize + (expcon|hospital) +
(wardtype|hospital) + (hospsize|hospital:ward),
data = nurses, control = lmerControl(optimizer = "bobyqa")
)
summary(model_full_3lvl)Linear mixed model fit by REML. t-tests use Satterthwaite's method [
lmerModLmerTest]
Formula: stress ~ age + gender + experien + expcon + wardtype + hospsize +
(expcon | hospital) + (wardtype | hospital) + (hospsize |
hospital:ward)
Data: nurses
Control: lmerControl(optimizer = "bobyqa")
REML criterion at convergence: 1608.4
Scaled residuals:
Min 1Q Median 3Q Max
-2.66405 -0.69679 -0.00233 0.63510 2.76914
Random effects:
Groups Name Variance Std.Dev. Corr
hospital.ward (Intercept) 0.10033 0.3167
hospsize 0.06597 0.2569 -0.56
hospital (Intercept) 0.01405 0.1185
wardtype1 0.03145 0.1773 -0.25
hospital.1 (Intercept) 0.17955 0.4237
expcon1 0.64520 0.8032 -0.49
Residual 0.21733 0.4662
Number of obs: 1000, groups: hospital:ward, 100; hospital, 25
Fixed effects:
Estimate Std. Error df t value Pr(>|t|)
(Intercept) 5.523076 0.158393 29.159813 34.870 < 2e-16 ***
age 0.022235 0.002196 909.104788 10.123 < 2e-16 ***
gender1 -0.453315 0.034894 914.051154 -12.991 < 2e-16 ***
experien -0.061739 0.004464 915.765535 -13.831 < 2e-16 ***
expcon1 -0.711134 0.175168 20.523124 -4.060 0.000586 ***
wardtype1 0.081922 0.077056 13.121689 1.063 0.306892
hospsize 0.277315 0.129053 22.157302 2.149 0.042827 *
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Correlation of Fixed Effects:
(Intr) age gendr1 expern expcn1 wrdty1
age -0.197
gender1 -0.168 -0.013
experien -0.004 -0.817 0.028
expcon1 -0.324 0.003 -0.002 0.000
wardtype1 -0.206 -0.012 -0.001 0.014 0.000
hospsize -0.633 -0.005 0.003 0.004 -0.008 -0.004De los resultados del modelo, vemos que todos los efectos fijos son significativos con excepción del efecto fijo del tipo de pabellón (wardtype).
Ahora, comparemos este modelo y el modelo de tres niveles con solo interceptos aleatorios:
anova(model_ri_3lvl, model_full_3lvl)refitting model(s) with ML (instead of REML)Data: nurses
Models:
model_ri_3lvl: stress ~ (1 | hospital) + (1 | hospital:ward)
model_full_3lvl: stress ~ age + gender + experien + expcon + wardtype + hospsize + (expcon | hospital) + (wardtype | hospital) + (hospsize | hospital:ward)
npar AIC BIC logLik deviance Chisq Df Pr(>Chisq)
model_ri_3lvl 4 1950.4 1970.0 -971.18 1942.4
model_full_3lvl 17 1606.7 1690.1 -786.35 1572.7 369.67 13 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Vemos que, según el AIC, este modelo con efectos mixtos tiene un mucho mejor ajuste que el modelo anterior con solo interceptos aleatorios. Además, la prueba de significancia resultó significativa, por lo que tenemos evidencia estadística para afirmar que el modelo de tres niveles con efectos mixtos brinda un mejor ajuste que el modelo de tres niveles con interceptos aleatorios.