5 Visualización de datos con ggplot2
5.1 Paquetes y data
En esta sección, utilizaremos los comandos de tidyverse de la plataforma R para la visualización de datos. Los paquetes y conjuntos de datos a ser utilizados en esta sección son:
El conjunto de datos who contiene datos del Global Health Observatory de la Organización Mundial de la Salud (OMS). Contiene 26 variables para 202 países y territorios. El diccionario de este conjunto de datos se encuentra en Anexos.
El motor de construcción de gráficos de tidyverse se llama ggplot2 e implementa la gramática de gráficos en capas (layered grammar of graphics) propuesta por Wickham (2010), una variación de la gramática de gráficos previamente descrita por Wilkinson (2012).
Para construir un gráfico con ggplot2 se necesita definir tres elementos básicos:
- Datos
- Estética (aesthetics)
- Geometría
Opcionalmente, también podemos definir:
- Escalas (scales)
- Temas (themes)
- Facetas (facets)
Siguiendo la estructura gramatical del tidyverse, un ejemplo del código básico para un gráfico sería el siguiente:
datos %>% # Definimos los datos
ggplot(aes(x, y)) + # Definimos la estética
geom_*() # Definimos la geometríaA lo largo de este libro, abordaremos la mayoría de elementos en la gramática de gráficos en capas. Para un panorama general, leer el artículo de Dipanjan (DJ) Sarkar, de donde proviene el siguiente gráfico con los elementos principales de la gramática de gráficos:
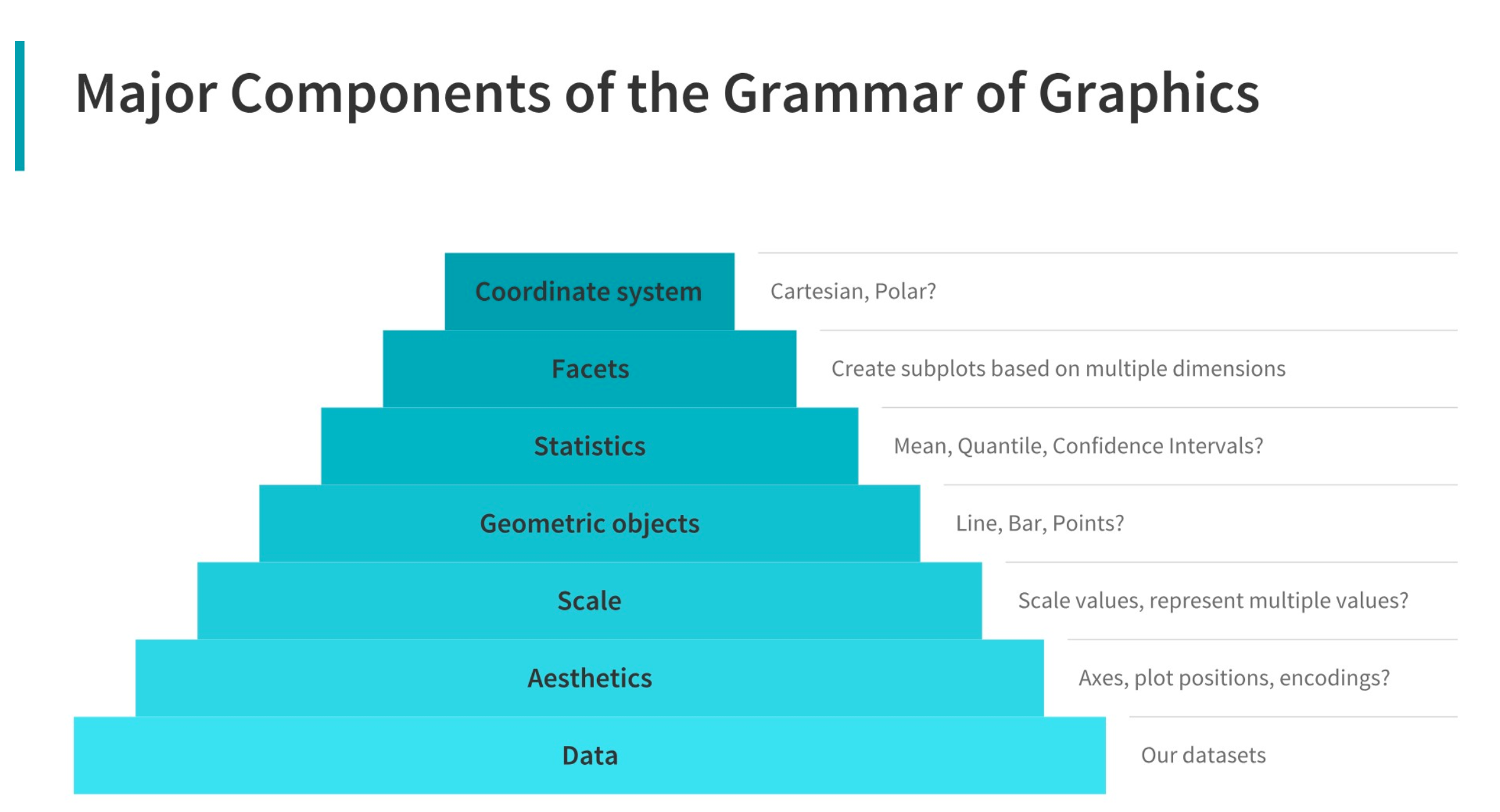
5.2 Definición de elementos
Utilizaremos el conjunto de datos who para mostrar los elementos.
- Los datos
Para mantener las buenas prácticas de los pipelines de análisis y visualización de datos, el conjunto de datos lo definiremos inicialmente y la conectaremos con el operador %>%.
El conjunto de datos incluida dentro ggplot (ya sea como un argumento o un elementro predecesor) gobierna los datos que se utilizan en todas la geometrías y facetas, a menos que se defina lo contrario dentro de cada geometría y/o faceta en específico*
Vemos un gráfico en blanco porque ggplot2 no tiene información de qué datos graficar.
- La estética
La estética está definida por el argumento aes(). Los elementos dentro de la estética (atributos) son parte de los datos y serán mapeados al gráfico en diferente representaciones (ej. geometrías).
Exploraremos la relación entre el índice de desigualdad (inequality_index) y la esperanza de vida al nacer (life_expectancy_birth) de cada país.
Ahora el gráfico muestra los ejes con los valores de ambas variables (con los rangos correspondientes en cada eje). Es decir, los datos ya están mapeados al gráfico. Sin embargo, no hemos definido qué representación visual se va a utilizar (en otras palabras, la geometría).
- La geometría
ggplot2 tiene varias geometrías que se pueden explorar en los cheatsheets. Hay múltiples paquetes que agregan una gran variedad de geometrías adicionales a las de ggplot2. Para el caso de la relación de dos variables numéricas, utilizaremos una geometría de puntos.
Recordar que después de la función
ggplot(), los siguientes elementos del gráfico son vinculados con un+en vez de un%>%.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth)) +
geom_point()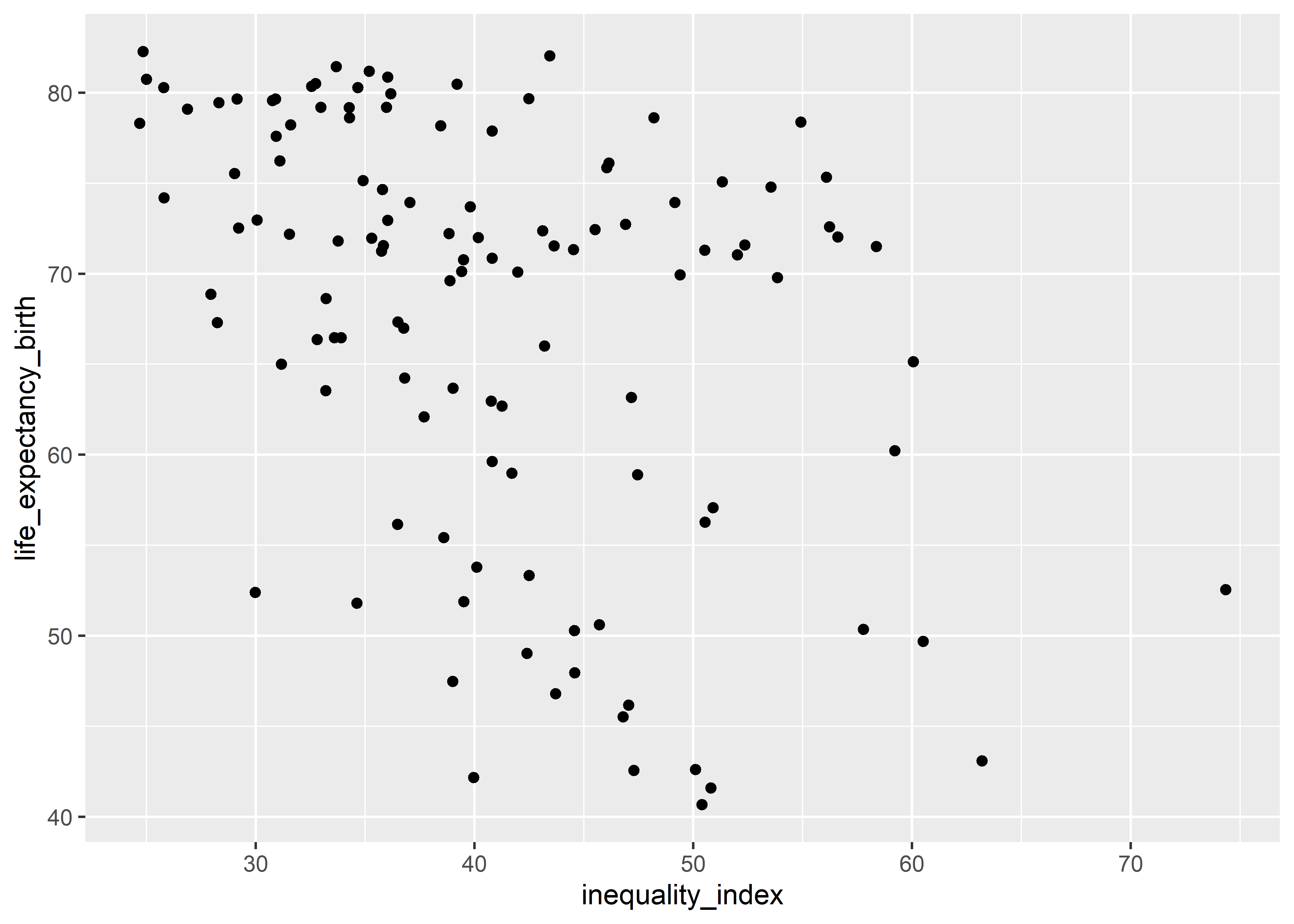
5.3 Elementos de aes()
Usando la misma estructura, ggplot2 permite agregar múltiples atributos para explorar más a profundidad los datos.
Al parecer hay una relación inversa pero dispersa entre el índice de desigualdad (inequality_index) y la esperanza de vida al nacer (life_expectancy_birth). Sin embargo, es posible que dentro de cada continente la relación sea diferente en magnitud o dirección (paradoja de Simpson). Para agregar mayor contexto al gráfico, podemos añadir colores a los puntos según el continente (continent) al que pertenecen. En este caso, el continente vendría a ser un atributo, ya que el color de los puntos va a estar en función de esta variable, y por tanto tiene que ir dentro de la estética aes().
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth, col = continent)) +
geom_point()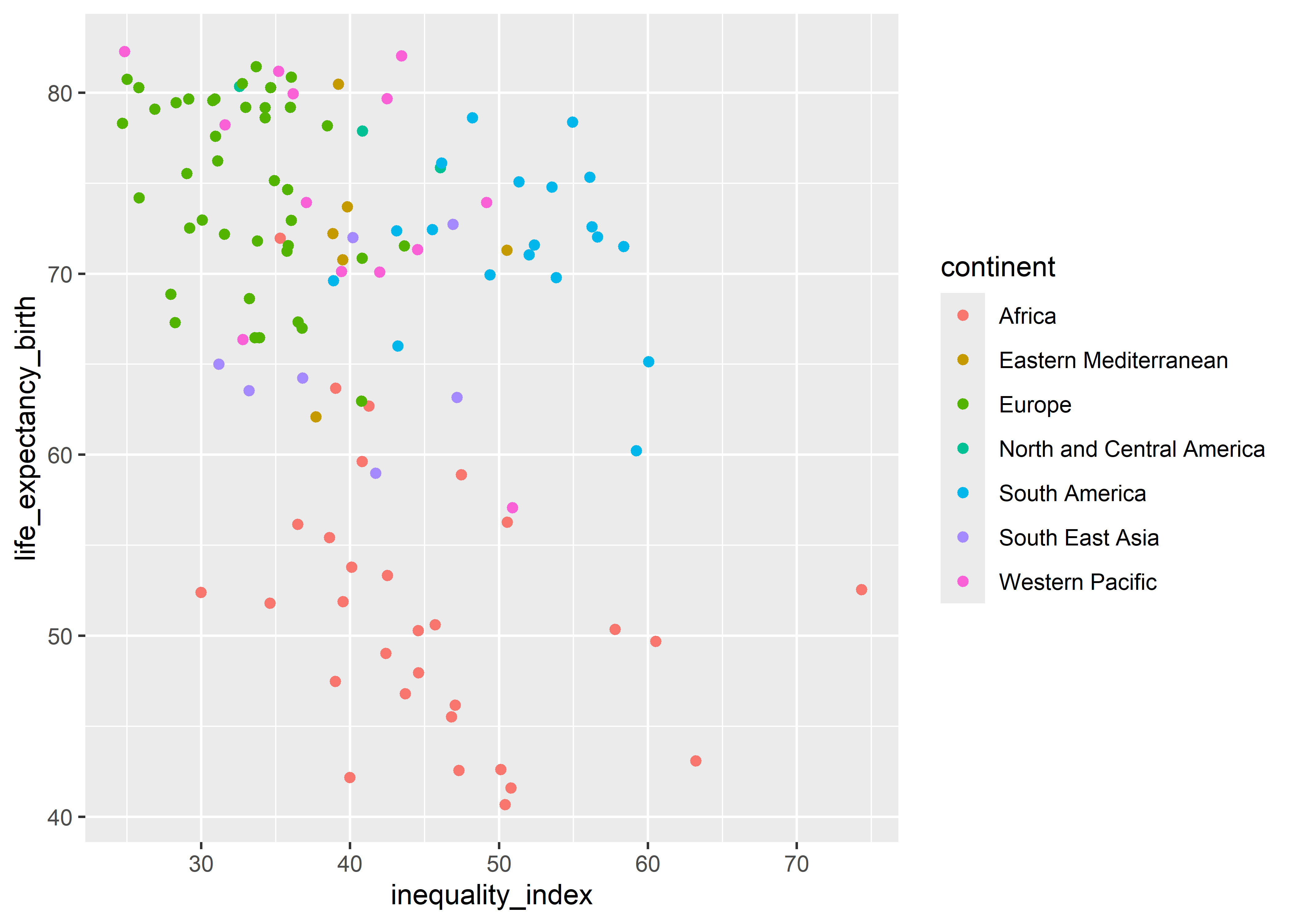
Vemos que, definitivamente, la magnitud y la dirección de la asociación entre el índice de desigualdad y la expectativa de vida al nacer es diferente para cada continente. Ahora, podríamos estar interesados también en ver la relación de estas variables con la cantidad de CO2 emitida por la actividad económica (co2_economic_output). Una forma de agregar esta variable adicional al gráfico es definirla como un atributo sobre alguna otra estética de los puntos. Agreguemos co2_economic_output como atributo para el tamaño de los puntos usando el argumento size.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point()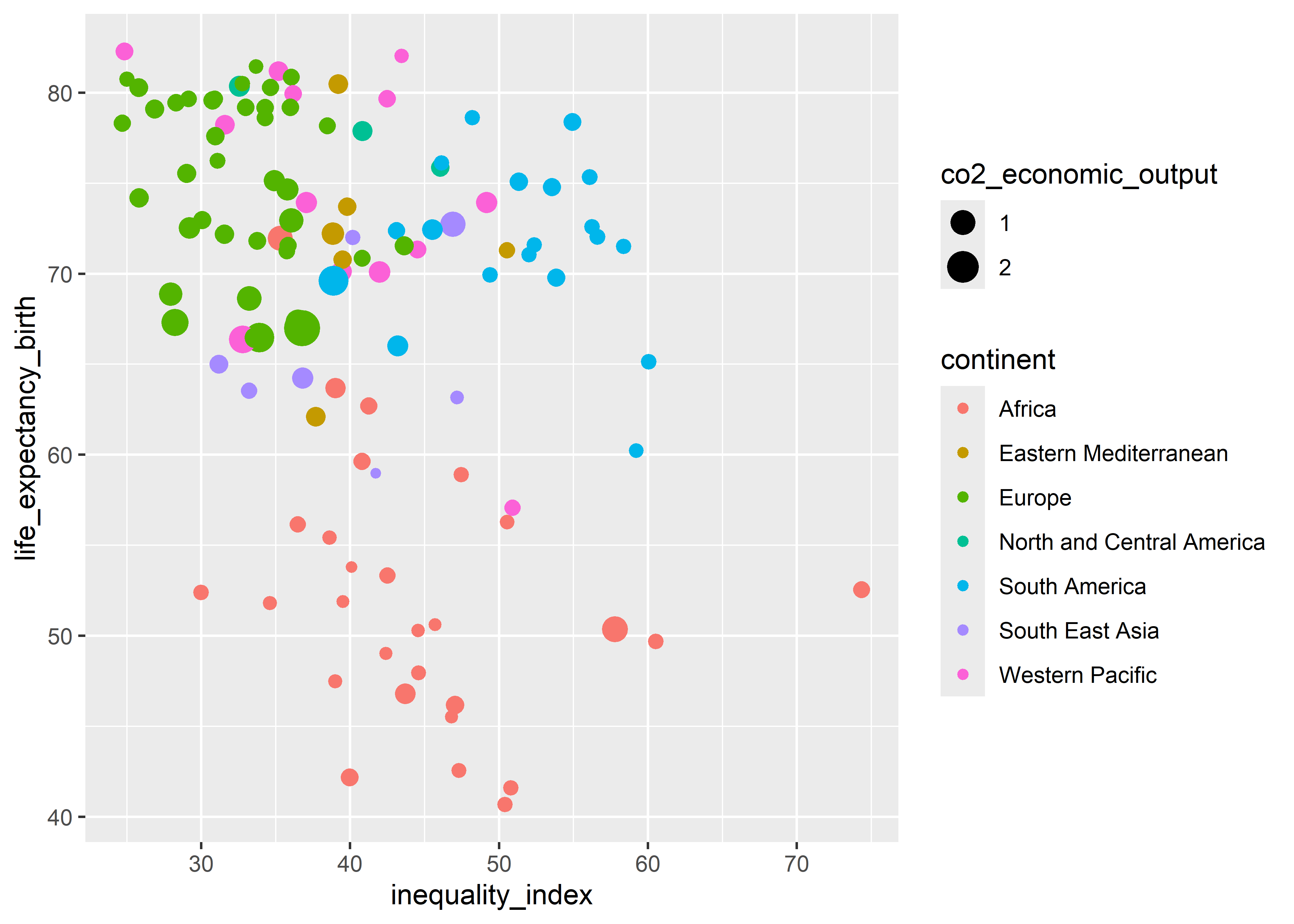
Podemos ver ahora, por ejemplo, que Europa tiene países con menor índice de desigualdad y alta expectativa de vida al nacer, pero la cantidad de CO2 que emiten los países por la actividad económica es bastante alto en comparación a los otros continentes.
En este ejemplo, la estética de cada punto está en función de inequality_index, life_expectancy_birth, continent y co2_economic_output y, por tanto, van dentro de aes().
5.4 Capas de geometrías
Ahora visualizaremos la relación entre una variable numérica y una variable categórica. Analizaremos las diferencias en la distribución de la esperanza de vida al nacer (life_expectancy_birth) en cada continente (continent).
Podemos empezar explorando la distribución de life_expectancy_birth global con un histograma.
who %>%
ggplot(aes(x = life_expectancy_birth)) +
geom_histogram()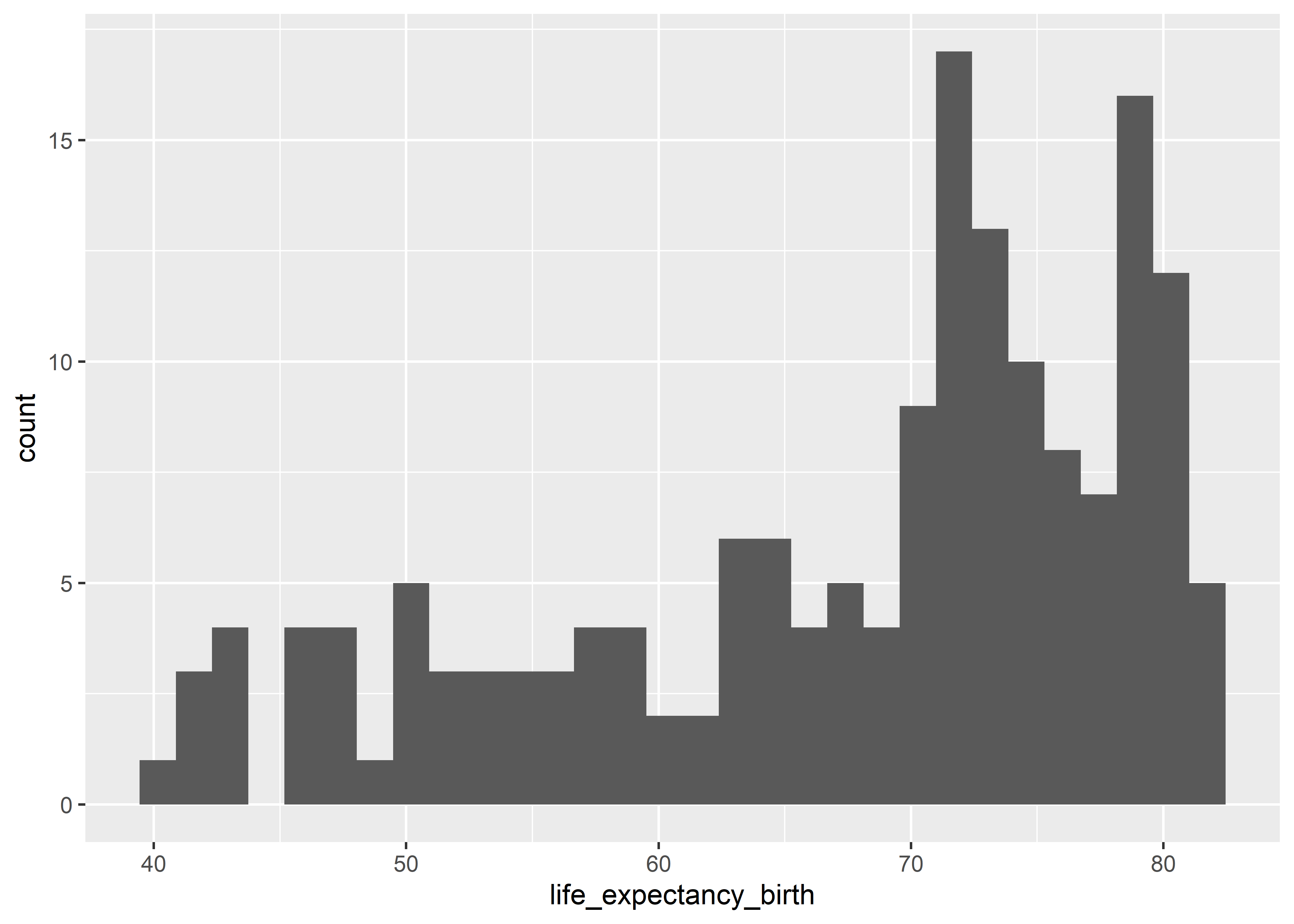
Podemos agregar una geometría de boxplot para comparar ambos gráficos usando los mismos datos y estética.
who %>%
ggplot(aes(x = life_expectancy_birth)) +
geom_histogram() +
geom_boxplot()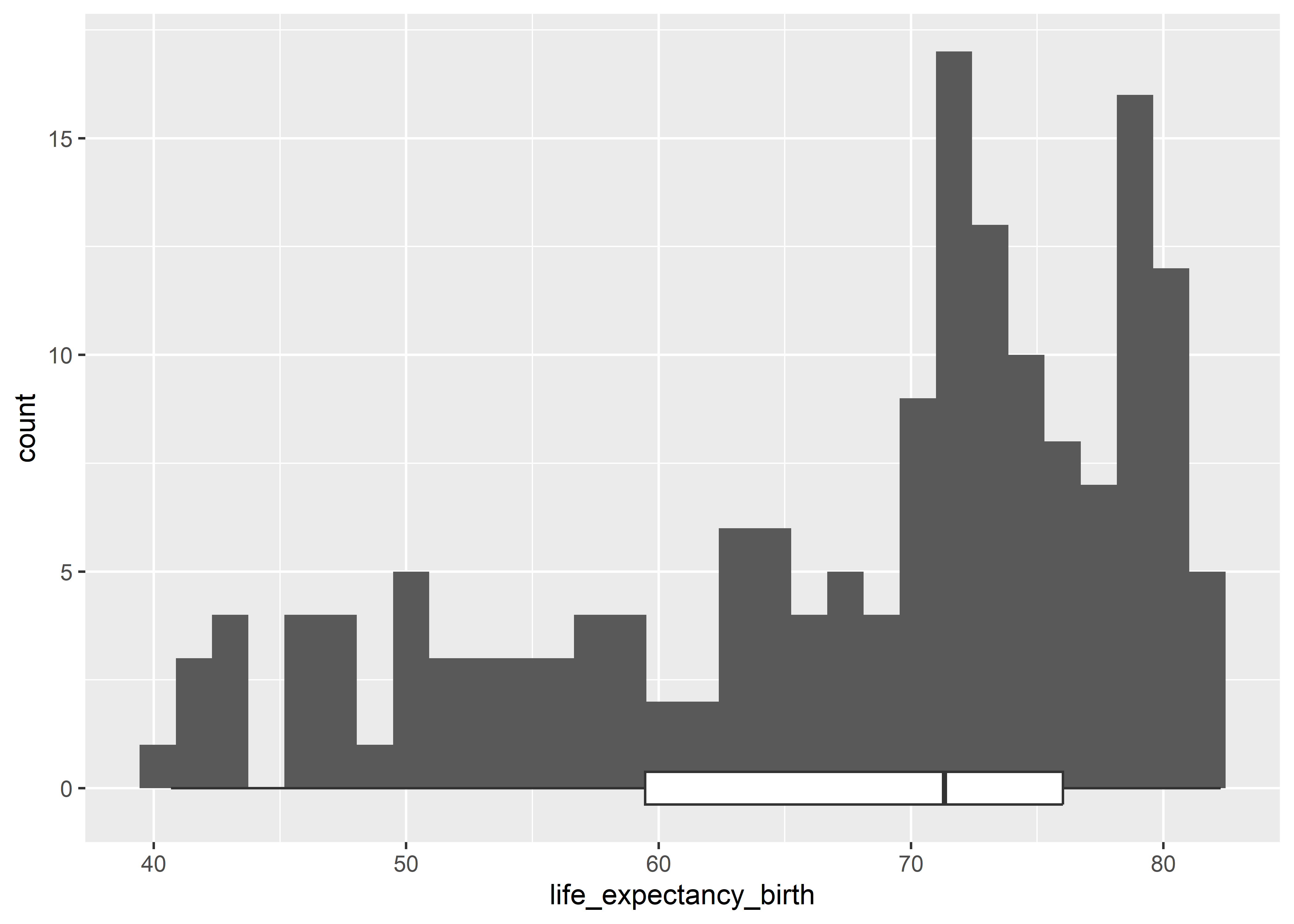
Ahora que hemos explorado las similitudes entre las representaciones visuales (geometrías) podemos expandir la exploración de datos.
Empezamos con la distribución global de la esperanza de vida al nacer usando una geometría de box plot.
who %>%
ggplot(aes(x = life_expectancy_birth)) +
geom_boxplot()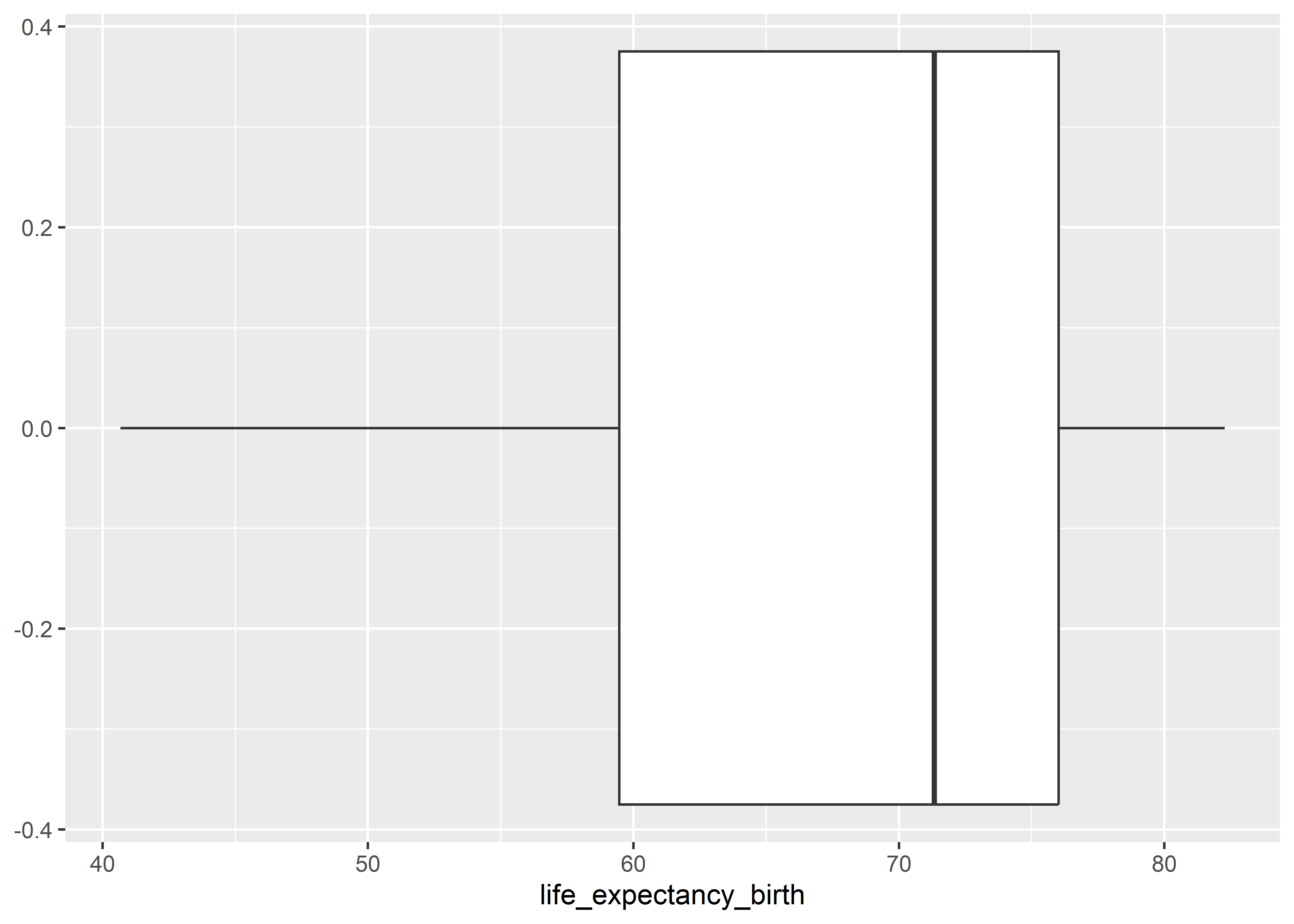
Expandimos esta exploración y agregamos un atributo para mapear esta distribución en los diferentes continentes.
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent)) +
geom_boxplot()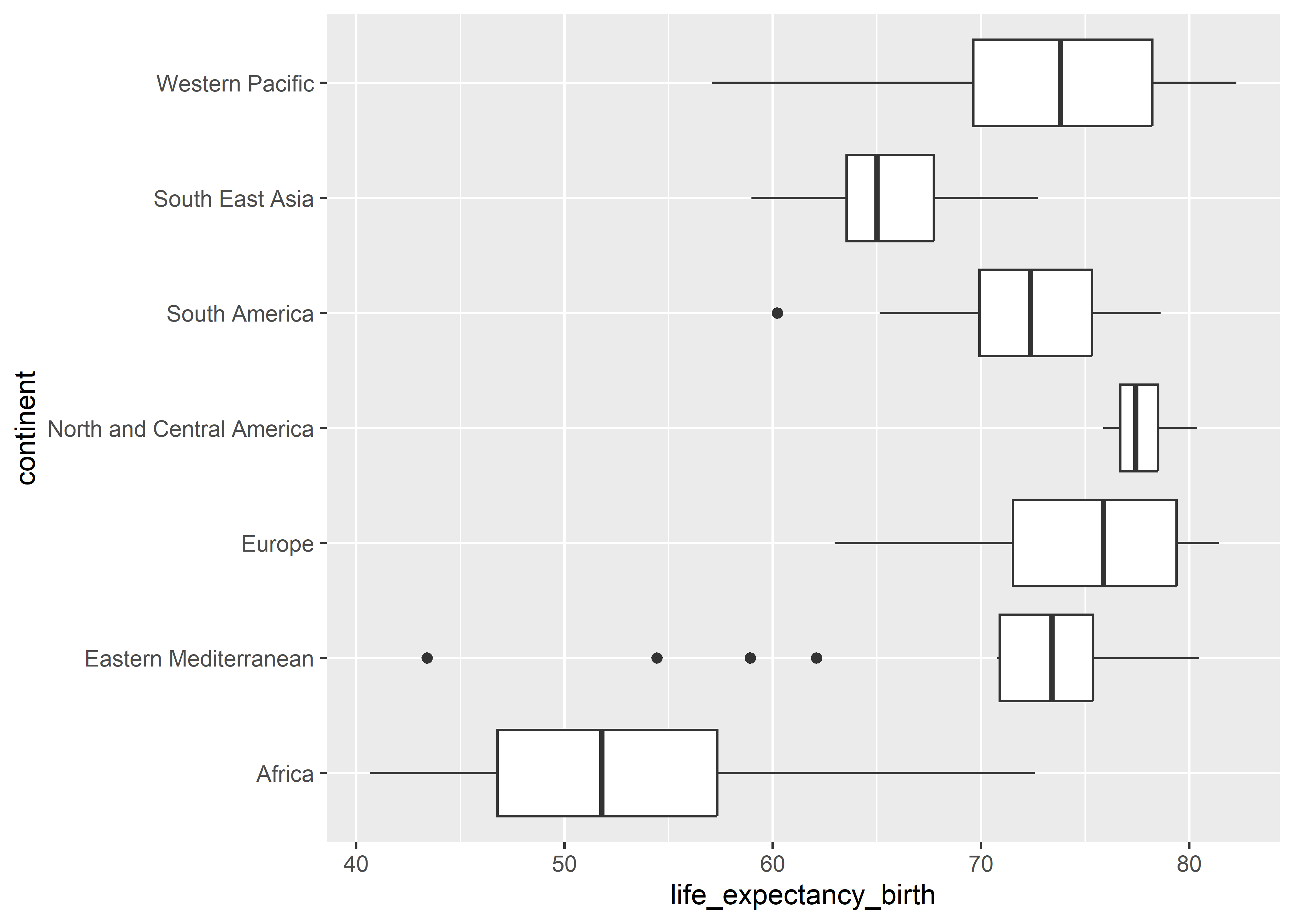
Por último, podemos agregar una capa de puntos para profundizar en la distribución de los países. La última capa en ser agregada se sobrepondrá al resto en la visualización.
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent)) +
geom_boxplot() +
geom_point()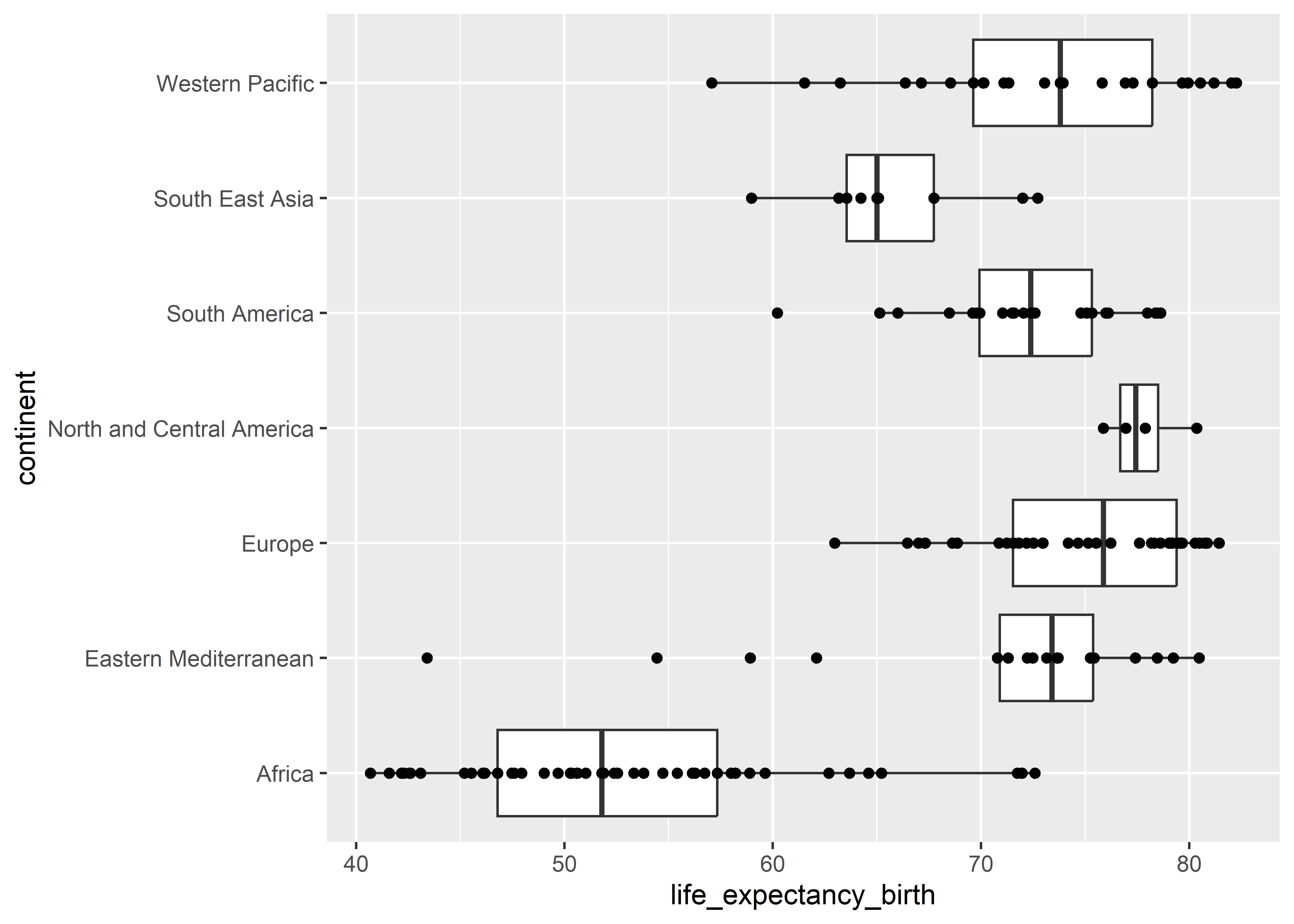
5.5 Especificaciones locales y globales
Los atributos del gráfico pueden especificarse de forma exclusiva para algunas (local) o para todas (global) las geometrías del gráfico.
Cuando la estética está definida dentro de la función ggplot(), gobierna la estética de las geometrías y otras funciones posteriores. Se puede usar una combinación de estéticas globales y estéticas específicas (locales) para cada geometría.
Podemos definir el color de continentes de forma local en la geometría de box plot.
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent)) +
geom_boxplot(aes(color = continent)) +
geom_point()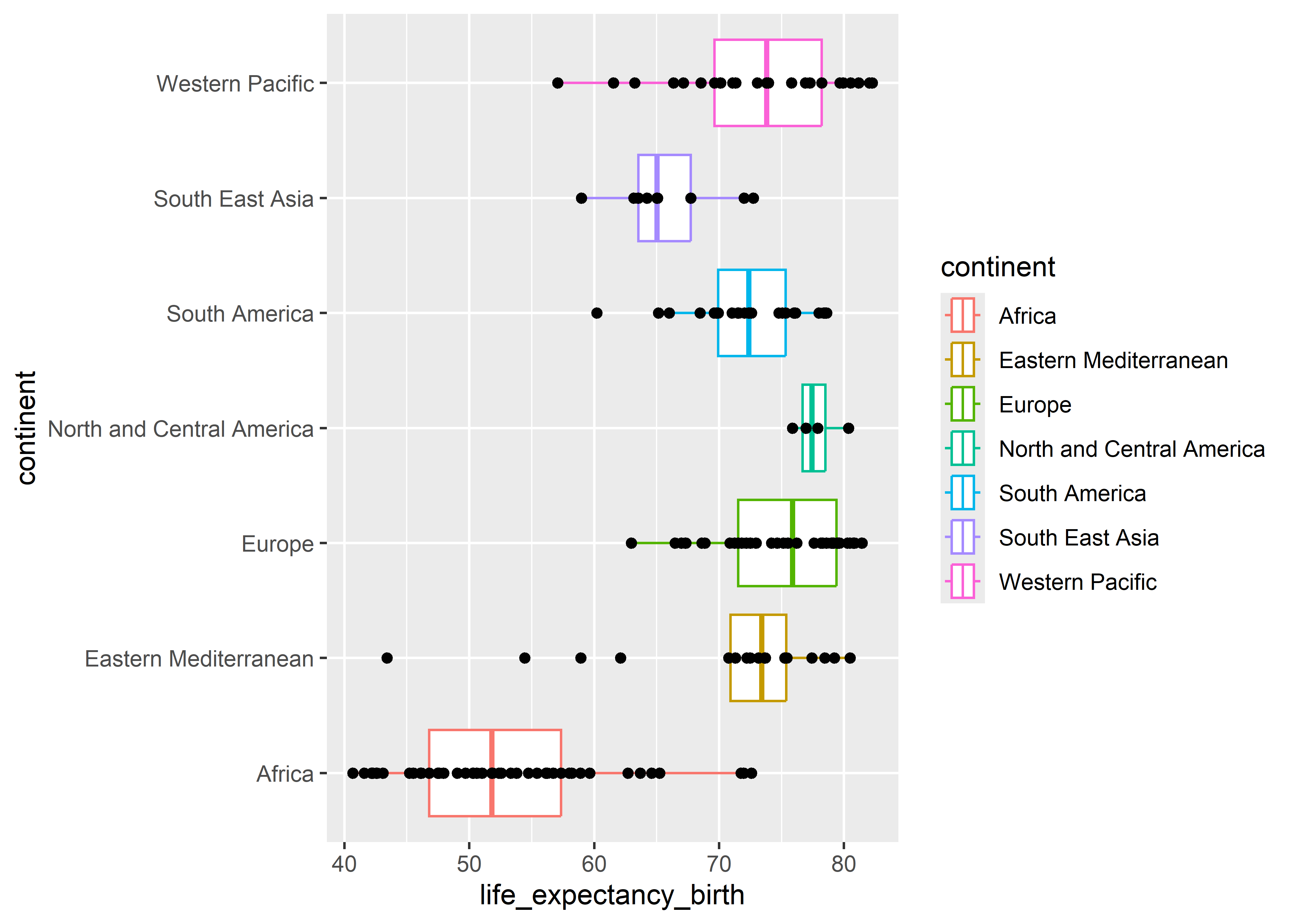
O en la geometría de puntos.
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent)) +
geom_boxplot() +
geom_point(aes(color = continent))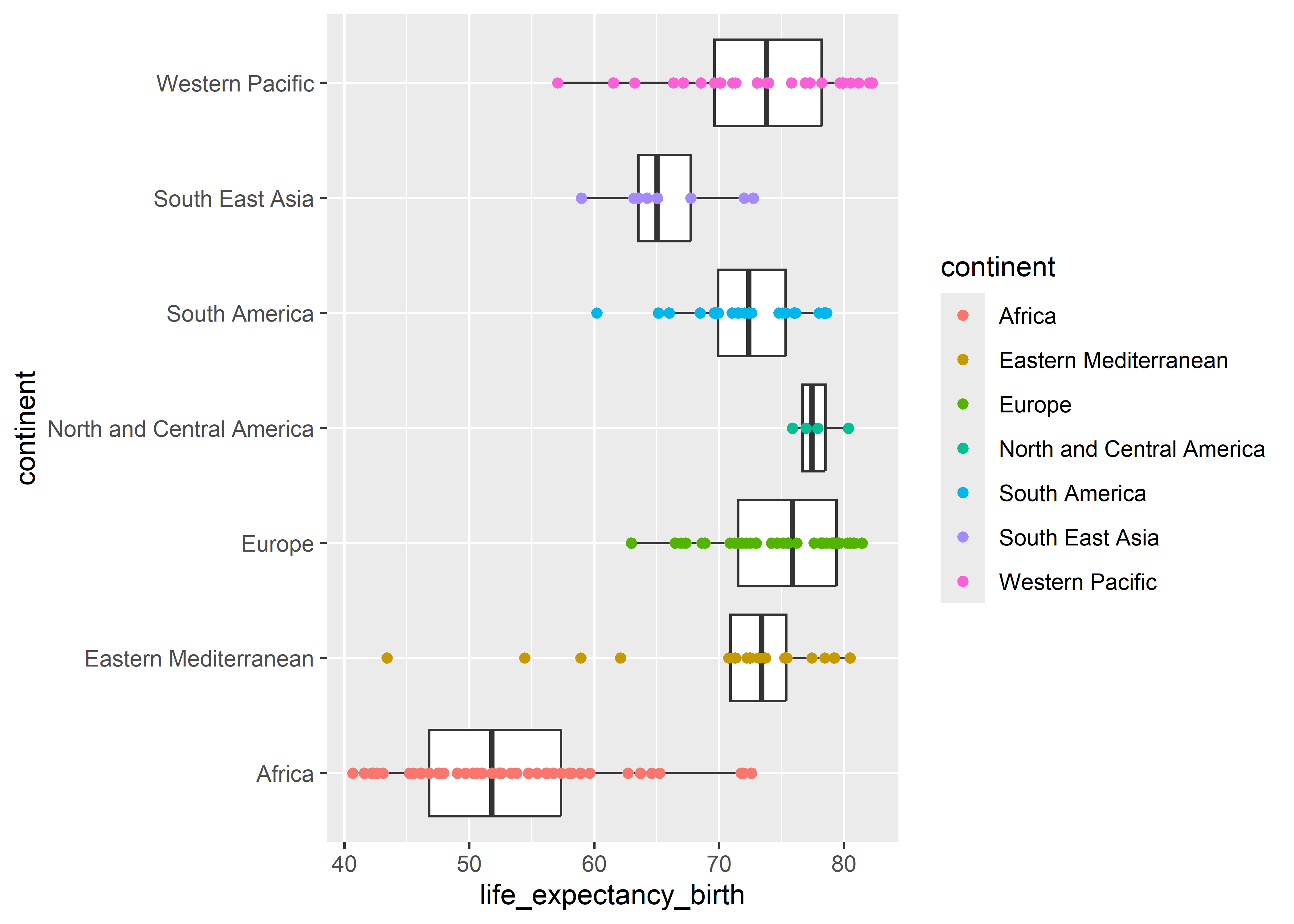
O de forma global en la función ggplot.
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent, color = continent)) +
geom_boxplot() +
geom_point()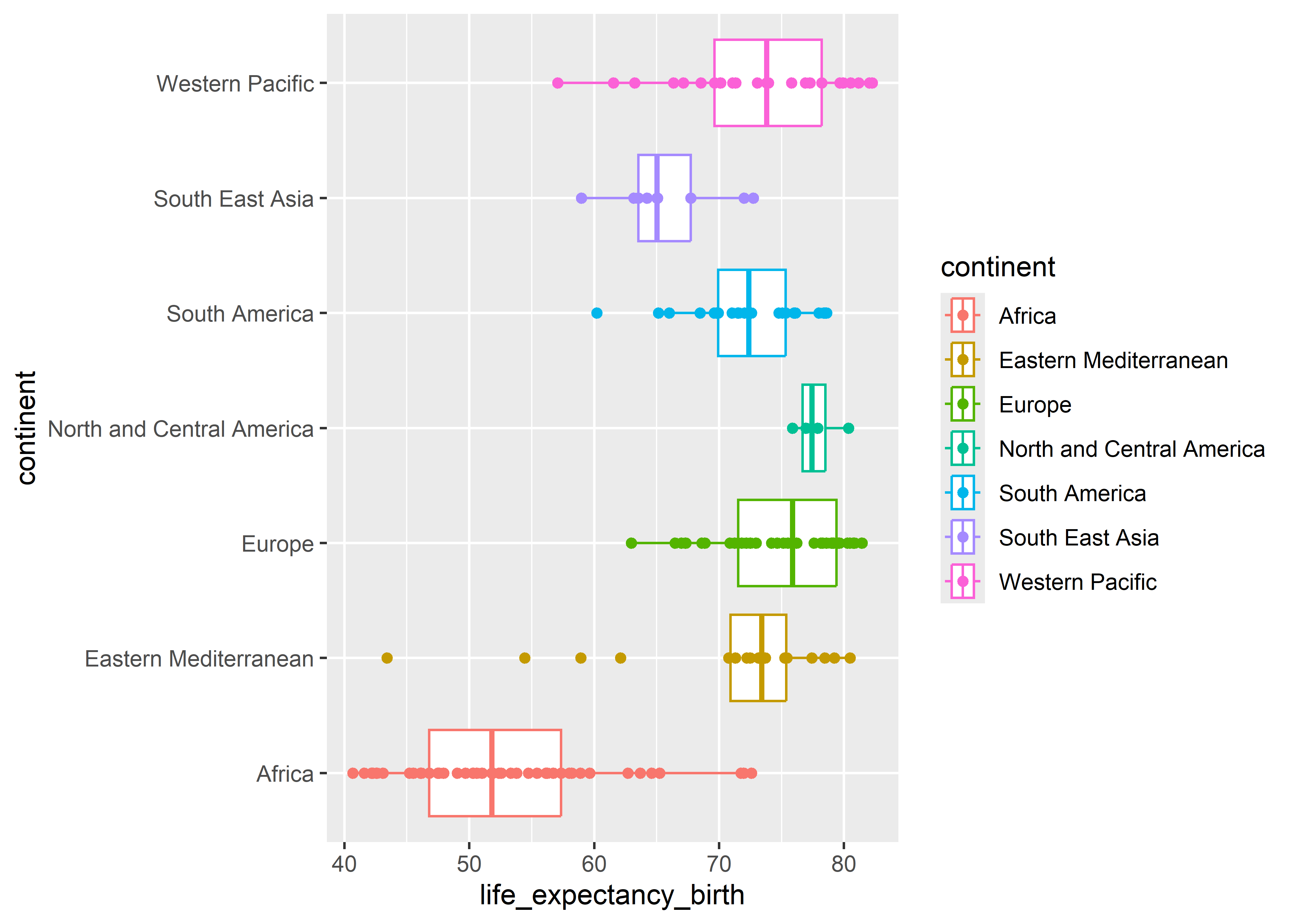
5.6 Propiedades fijas
En constraste, las propiedades son especificaciones fijas y se definen fuera de aes().
Es importante notar también que las propiedades, al ser valores que no están “mapeados”, no son incluidos en la leyenda del gráfico.
En el ejemplo anterior, podemos especificar el color de cada geometría. Se aplicará a todo la geometría por igual.
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent)) +
geom_boxplot(color = "red") +
geom_point(color = "orange")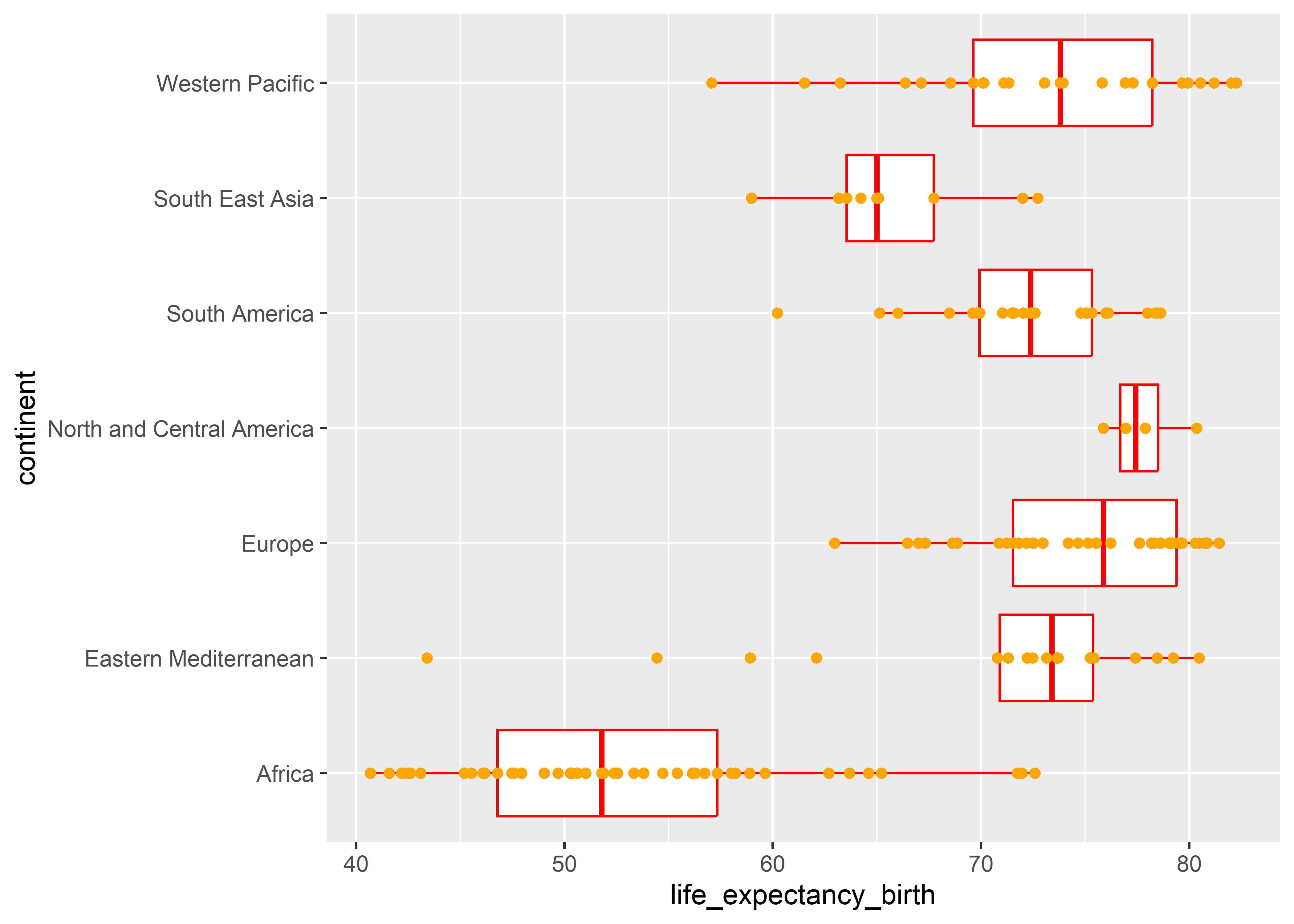
En resumen, para este libro, las diferencias entre atributos y propiedades se muestran en esta tabla:
| Atributos | Propiedades | |
|---|---|---|
| Se vinculan a: | Los datos | La geometría |
| Localización: | Dentro de la estética aes()
|
Fuera de la estética aes()
|
| Se incluyen en la leyenda: | Sí | No |
| Tipo de ajuste: | Con escalas | Manual |
5.7 Personalización
ggplot2 tiene un gran repertorio para personalizar los gráficos. A continuación, mostraremos algunos de ellos.
5.7.1 Etiquetas
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent, color = continent)) +
geom_boxplot() +
geom_point() +
labs(x = "Esperanza de vida", y = "Continente",
color = "Continente")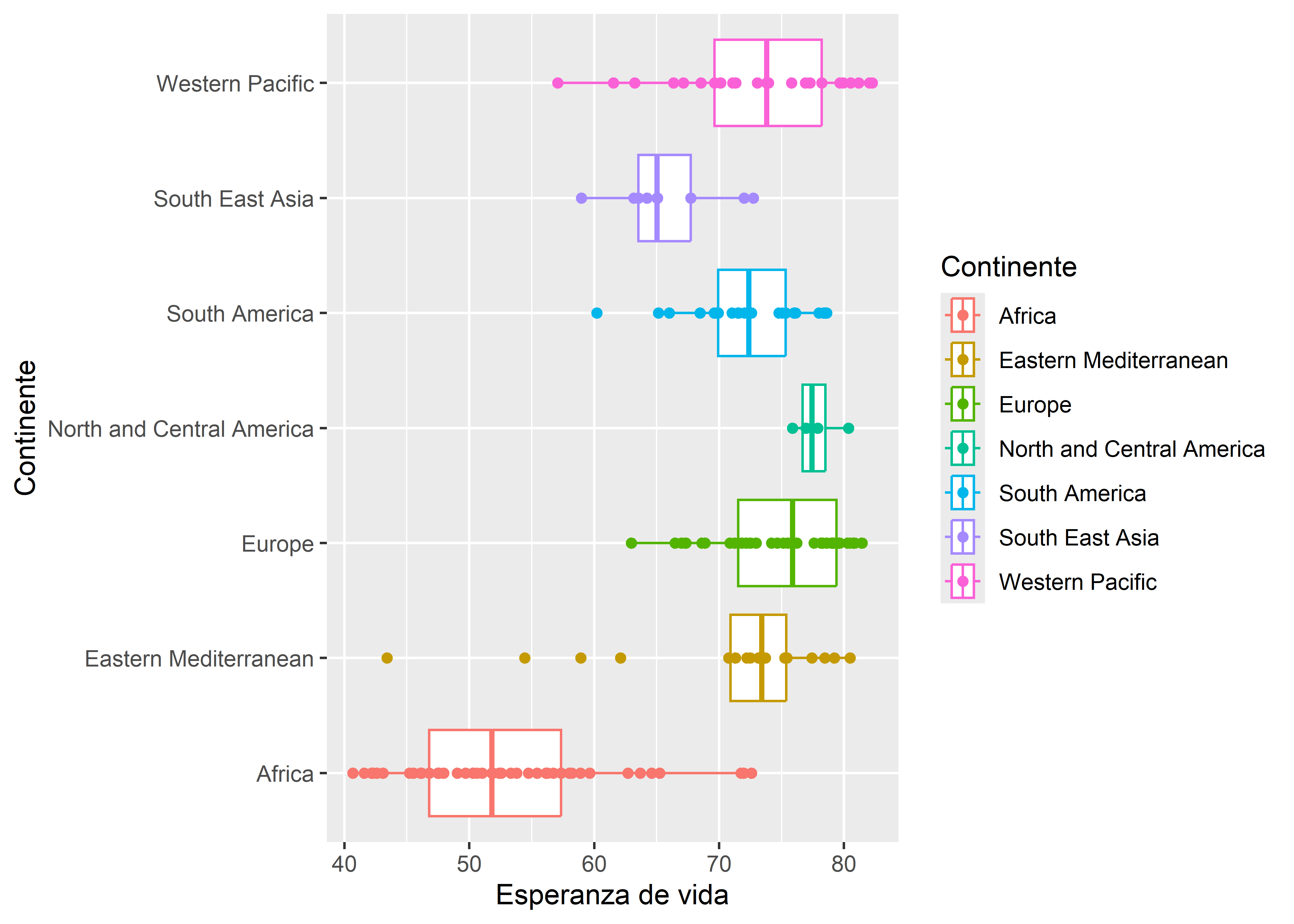
5.7.2 Temas
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent, color = continent)) +
geom_boxplot() +
geom_point() +
labs(x = "Esperanza de vida", y = "Continente",
color = "Continente") +
theme_bw()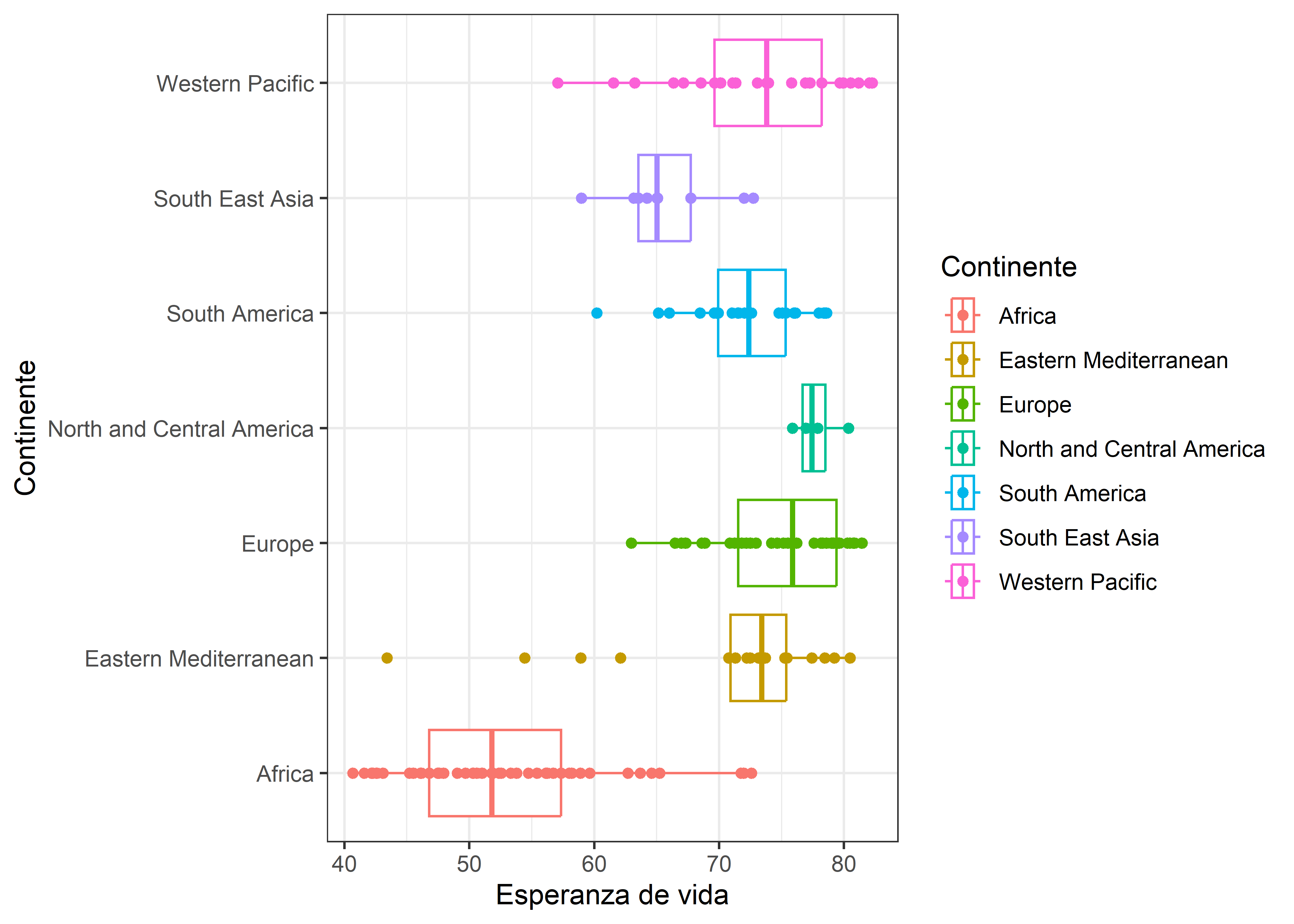
who %>%
ggplot(aes(x = life_expectancy_birth, y = continent, color = continent)) +
geom_boxplot() +
geom_point() +
labs(x = "Esperanza de vida", y = "Continente",
color = "Continente") +
theme_minimal()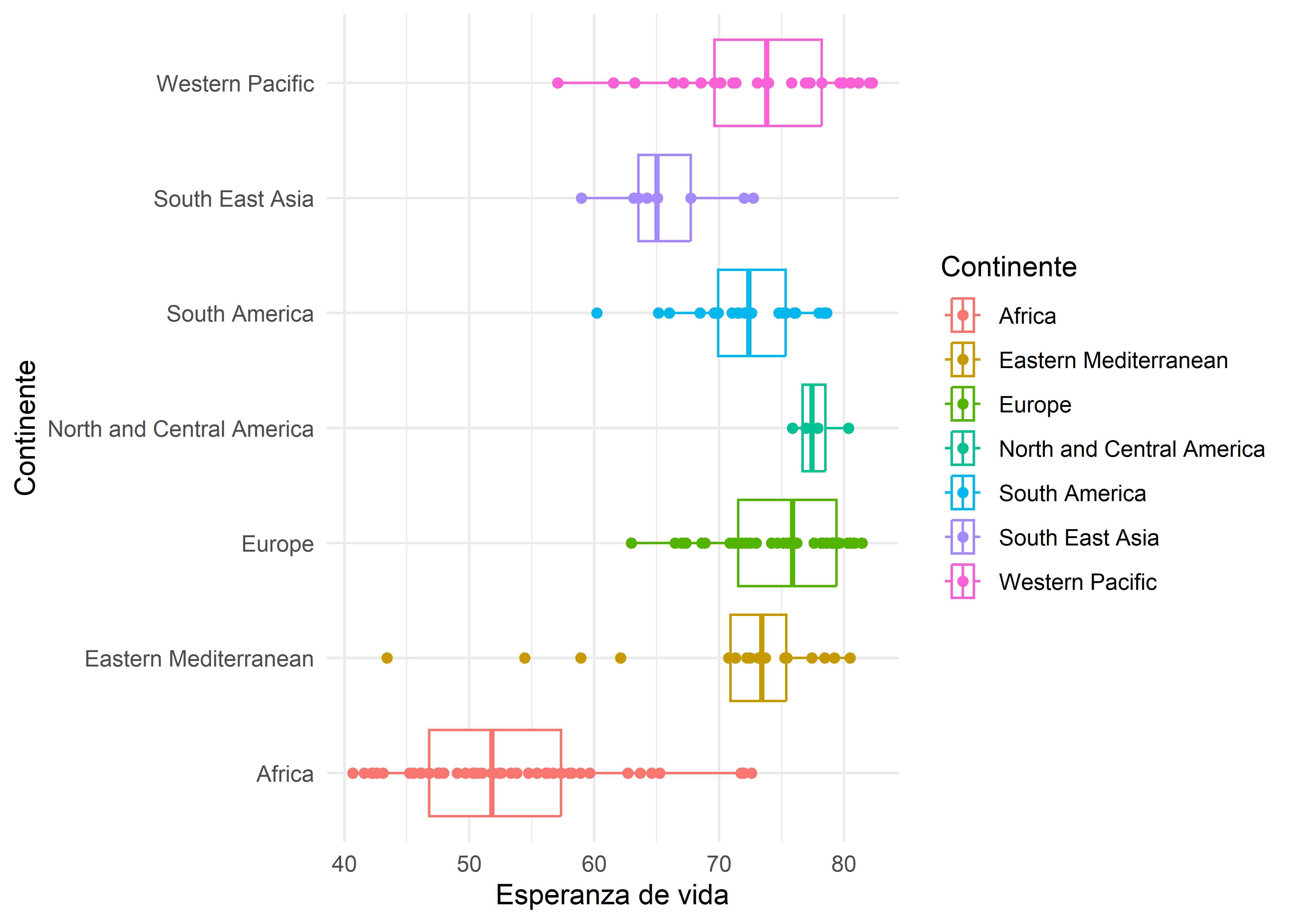
5.7.3 Escalas
Los atributos al ser “mapeados” al gráfico, pueden ser modificado mendiante el uso de escalas. Las funciones para modificar la apariencia de los atributos empiezan con scale_*.
5.7.3.1 Escala Manual
En el siguiente gráfico, además de los ejes, hemos “mapeado” en la estética 2 atributos: el color (col) y el tamaño (size).
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point()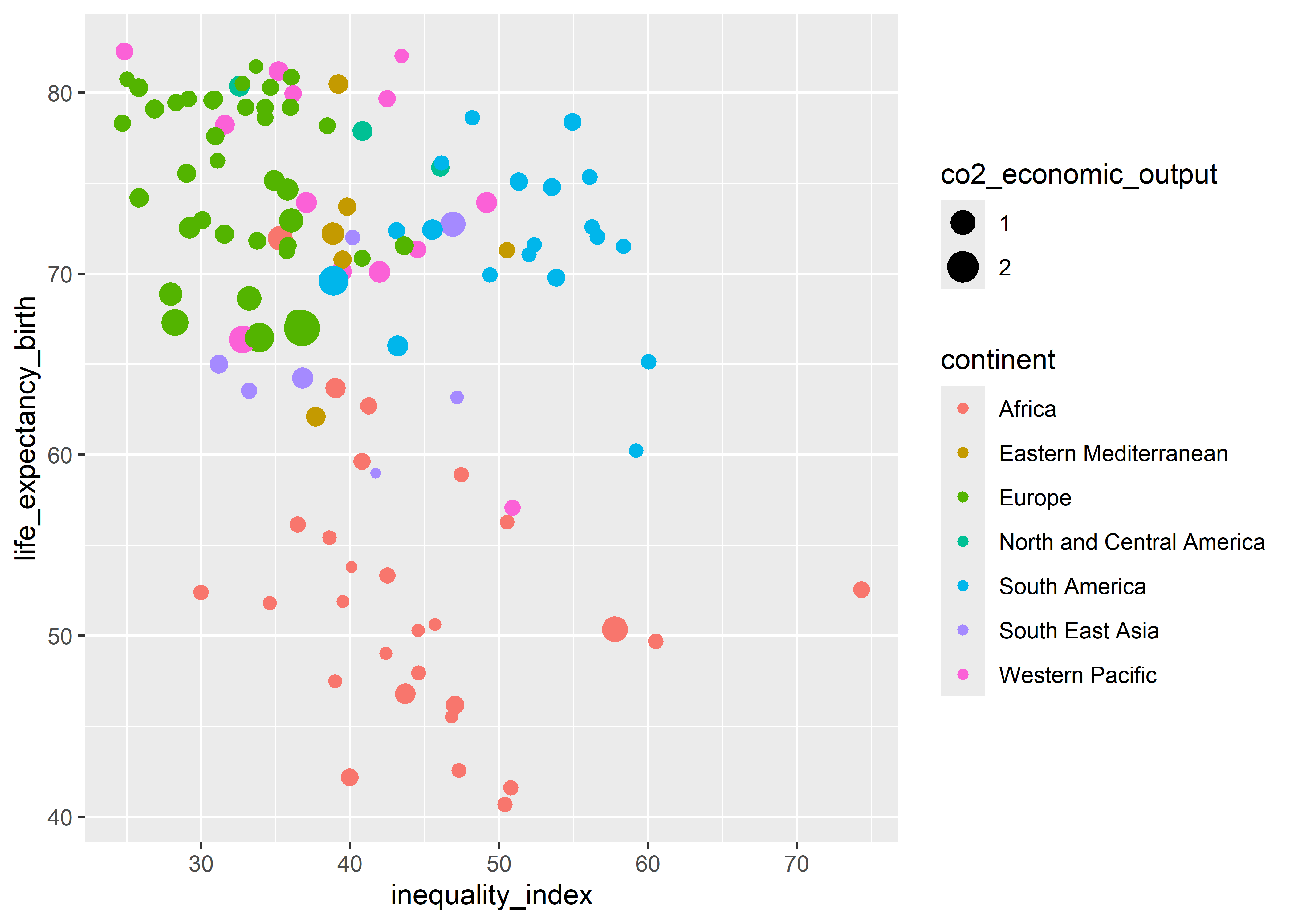
Al “mapear” estos valores, ggplot2 le asigna una escala por defecto. En el caso del color, utiliza una escala equidistante:
Empezaremos por modificar las escalas de forma manual, es decir, vamos a especificar el valor de los colores que queremos sean mapeados a nuestros datos. Para esto utilizaremos la función scale_color_manual y pasaremos el “nombre” de los colores al argumento values.
Es importante notar que el orden de ingreso de los valores manuales tiene que estar en el mismo orden de las categorias que son mapeadas.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point() +
scale_color_manual(values=c("red","blue","green","pink","yellow","purple","skyblue"))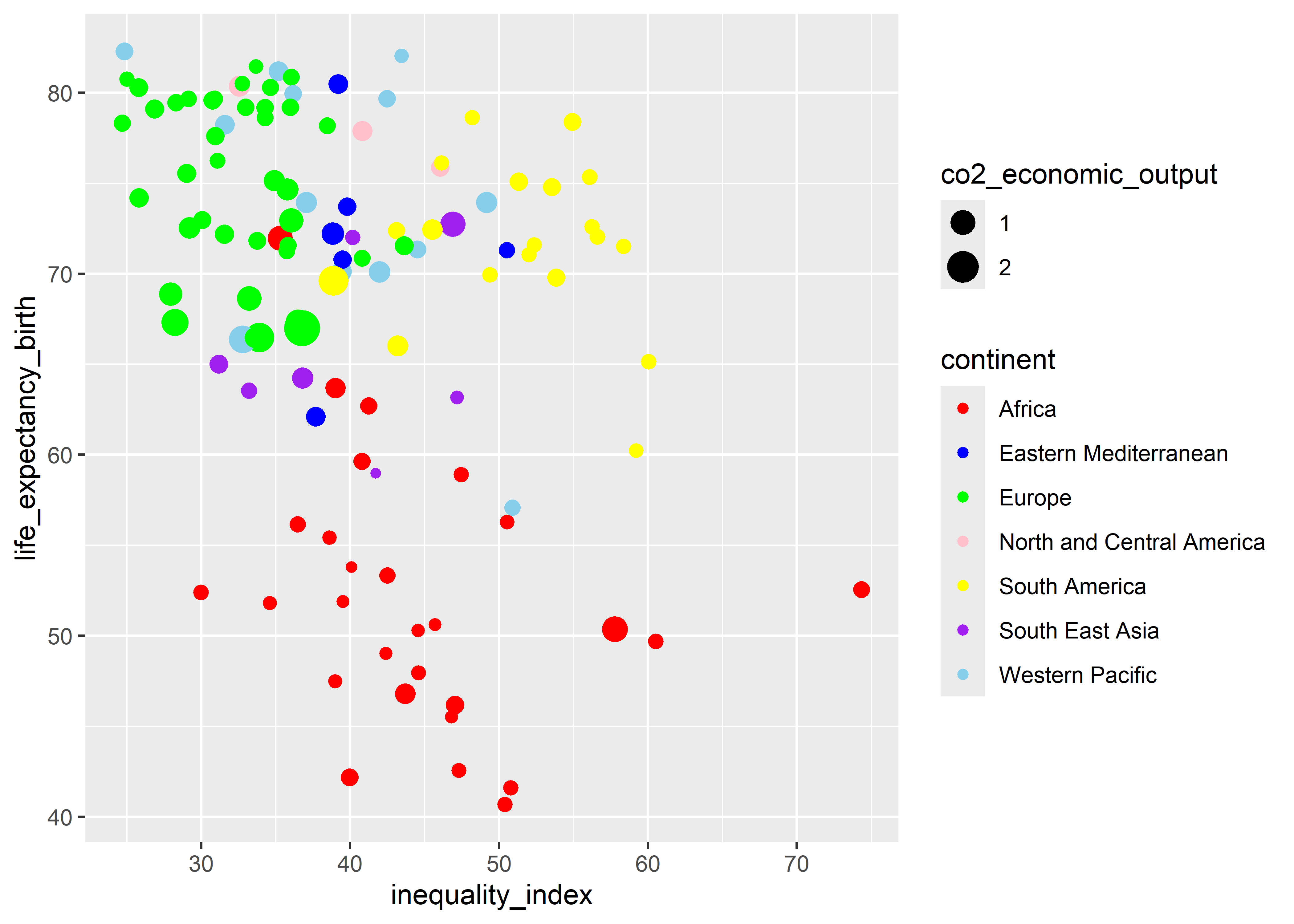
ggplot2 tambien puede usar codificación de color como por ejemplo HEX. El paquete colourpicker permite seleccionar de forma sencilla los colores en formato HEX.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point() +
scale_color_manual(values=c("#4bb9c3","#88d78c", "#d3d483", "#2b8282", "#9ad581", "#b4d3d4", "#ecd9bc"))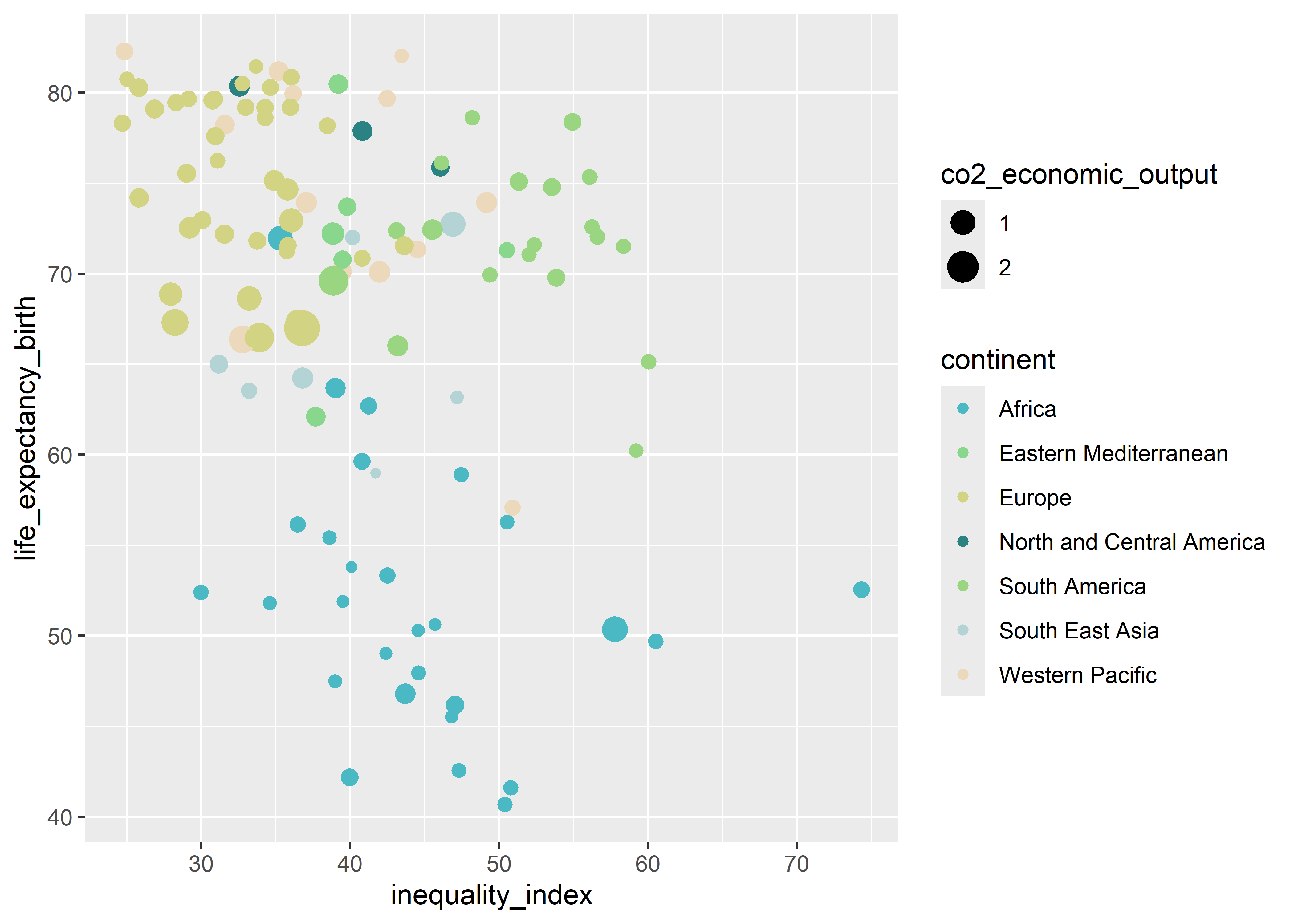
El conjunto de colores se conoce como paleta. Lo veremos con más detalle en la sección de escalas predefinidas. Un buen recurso para encontrar paletas curadas por especialistas o entusiastas es ColorHunt.
De la misma forma que el color, el atributo tamaño (size) del gráfico puede ser controlado con la escala scale_size_continuous.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point() +
scale_color_manual(values=c("#4bb9c3","#88d78c", "#d3d483", "#2b8282", "#9ad581", "#b4d3d4", "#ecd9bc")) +
scale_size_continuous(breaks = c(.1,1,5,10))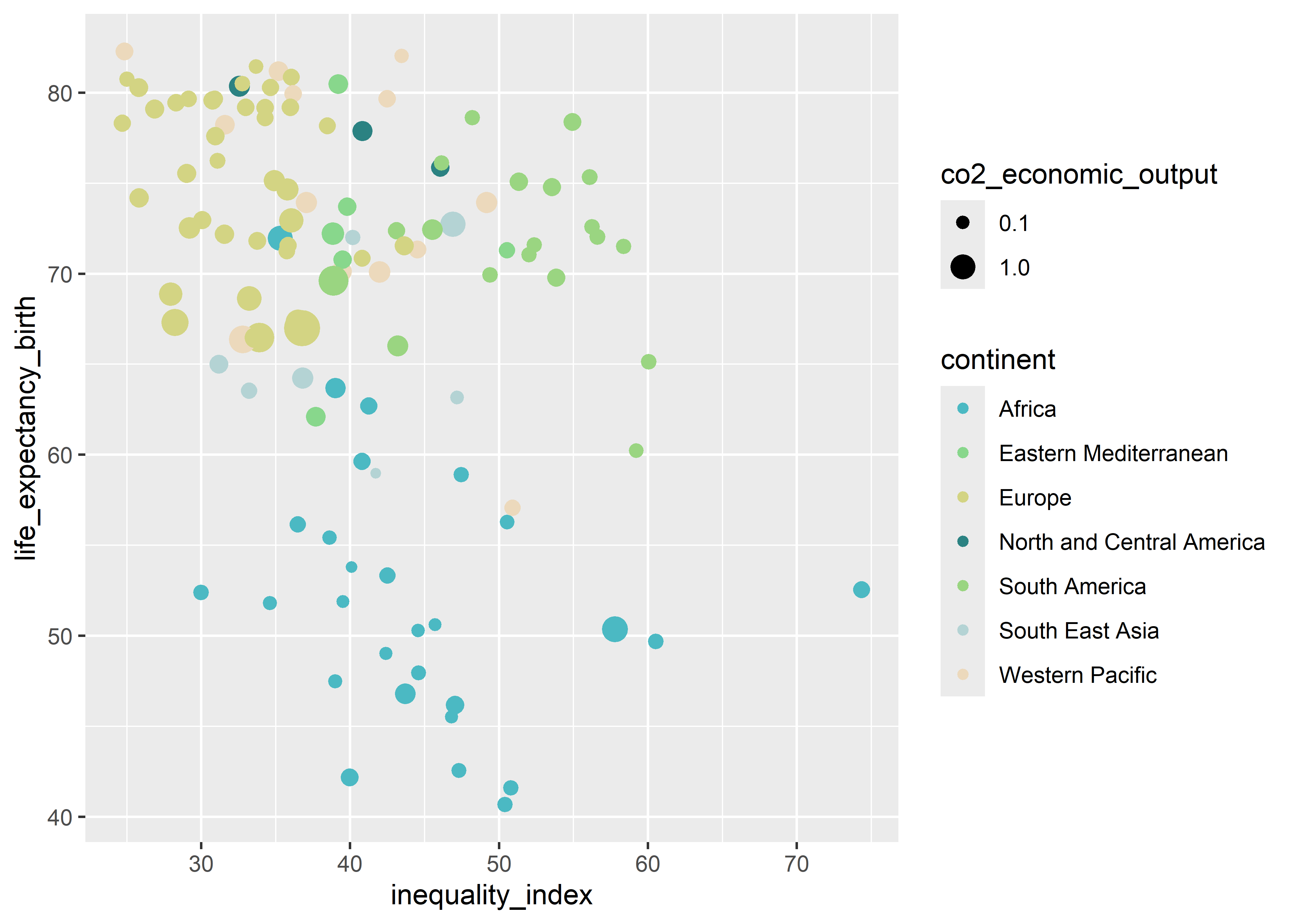
Dado que los puntos se sobreponen, agregaremos un atributo de transparencia (alpha).
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output, alpha = co2_economic_output)) +
geom_point() +
scale_color_manual(values=c("#4bb9c3","#88d78c", "#d3d483", "#2b8282", "#9ad581", "#b4d3d4", "#ecd9bc")) +
scale_size_continuous(breaks = c(.1,1,5,10))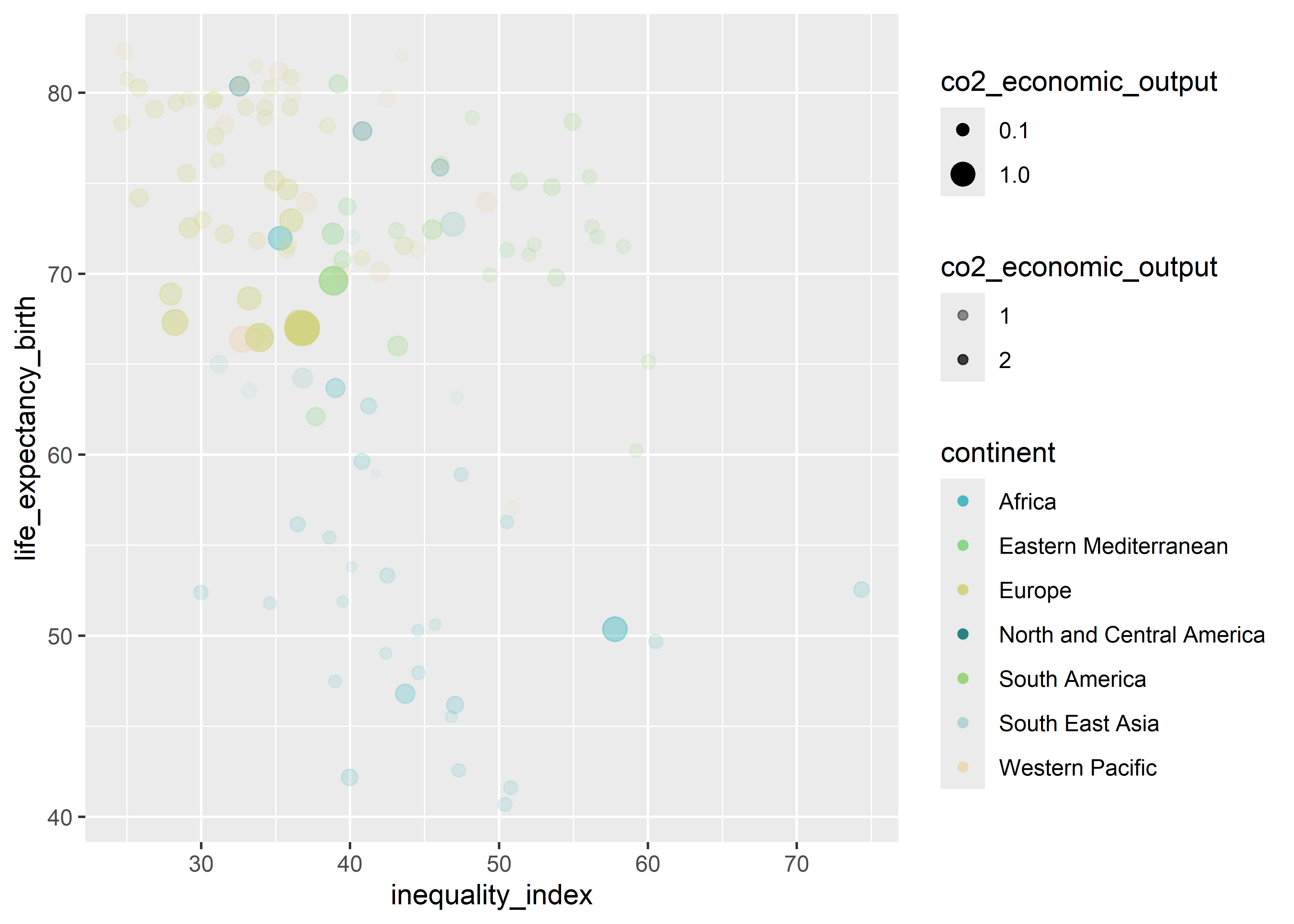
Y lo controlaremos usando la escala scale_alpha_continuous
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output, alpha = co2_economic_output)) +
geom_point() +
scale_color_manual(values=c("#4bb9c3","#88d78c", "#d3d483", "#2b8282", "#9ad581", "#b4d3d4", "#ecd9bc")) +
scale_size_continuous(breaks = c(.1,1,5,10)) +
scale_alpha_continuous(breaks = c(.1,1,5,10))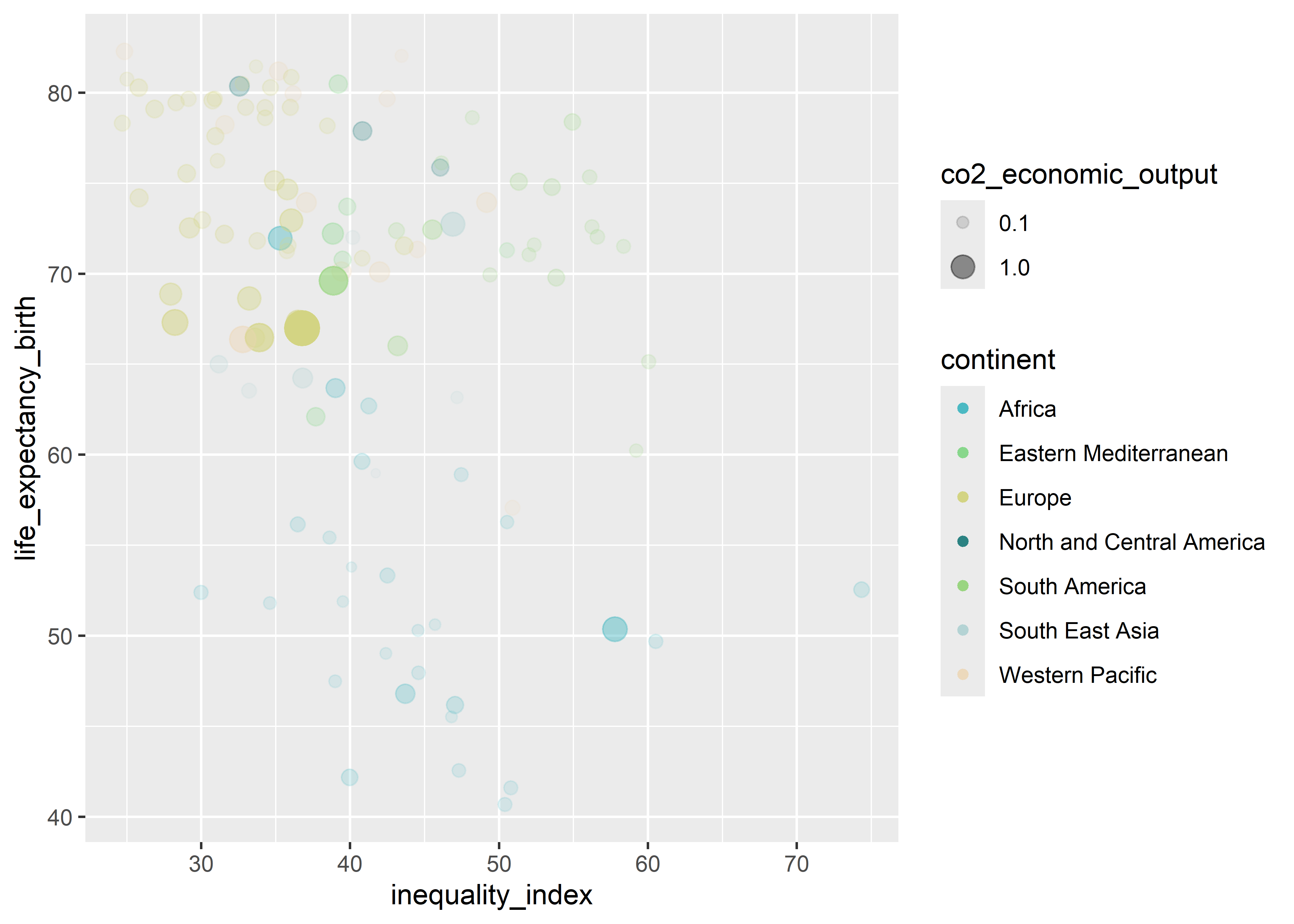
5.7.3.2 Escala Predefinida
RColorBrewer es un paquete que contiene diferentes paletas predefinidas para gráficos en R y viene implemantado de forma nativa en ggplot2.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point() +
labs(x = "Indice de Inequidad", y = "Esperanza de vida",
color = "Continente", size = "Emision de CO2") +
theme_minimal() +
scale_color_brewer(palette = "Blues")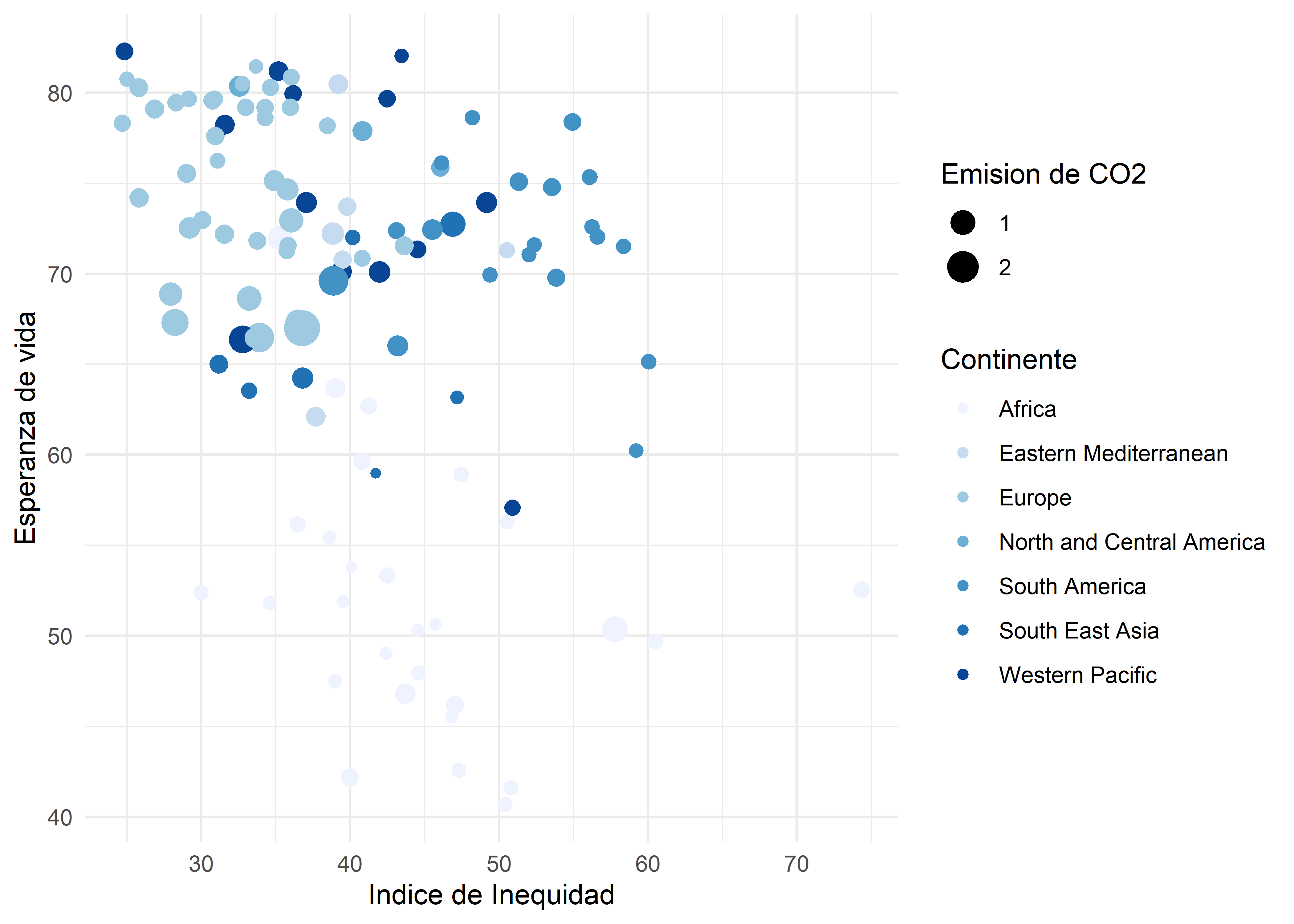
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point() +
labs(x = "Indice de Inequidad", y = "Esperanza de vida",
color = "Continente", size = "Emision de CO2") +
theme_minimal() +
scale_color_brewer(palette = "Spectral")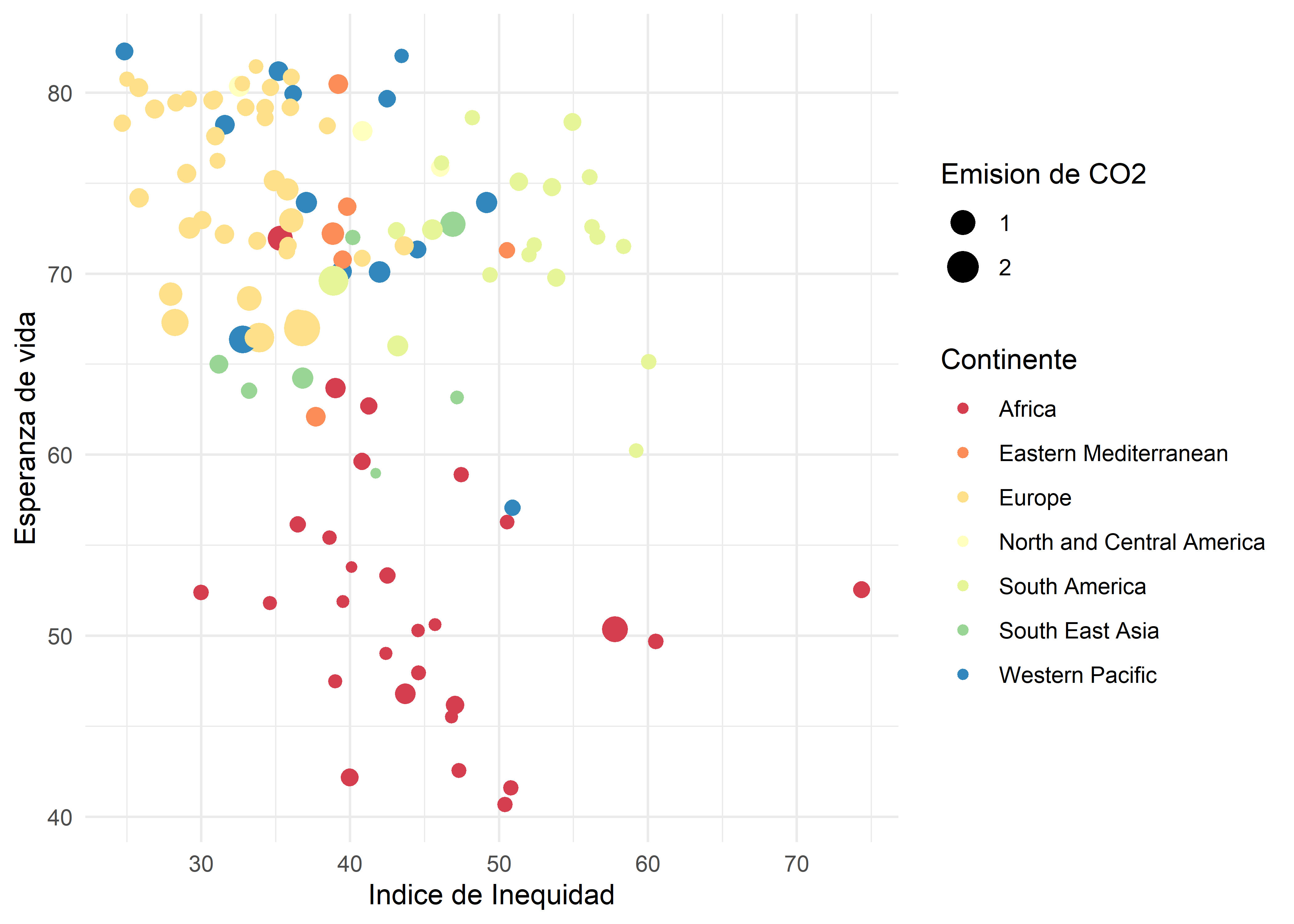
Existen algunos paquetes que tienen un repertorio de paletas más amplio como ggsci, viridis y colorspace.
library(ggsci)
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent, size = co2_economic_output)) +
geom_point() +
labs(x = "Indice de Inequidad", y = "Esperanza de vida",
color = "Continente", size = "Emision de CO2") +
theme_minimal() +
scale_color_lancet()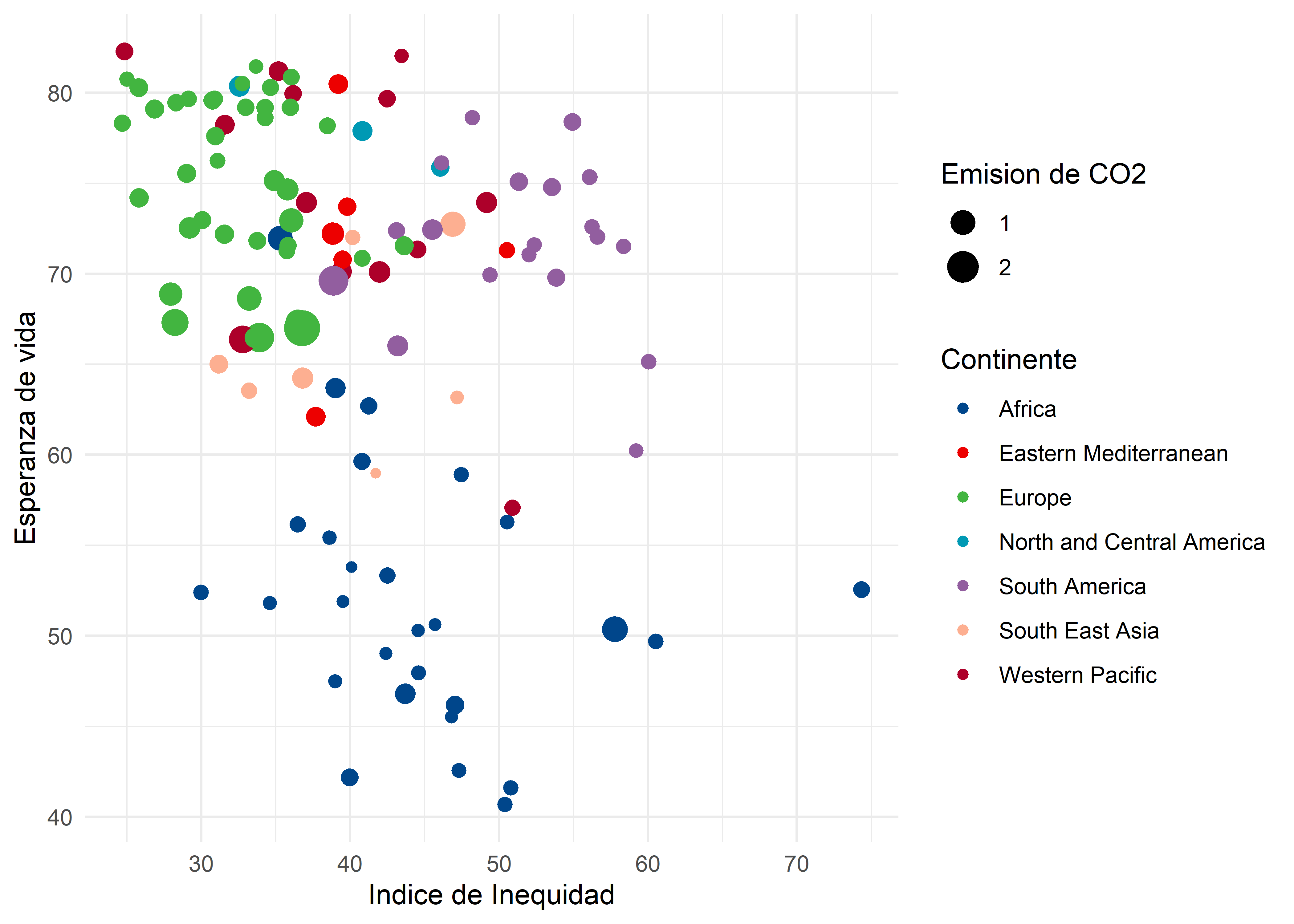
5.7.4 Facetas
Por razones estéticas o de análisis/exploración, es conveniente estratificar (o separar) los datos de acuerdo a categorías de interés. Hemos observado cómo podemos “mapear” estas variables utilizando atributos, sin embargo en algunos casos es importante generar paneles independientes para cada categoría. Podemos asignar un gráfico en un espacio separado para cada categoría de una variable utilizando las facetas con los argumentos facet_grid o facet_wrap.
facet_gridgenera un arreglo de gráficos en columnas y filas, mientras quefacet_wrappermite un ordenamiento más flexible. En ambos casos usaremos la expresión (~) para especificar la variable que queremos utilizar como faceta en el formatofilas~columnas.
5.7.4.1 Facetas en grillas
Empezaremos con nuestro gráfico donde mapeamos la relación entre el índice de inequidad y la esperanza de vida al nacer con un color por cada continente (3 artibutos):
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
scale_color_npg()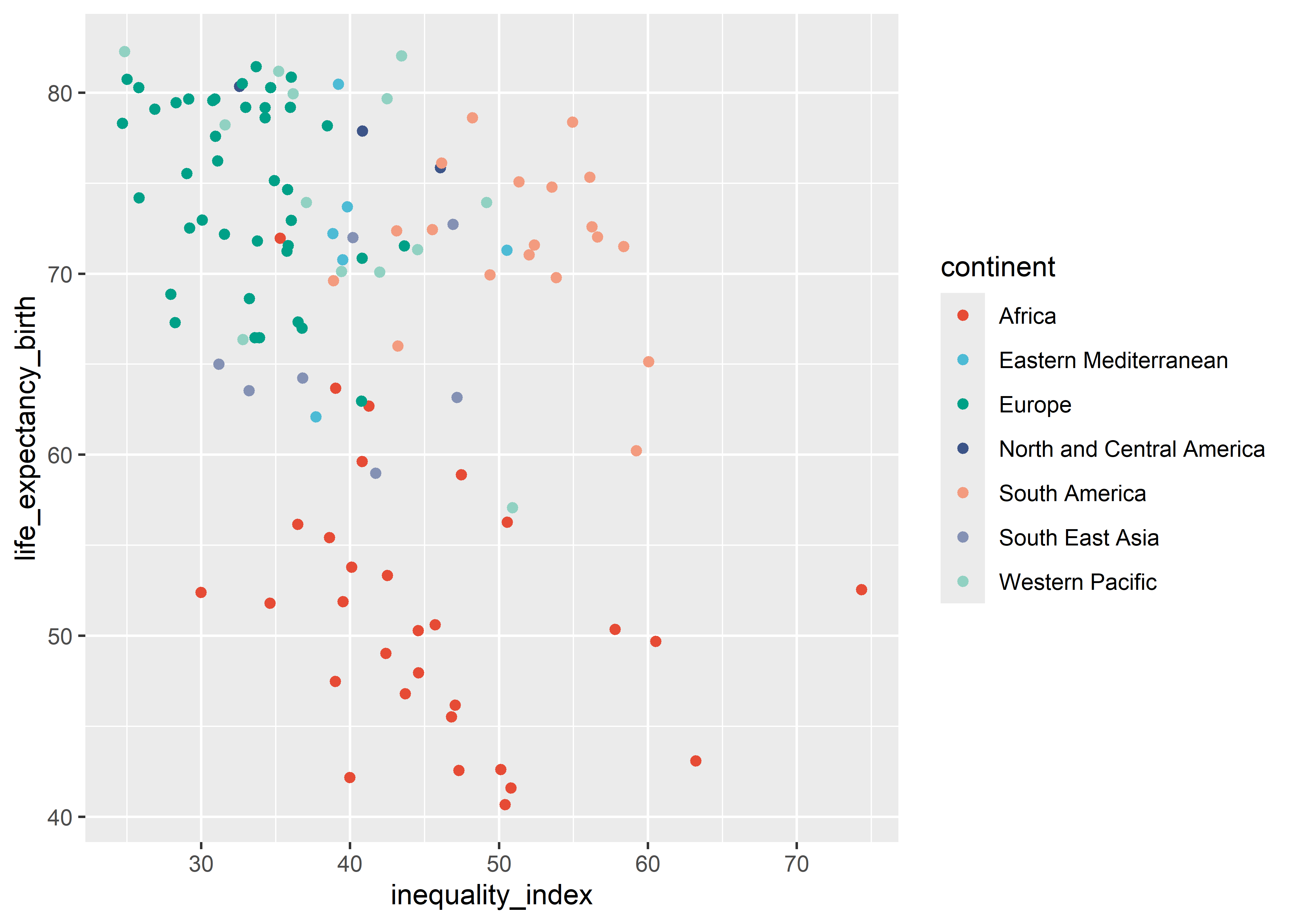
A continuación, separaremos en paneles de acuerdo a cada continente en diferentes filas usando la función facet_grid().
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
scale_color_npg() +
facet_grid(continent ~ .)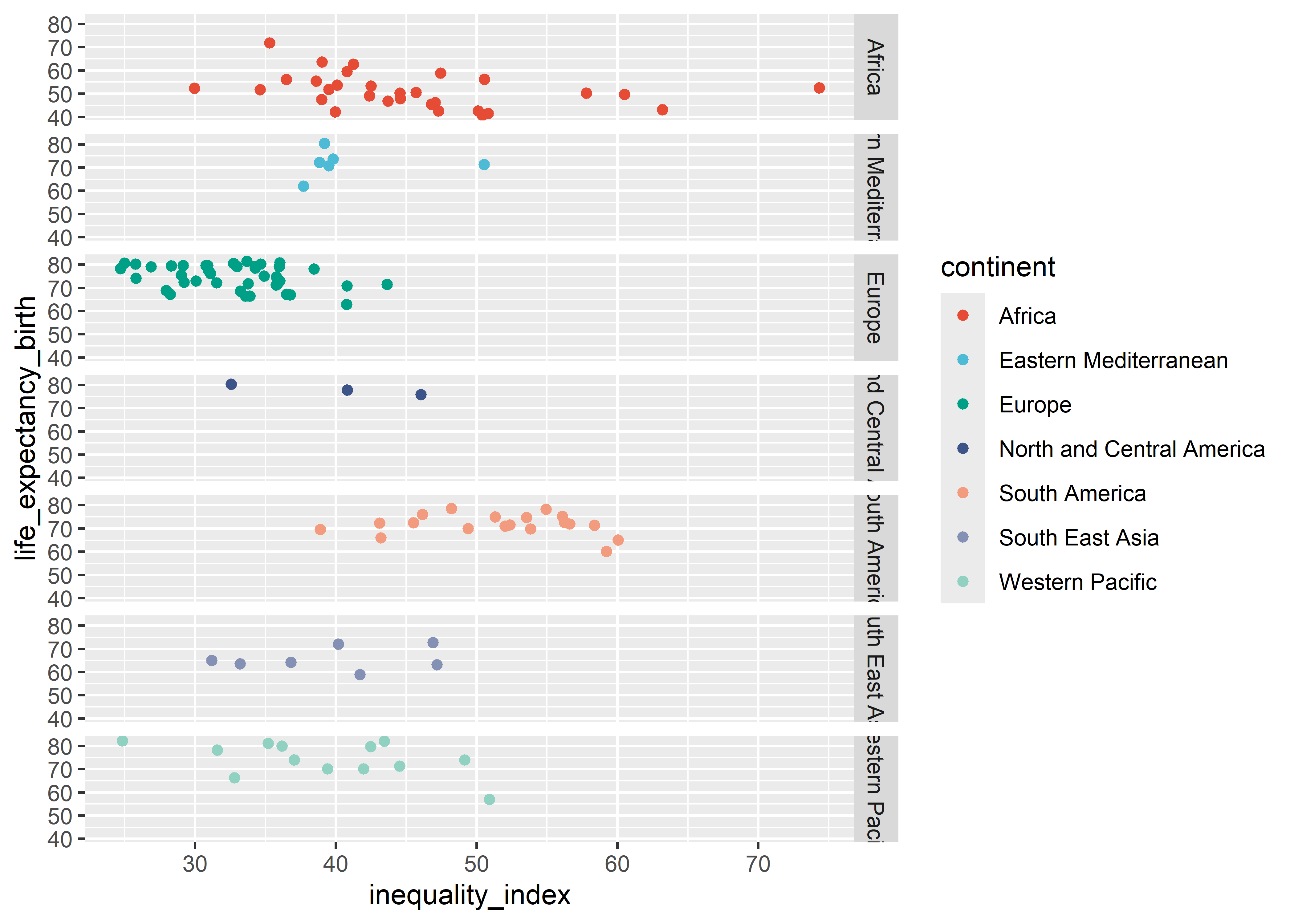
Alternativamente, podemos especificar que las facetas (o paneles) tengan un ordenamiento por columnas.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
scale_color_npg() +
facet_grid(. ~ continent)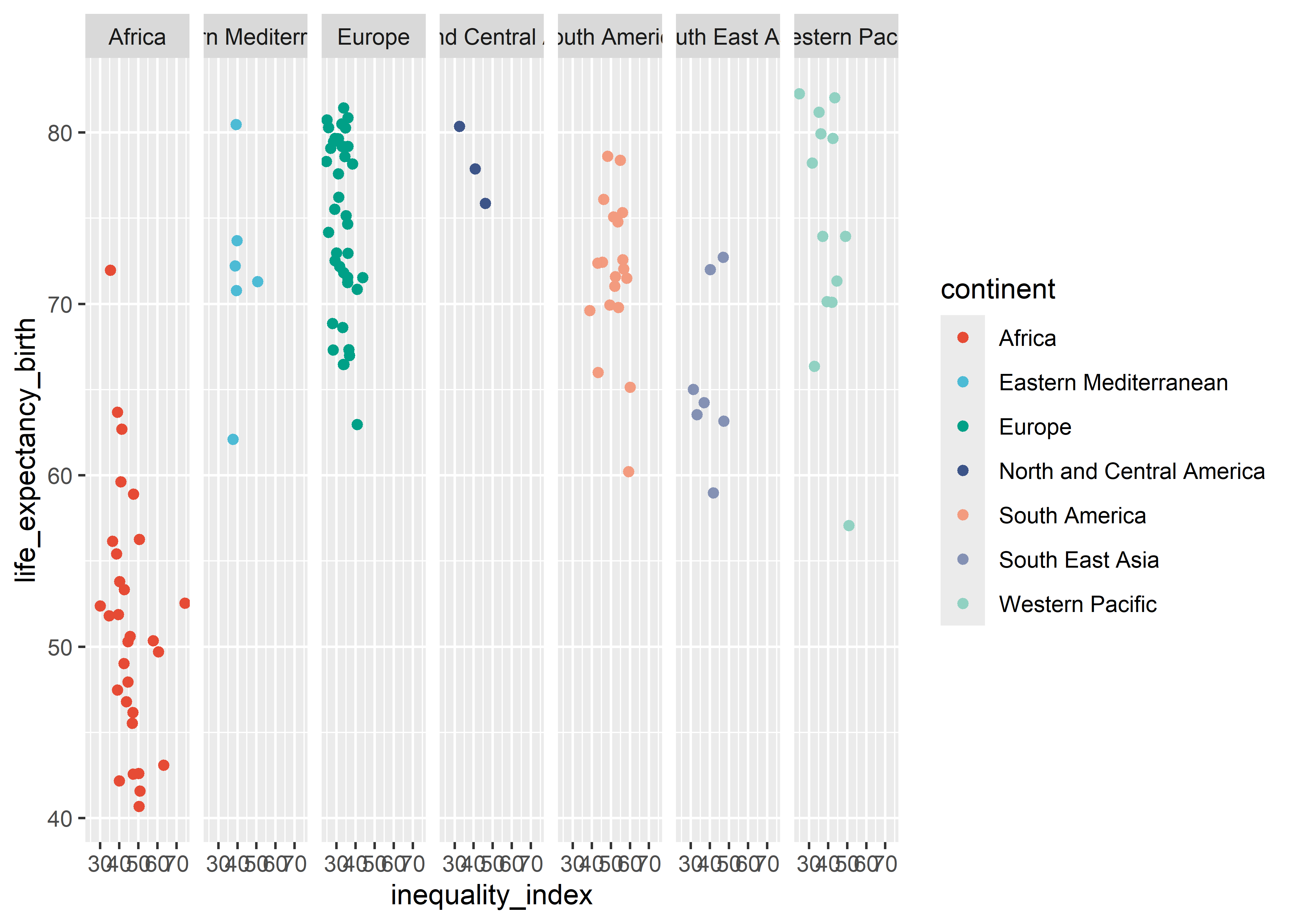
Por último, podemos agregar más de una variable de agrupamiento.
who %>%
mutate(low_middle_income = ifelse(gni_per_capita/1000 < 4.465, "Yes", "No")) %>%
drop_na(low_middle_income) %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
scale_color_npg() +
facet_grid(low_middle_income ~ continent)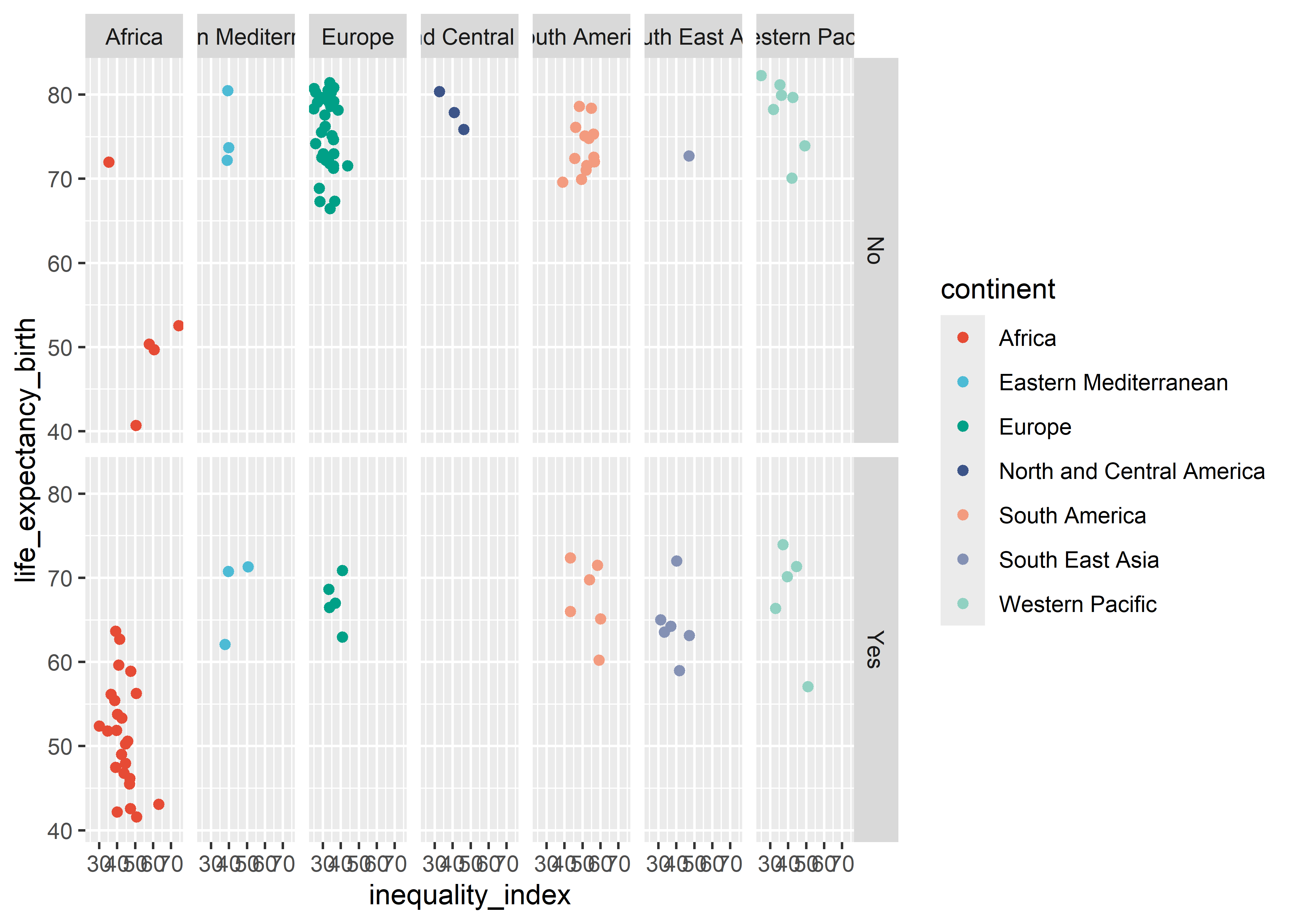
5.7.4.2 Facetas agrupadas
Los mismos principios sirven para gráficos más elaborados o con más variables. Ahora utilizaremos facet_wrap.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
scale_color_npg() +
facet_wrap(. ~ continent)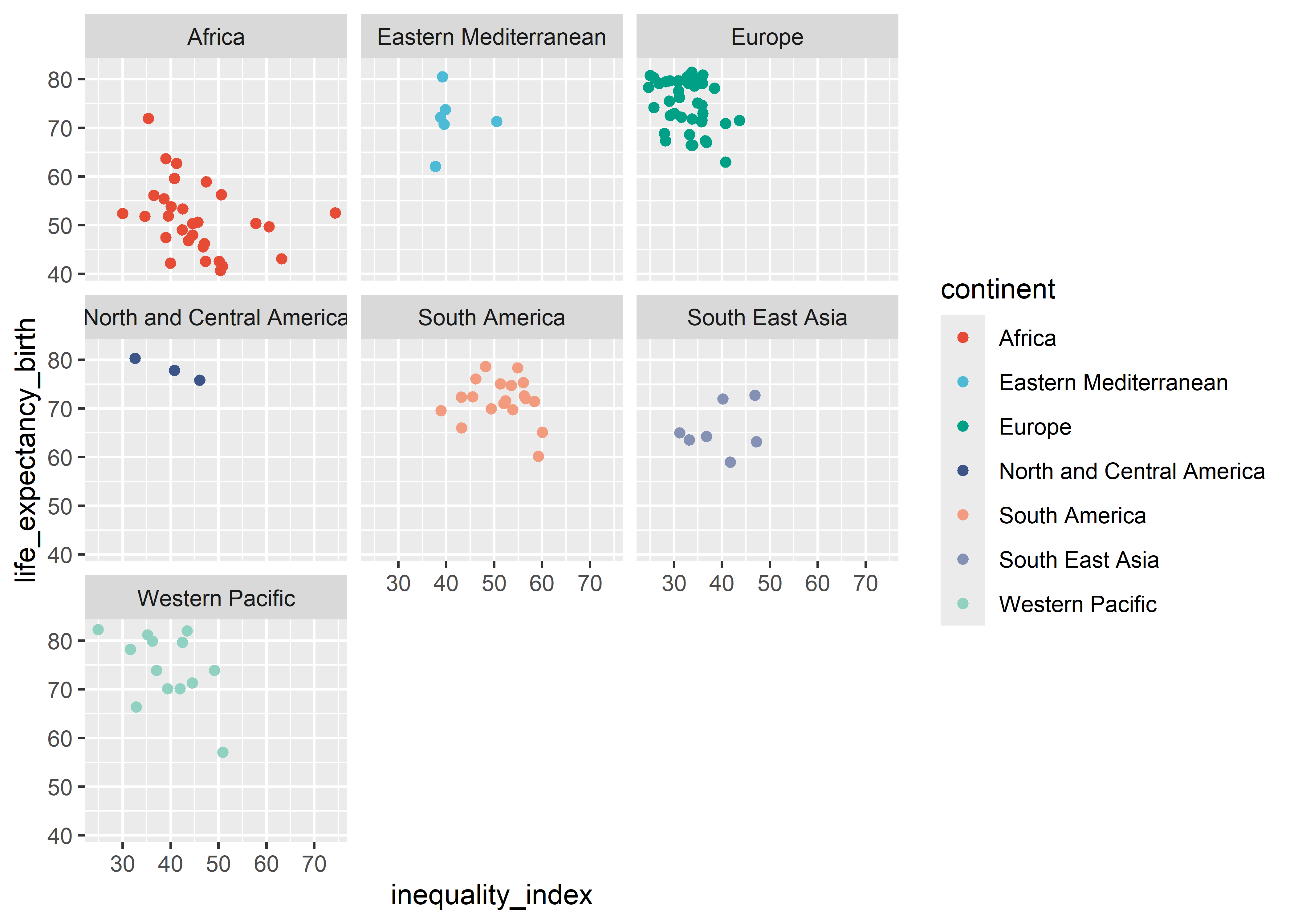
Dado que
facet_wrapno organiza las facetas de acuerdo a una variable, cada panel puede ser ordenado independientemente, y en consecuencia uno puede cambiar el número de columnas o filas con los argumentosncolonrow, respectivamente.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
scale_color_npg() +
facet_wrap(. ~ continent, ncol = 2)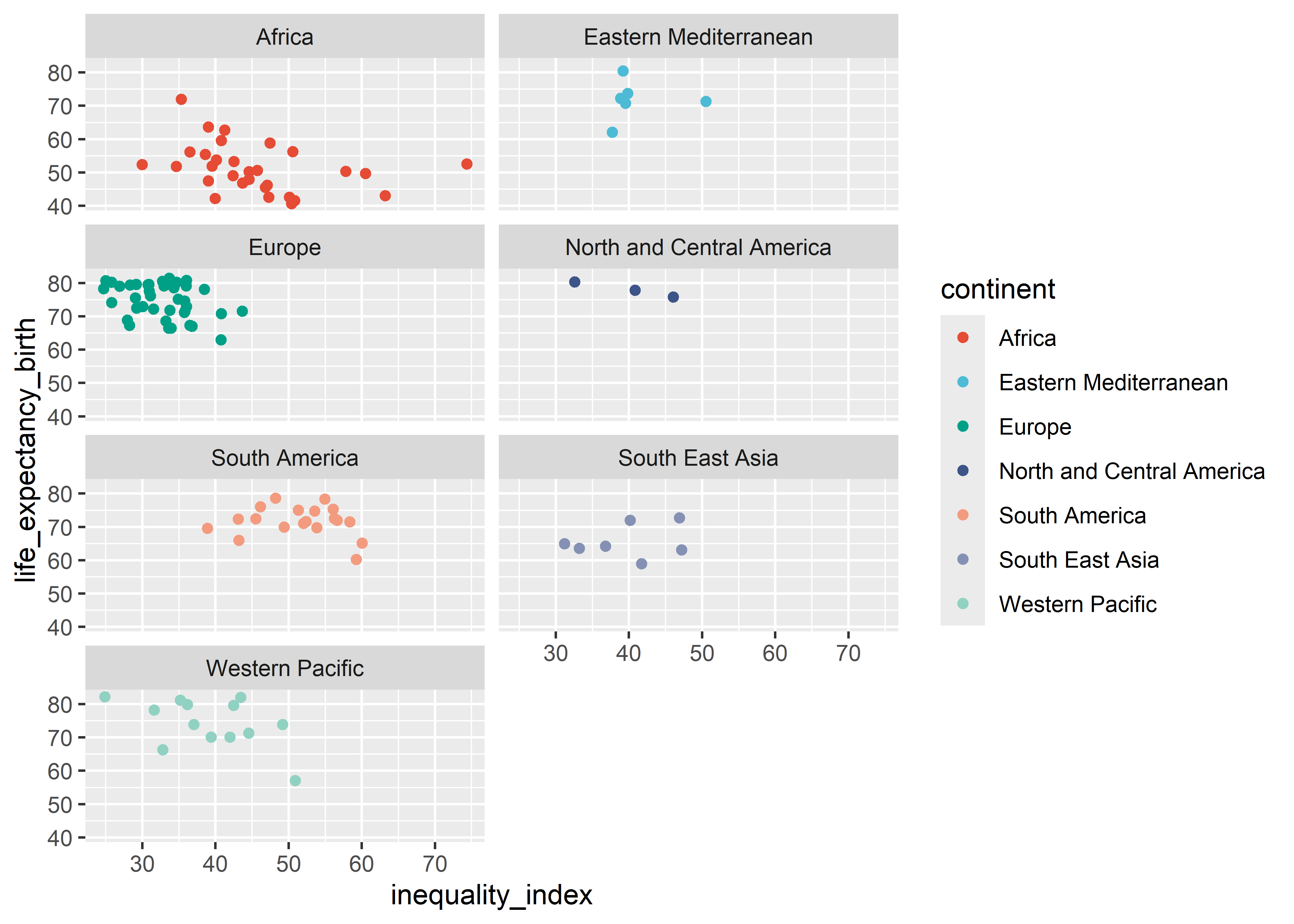
En la organización por facet_wrap, es frecuente que cada sub-panel (o categoria de la variable) tengan una escala de valores diferente. Por defecto, facet_wrap utiliza una escala fija lo cual es recomendable para poder comparar entre sub-paneles. Sin embargo, si se quiere modificar este comportamiento se puede flexibilizar las escalas en cada panel utilizando el argumento scales. Para flexibilizar el eje x se utiliza “free_x”, para el eje y se utiliza “free_y” y para ambos ejes “free”.
Flexibilizar los ejes nos permite explorar en más detalle cada sub-grupo, sin embargo dificulta la comparación entre sub-grupos.
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
scale_color_npg() +
facet_wrap(. ~ continent, ncol = 2, scales = "free")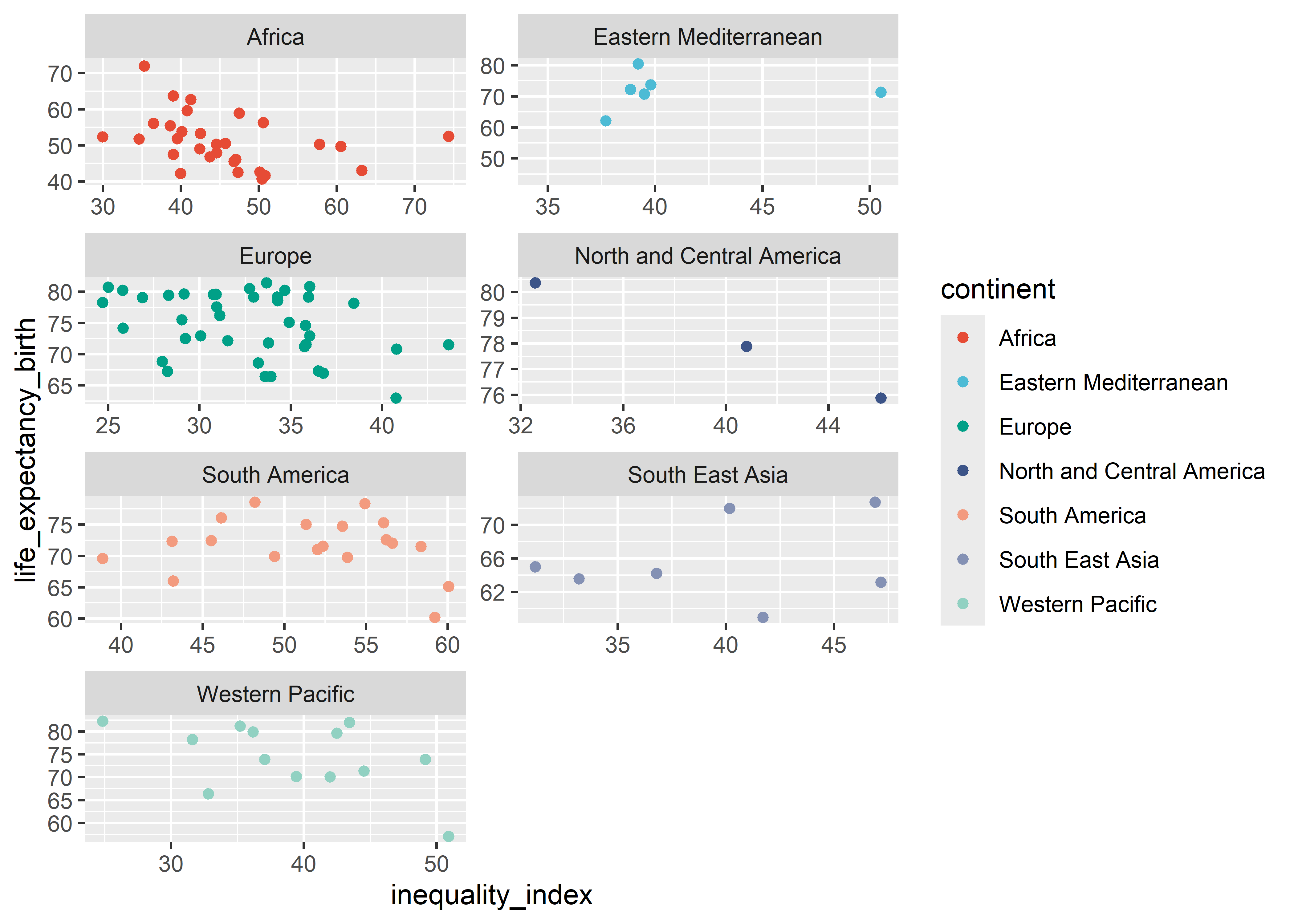
Con esta visualización se puede distinguir mejor las tendencias en cada uno de las facetas (paneles/grupos).
who %>%
ggplot(aes(x = inequality_index, y = life_expectancy_birth,
col = continent)) +
geom_point() +
geom_smooth(aes(fill = continent), method = "lm") +
scale_color_npg() +
scale_fill_npg() +
facet_wrap(. ~ continent, ncol = 2, scales = "free")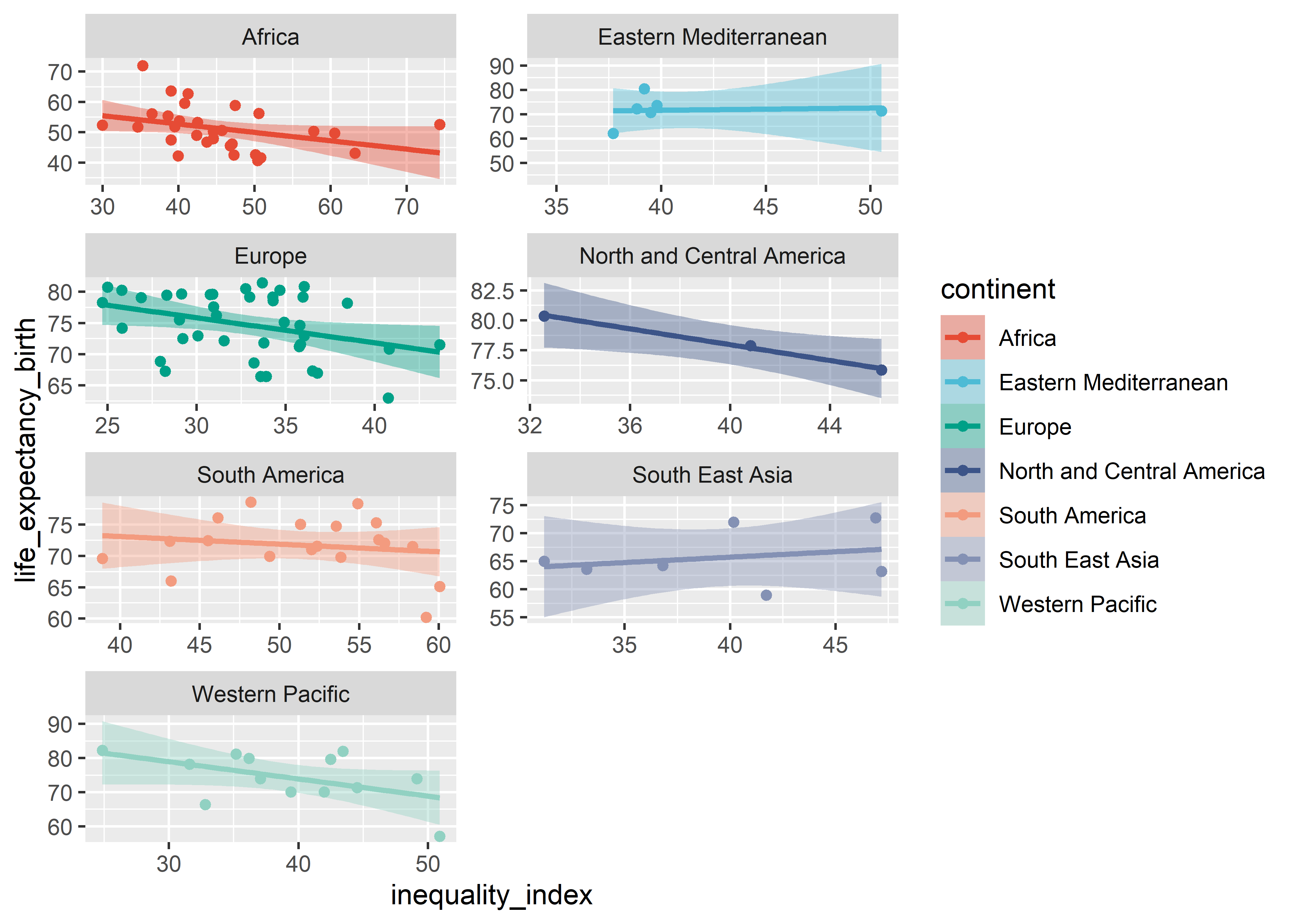
Si necesitas inspiración para realizar un gráfico, puedes visitar el portal dataviz-inspiration.com
5.8 Exportación de imágenes
Para poder guardar y exportar su ggplot se puede seguir dos vías:
- Manualmente
En el panel de “Salidas”, ir a Plot > Exportar > Guardar como imagen/Guardar como pdf e indicar dónde se guardará el archivo.
- Codificando
Para guardar y exportar por medio de un código, se deberá usar la función ggsave() e indicar la ruta, luego el objeto de donde se extraerá la imagen, y opcionalmente se podrá añadir algunas características como se muestra en el ejemplo.
ggsave("figures/map4.png", plot3, height = 8, width = 7)5.9 Ejercicios
Ejercicio 1
A) Usando el conjunto de datos who explorar la relación entre la intensidad de emisiones de CO2 debido a actividades económicas (co2_economic_output) y malnutrición infantil (malnutrition_weight_age). Diferenciar los países de acuerdo al continente (continent) donde se encuentran localizados
B) ¿Qué conclusiones podría sacar de este gráfico?
Ejercicio 2
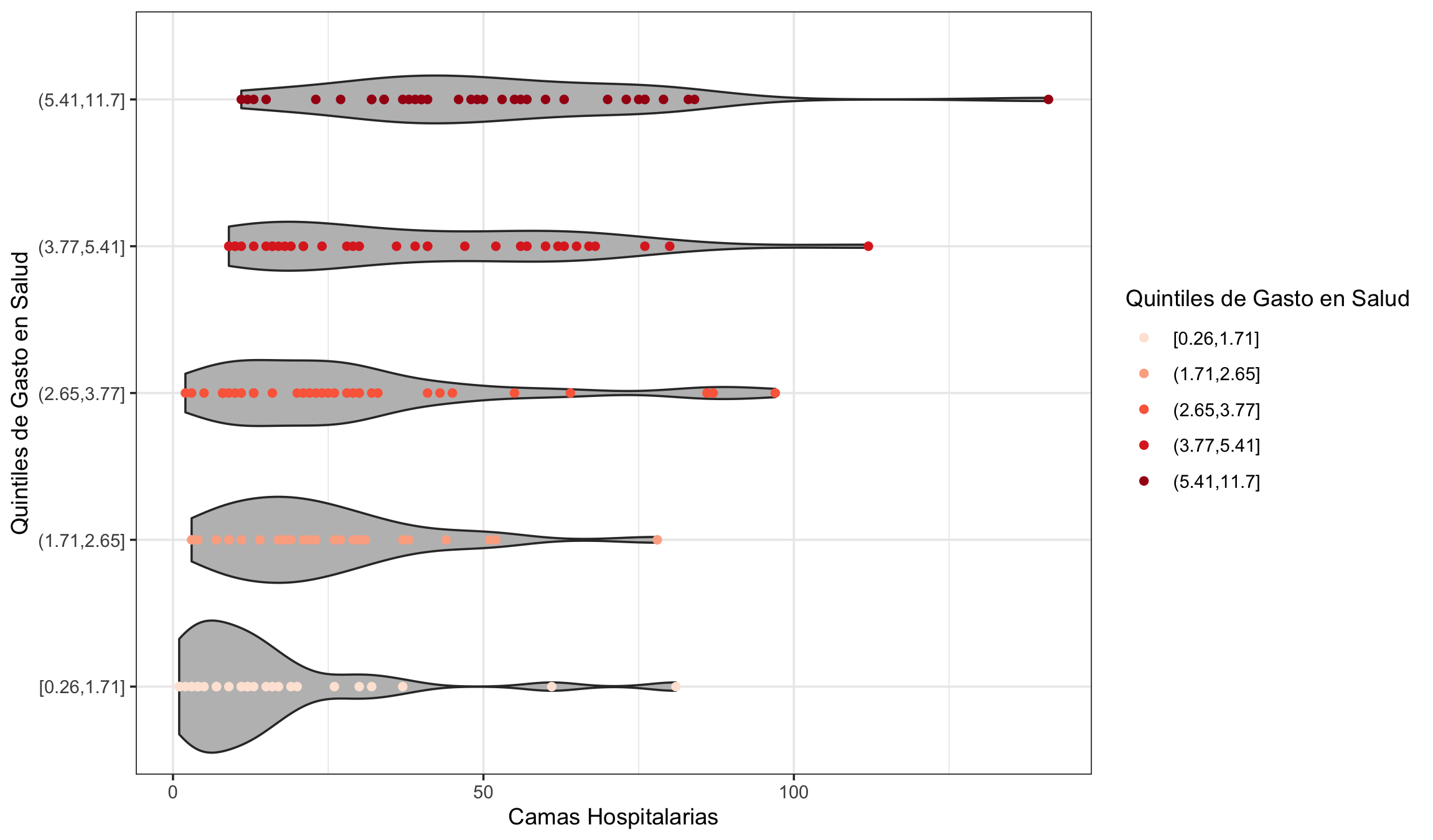
A) Reproducir el siguiente gráfico que muestra la diferencia en la distribución de camas hospitalarias (hospital_beds) en los diferentes quintiles de gasto en salud (contruir en base a la variable health_expenditure_gdp).