4 tidyverse II: tidyr y uniones
4.1 Paquetes y data
Para esta sección, continuaremos usando funciones del paquete tidyverse y los mismos datos de who y covid del capítulo anterior.
Cargamos el paquete tidyverse:
Ahora, leeremos los archivos de datos a utilizar:
El conjunto de datos
whocontiene datos del Global Health Observatory de la Organización Mundial de la Salud (OMS). Contiene 359 variables para 202 países y territorios. El diccionario de este conjunto de datos se encuentra en Anexos.El conjunto de datos
covidcontiene los datos de casos de COVID-19 reportados por el Ministerio de Salud del Perú en la Plataforma Nacional de Datos Abiertos (hasta el15-09-2020). Contiene las siguientes variables:
| Variable | Descripción |
|---|---|
| date | Fecha de reporte |
| REGION | Región (Adm01) |
| new_cases | Casos nuevos de COVID-19 |
4.2 Paquete tidyr
Antes de comenzar a usar algunas de las funciones de la paquete tidyr, haremos una breve explicación de lo que significa tener un conjunto de datos en formato long y en formato wide.
Long: Las características son almacenadas en dos variables
keyyvalue. Enkeyse tiene el label de la característica a representar y envalueel valor respectivo. Este conjunto de datos rectangular tiene más filas que columnas.Wide: Para cada observación, las características están contenidas en variables. Este conjunto de datos rectangular tiene más columnas que filas.
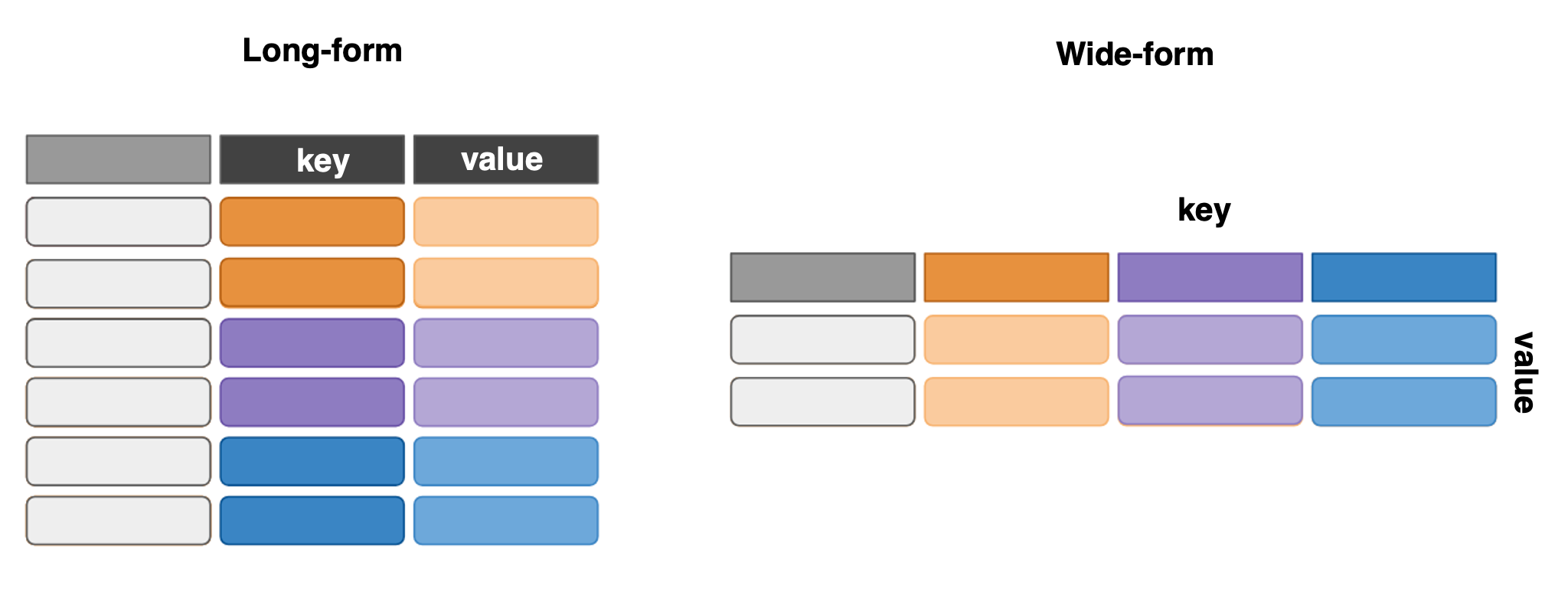
El conjunto de datos covid está en formato long, es decir, tiene más filas que columnas. La key es la fecha y el value son los casos y/o muertos.
EL conjunto de datos who está en formato wide, es decir, tiene más columnas que filas. Cada variable está almacenada en una columna.
La función dim() muestra las dimensiones (# filas, # columnas) del conjunto de datos.
4.2.1 Verbo pivot_wider()
La función pivot_wider() nos permite transformar un conjunto de datos de formato long a formato wide. La función tiene los siguientes parámetros principales:
data %>%
pivot_wider(
names_from,
values_from,
values_fill = NULL
)-
data= El dataframe con el que se trabajará. -
names_from= Nombre de la variable que contiene los valores que pasarán a ser columnas. -
values_from= Nombre de la variable que contiene los valores que llenarán las columnas. -
values_fill= Es opcional e indica qué debe pasar si hay valores missing (ej.values_fill = 0).
covid_wide <- covid %>%
pivot_wider(
names_from = date,
values_from = new_cases
)Ahora los datos (número de casos) están en columnas por fecha. Después de la transformación hay más columnas que filas, es decir, los datos están en formato wide.
dim(covid_wide)[1] 25 195Antes de la versión [tidyr 1.0], la función que se usaba para estos fines era spread(), pero ya está en desuso y pronto desaparecerá del paquete.
4.2.2 Verbo pivot_longer()
La función pivot_longer() es el operador opuesto a pivot_wider(), es decir, convierte un conjunto de datos de formato wide a long. La función tiene los siguientes principales parámetros:
data %>%
pivot_longer(
cols,
names_to,
values_to,
values_drop_na = FALSE
)-
data= El dataframe con el que se trabajará. -
cols= Todas las variables a las que se aplicará la función. Se puede usar funciones<tidy-select>. -
names_to= Es el nombre de la variable donde se agrupará loscolnames()de las columnas afectadas. -
values_to= Es el nombre de la variable donde se almacenará los valores de las columnas en análisis. -
values_drop_na= Si esTRUE, se retirará las observaciones donde haya valoresNA.
who_long <- who %>%
pivot_longer(
cols = population:tb_treatment_success,
names_to = "variable",
values_to = "count"
)Hemos convertido el conjunto de datos who a formato long. Hemos preservado las variables identificadoras del país y continente porque hemos especificado cuáles son las variables que ingresarán a la transformación population:tb_treatment_success.
dim(who_long)[1] 4444 6Antes de la versión [tidyr 1.0], la función que se usaba para estos fines era gather(), pero ya está en desuso y pronto desaparecerá del paquete.
4.2.3 Verbo unite()
La función unite() convierte el contenido de varias variables en una sola. Podemos agregar un separador con el argumento sep y controlar si las variables se eliminan después de juntarse con el argumento remove.
4.2.4 Verbo separate()
La función separate() es el operador opuesto a unite(). Convierte el contenido de una variable en múltiples variables. Tenemos que especificar el nombre de las variables que van a recibir el contenido separado (into). Podemos controlar si la variable original es eliminada con remove.
4.3 Manejo de textos
El proceso de manejo de variables que contengan texto (character) es parte esencial y fundamental durante un análisis de datos. Aunque en algunas ocasiones, este proceso sea mínimo o nulo, puede darse el caso de que nos enfrentemos a distintos tipos de situaciones, como el renombrar variables con tildes, espacios o nombramiento irregular, o tener variables con informaciones como identificadores personales (ej. DNI), condiciones, diagnósticos, etc., que puedan requerir tareas específicas. En esta sección desarrollaremos algunas de las tareas principales asociados al manejo de textos.
4.3.1 Limpieza de nombre de variables
En algunas ocasiones nos enfrentamos a una base de datos que puede tener más de una forma de nombrar a las variables, con situaciones como:
- Nombres con mayúsculas, minúsculas. Ej:
Var1,VAR2,var4 - Nombres con y sin espacio. Ej:
Var1,Var 2,Var 3 - Presencia de tildes o carácteres extraños. Ej:
Var única,var ✔,var ✘
Veamos un ejemplo con los datos con la siguiente estructura:
dirty_data <- tibble(
"Var1" = rnorm(10),
"var 2" = rnorm(10),
"VAR ÚNICA" = rnorm(10),
"var ✔" = rnorm(10)
)
dirty_data# A tibble: 10 × 4
Var1 `var 2` `VAR ÚNICA` `var ✔`
<dbl> <dbl> <dbl> <dbl>
1 1.90 -1.33 -0.221 -1.04
2 1.11 1.56 -0.654 1.75
3 0.133 0.517 -0.235 -1.98
4 0.307 -0.659 1.05 -0.583
5 -0.735 0.0848 1.07 -0.251
6 -0.109 -0.415 -0.475 -1.10
7 -1.17 -0.592 -1.46 0.757
8 0.707 0.211 0.263 -0.658
9 1.70 0.581 -0.717 -0.891
10 -0.152 -0.690 1.03 -1.28 Para lograr la limpieza de los nombres de las columnas podemos utilizar la función clean_names() del paquete janitor que tiene varias pre-configuraciones para la limpieza.
Attaching package: 'janitor'The following objects are masked from 'package:stats':
chisq.test, fisher.testclean_names(dirty_data)# A tibble: 10 × 4
var1 var_2 var_unica var
<dbl> <dbl> <dbl> <dbl>
1 1.90 -1.33 -0.221 -1.04
2 1.11 1.56 -0.654 1.75
3 0.133 0.517 -0.235 -1.98
4 0.307 -0.659 1.05 -0.583
5 -0.735 0.0848 1.07 -0.251
6 -0.109 -0.415 -0.475 -1.10
7 -1.17 -0.592 -1.46 0.757
8 0.707 0.211 0.263 -0.658
9 1.70 0.581 -0.717 -0.891
10 -0.152 -0.690 1.03 -1.28 Como se observa, todas las variables pasan a minúscula, se reemplaza los espacios con _ y se retira la tilde así como caracteres extraños.
4.3.2 Detectar patrón de texto
Poder detectar un patrón de texto es una tarea recurrente cuando nos enfrentamos a un proceso de manipulación de texto. Esto comprende el señalar una coincidencia exacta, parcial, o definir un patrón más genérico de como podría estar conformado un texto. Para ejemplificar estos distintos escenarios, emplearemos la función str_detect() del paquete stringr que se carga con tidyverse. Esta función tiene 3 argumentos básicos: str_detect(string, pattern, negate = FALSE). El primero recibe al texto (vector/columna) que contenga los caracteres; el segundo, el patrón de texto; y el tercero, si se indica en TRUE estaría pidiéndose que se devuelva lo contrario a lo deseado, es decir saber los elementos con los que no se encontró coincidencia.
Imaginemos el siguiente vector:
situacion_texto<- c("diagnóstico", "síntomas", "medicamento", "síntomas2", "000", "correo@gmail.com")- Coincidencia exacta: En este caso se busca detectar un texto tal y como está escrito. Por ej. detectar a la palabra medicamento:
str_detect(situacion_texto, "medicamento")[1] FALSE FALSE TRUE FALSE FALSE FALSE- Coincidencia parcial: En el caso de la coincidencia parcial, solo requiere la escritura de una parte de la variable para buscar las coincidencias. Por ej.
str_detect(situacion_texto, "sínto")[1] FALSE TRUE FALSE TRUE FALSE FALSE-
Patrón genérico:
- Detectar que el texto que tenga al menos 1 número: Eso se podrá lograr gracias a una especificación llamada regex. No profundizaremos demasiado sobre esto, pero si algunos ejemplos importantes en el manejo, como
[0-9]+, que básicamente revisará que en el texto haya cualquier número entre 0 a 9 ([0-9]) que pueda repetirse más de una vez (+):[0-9]+.
str_detect(situacion_texto, "[0-9]+")[1] FALSE FALSE FALSE TRUE TRUE FALSE- Detectar que el texto solo contenga caracteres: De forma similar a lo anterior se buscará que hay alguna letra entre la
ay lazen el texto, con la diferencia que tendremos que incluir en esta ocasión las letras en mayúsculasAaZen la verificación.
str_detect(situacion_texto, "[A-Za-z]")[1] TRUE TRUE TRUE TRUE FALSE TRUE- Detectar que el texto contengan un correo (gmail.com?): En este caso buscamos algo muy parecido a la coincidencia parcial, agregando una especificación adicional, y es que este texto de
@gmail.comdebe estar al final del texto. Esto lo conseguimos agregando un$al final del patrón.
str_detect(situacion_texto, "@gmail.com$")[1] FALSE FALSE FALSE FALSE FALSE TRUE - Detectar que el texto que tenga al menos 1 número: Eso se podrá lograr gracias a una especificación llamada regex. No profundizaremos demasiado sobre esto, pero si algunos ejemplos importantes en el manejo, como
Ahora, que str_detect() tenga como devolución TRUE o FALSE tiene distintos usos potenciales, pero podemos concentrarnos en 2:
- Filtrado de casos de una variable: Si en la data
whoquisiéremos filtrar a todos los contienentes que tengan el términoAmericaen su nombre, podríamos:
who %>%
filter(str_detect(continent, "America"))- Recodificación de variable en base a coincidencia: En este caso usaremos la función
mutate()para modificar una columna en base a una condición (case_when()): que si un contienente contiene el términoAmericaen su nombre, el país deberá estar totalmente en mayúscula, para lo cual usaremos la funciónstr_to_upper().
who %>%
mutate(
country = case_when(
str_detect(continent, "America") ~ str_to_upper(country),
.default = country
)
)str_to_upper() (note)
Existen algunas otras funciones que logran cambios rápidos bajo esta dinámica, como lo son:
-
str_to_lower(): lleva a todas los caracteres a minúsculas. -
str_to_title: en un texto de más de una palabra, eleva a todas en sus primeras letras a mayúsculas. Por ej.
4.3.3 Extraer, reemplazar y eliminar texto
En otras situaciones, además de detectar un patrón específico de texto, será necesario poder extraerlo (str_extract()), eliminarlo (str_remove()) o reemplazarlo (str_replace()). Cada una de estas tareas tiene su propia función específica con la que se podrá proceder, y tienen como segundo argumento el patrón de texto regex, indicado previamente (ej. [0-9]+).
Vamos a observar y analizar el uso de estas funciones en base a un objetivo de análisis que puede ser recurrente, y es el hecho de uniformizar los valores de una variable: continent. Veamos ahora mismo como se encuentran codificados los elementos de esta variable:
# A tibble: 7 × 2
continent n
<chr> <int>
1 Africa 48
2 Eastern Mediterranean 21
3 Europe 51
4 North and Central America 7
5 South America 31
6 South East Asia 9
7 Western Pacific 35Como observamos, se usa una clasificación que podría no ser el que estamos buscando. De similar forma a este, podríamos tener variables con 10, 20, o 50 formas distintas de haber sido escritas que podrías querer uniformizar. Por ej., si estuvieramos en una situación en el que tenemos Africa, africa y AFRICA, convendría primero reducir estas múltiples forma de escribir un mismo nombre con la función str_to_lower() que vimos anteriormente.
En este caso, tenemos una sola forma de escribir por clasificación, pero si podríamos por ejemplo, usar solo la clasificación America en lugar de North and Central America y South America. Es lo que intentaremos realizar con la función str_extract()
who %>%
mutate(
continent = str_extract(continent, "America")
)# A tibble: 202 × 26
continent_id country_id continent country population urban_population
<dbl> <dbl> <chr> <chr> <dbl> <dbl>
1 1 1 <NA> Afghanistan 29900000 5740436
2 2 2 <NA> Albania 3563112 1431794.
3 3 3 <NA> Algeria 32500000 20800000
4 2 4 <NA> Andorra NA NA
5 3 5 <NA> Angola 11800000 8578749
6 4 6 America Antigua and Ba… 68722 32468.
7 5 7 America Argentina 39500000 34900000
8 2 8 <NA> Armenia 2982904 1934321.
9 6 9 <NA> Australia 20100000 18000000
10 2 10 <NA> Austria 8184691 5433978
# ℹ 192 more rows
# ℹ 20 more variables: pop_under_poverty_line <dbl>,
# life_expectancy_birth <dbl>, inequality_index <dbl>, literacy_rate <dbl>,
# literacy_rate_males <dbl>, literacy_rate_females <dbl>,
# co2_economic_output <dbl>, gni_per_capita <dbl>,
# health_expenditure_gdp <dbl>, health_expenditure_person <dbl>,
# hospital_beds <dbl>, children_out_school_primary <dbl>, …Aunque, logramos quedarnos solamente con el texto America, observamos que el resto de las clasificaciones pasaron a ser NA:
who %>%
mutate(
continent = str_extract(continent, "America")
) %>%
count(continent)# A tibble: 2 × 2
continent n
<chr> <int>
1 America 38
2 <NA> 164Esto significa que deberíamos especificar más de una coincidencia en lo que estamos intentado realizar. Para lograr esto podemos utilizar el operador |, que significa o, encapsulado entre paréntesis: (America|Asia|Africa|Europe). De esta manera estaríamos buscando ya sea el texto America, Asia, Africa o Europa, cualquiera de esas palabras:
who %>%
mutate(
continent = str_extract(continent, "(America|Asia|Africa|Europe)")
) %>%
count(continent)# A tibble: 5 × 2
continent n
<chr> <int>
1 Africa 48
2 America 38
3 Asia 9
4 Europe 51
5 <NA> 56Obtenemos un mejor resultado, pero perdemos algunas otras clasificaciones importantes como Eastern Mediterranean y Western Pacific, lo cual no es nuestra intención. Podríamos primero reemplazar esas clasificaciones re-clasificándolas en algunas de las categorías anteriores, pero esto solo funcionaría en este caso en específico. Recordemos que podríamos enfrentarnos a variables con 10, 20, o 50 formas distintas de haber sido escritas o clasificadas, y hacer esta operación de renombrar manualmente, no es lo ideal. Por lo que intentaremos otro enfoque, ahora con str_remove():
who %>%
mutate(
continent = str_remove(continent, "(North|Central|South|East|and)")
) %>%
count(continent)# A tibble: 7 × 2
continent n
<chr> <int>
1 " America" 31
2 " East Asia" 9
3 " and Central America" 7
4 "Africa" 48
5 "Europe" 51
6 "Western Pacific" 35
7 "ern Mediterranean" 21Esta vez estamos intentando eliminar los textos adicionales que bordean a America o Asia y así lograr lo que estamos buscando. Sin embargo, nos enfrentamos a algunos problemas:
- En algunas palabras solo se borró la primera coincidencia encontrada. Por ej.
South East Asiapasó aEast Asiay ya no borró aEasta pesar de estar mencionado. - Algunas palabras parecen haberse afectado a pesar de que no era nuestra intención afectarlas. Por ej.
Eastern Mediterraneanpasó a serern Mediterranean, debido a que el textoEasterntiene parcial coincidencia conEast. - Finalmente, hay espacios a lado izquierdo de los nombres. Por ej.
Americaen lugar deAmerica
Vamos a revisarlo punto por punto para ver cómo podemos solucionar cada uno de estos efectos indeseados al emplear str_remove():
- En el caso de que solo se está borrando la primera coincidencia encontrada, esto es un comportamiento deseado de la función. Al revisar la documentación observaremos que hay 2 versiones para esta misma idea de tarea:
str_remove()ystr_remove_all(). La primera solo borrará la primera coincidencia encontrada; mientras que la segunda función, todas las coincidencias que haya en un texto.
who %>%
mutate(
continent = str_remove_all(continent, "(North|Central|South|East|and)")
) %>%
count(continent)# A tibble: 7 × 2
continent n
<chr> <int>
1 " America" 7
2 " Asia" 9
3 " America" 31
4 "Africa" 48
5 "Europe" 51
6 "Western Pacific" 35
7 "ern Mediterranean" 21Vemos que efectivamente South East Asia pasó a ser Asia ya sin la palabra East a su izquierda. Por lo que logramos solucionar este primer problema.
- El segundo problema está asociado a que los términos que ingresamos en múltiples coincidencias no están buscando solo palabras completas, sino cualquier coincidencia en texto que exista. Para lograr que busquen palabras completas, tenemos que encampsular al patrón regex en con
\\bal inicio y al término de la palabra. Por ej. si quisiera retirar solo la palabraEastcuando es exactamente así (solo palabra aislada), podríamos escribirlo como\\b(East)\\b. Probemos:
who %>%
mutate(
continent = str_remove_all(continent, "\\b(North|Central|South|East|and)\\b")
) %>%
count(continent)# A tibble: 7 × 2
continent n
<chr> <int>
1 " America" 7
2 " Asia" 9
3 " America" 31
4 "Africa" 48
5 "Eastern Mediterranean" 21
6 "Europe" 51
7 "Western Pacific" 35Podemos ver que ahora Eastern Mediterranean se mantiene como tal a pesar de que tenga la primera parte de su nombre una coincidencia parcial con East.
- Finalmente, lo que observamos se encuentra más cerca de lo que buscamos, pero tienen espacios indeseados dentro del texto. Podríamos buscar eliminar esos espacios uno por uno, pero esto sería problemático. Para ello, existe la función
str_squish()que nos permitirá rápidamente eliminar espacios indeseados. Probemos:
who %>%
mutate(
continent = str_remove_all(continent, "\\b(North|Central|South|East|and)\\b"),
continent = str_squish(continent)
) %>%
count(continent)# A tibble: 6 × 2
continent n
<chr> <int>
1 Africa 48
2 America 38
3 Asia 9
4 Eastern Mediterranean 21
5 Europe 51
6 Western Pacific 35Podemos apreciar que ahora la variable continent se encuentra de la forma en cómo buscábamos que quede. Conviene señalar que str_remove() es una forma simplificada de str_replace(), en el que se tiene predeterminado que se reemplazará una palabra con nada, entendido como "" un texto vacío que se encuentra dentro de las comillas. Por lo que podríamos conseguir exactamente el mismo efecto con str_replace():
who %>%
mutate(
continent = str_replace_all(continent, "\\b(North|Central|South|East|and)\\b", ""),
continent = str_squish(continent)
) %>%
count(continent)# A tibble: 6 × 2
continent n
<chr> <int>
1 Africa 48
2 America 38
3 Asia 9
4 Eastern Mediterranean 21
5 Europe 51
6 Western Pacific 35La ventaja de ello es que podríamos no solo reemplazar un patrón de texto con nada (es decir, eliminarlo), sino con algún texto en específico.
str_extract()|str_remove()|str_replace() (note)
Como habrán podido notar, str_remove() no es el único que tiene una extensión con terminación _all(). Dentro del paquete stringr hay algunas otras funciones que lo tienen. En el caso de las funciones que observamos en esta sección, todas pueden usarse de esa manera:
| Función | Extensión |
|---|---|
str_extract() |
str_extract_all() |
str_remove() |
str_remove_all() |
str_replace() |
str_replace_all() |
4.3.4 Rellenar texto con str_pad()
En algunas situaciones tenemos variables que sirven de identificador (código de hospital, código de inventariado, etc.) o lo son en realidad (DNI, historia médica, etc.), y tienen una nomenclatura específica. Por ej. 001, 002, … 142. Sin embargo, al momento de almacenarse la información en un archivo excel o haber sido modificado por otras personas, este formato o nomenclatura se pierde y llega a nostros totalmente numérico, es decir: 1, 2, … 142. En estas ocasiones, str_pad() puede ayudarnos con rapidez a recuperar esta estructura, rellenándo el texto en base a un texto e indicando el ancho o longitud que debería tener. En el mismo ejemplo, 1 que tiene un ancho o longitud de 1, debería ser rellenado con 0 hasta que ahora tenga un ancho o longitud de 3:
str_pad("1", width = 3, pad = "0")[1] "001"Podemos observar en nuestra data who que la variable country_id es un identificador del país y tenemos 202 filas. Intentemos replicar este comportamiento en esta columna:
De esta manera tendríamos recuperado el formato o nomenclatura deseada en nuestros datos.
4.4 Uniones
Existen diversas opciones para poder unir conjuntos de datos dependiendo de lo que queremos obtener. En esta sección, exploraremos las opciones que ofrece el paquete dplyr.
dplyr tiene dos funciones para juntar conjuntos de datos respetando el orden de las columnas/filas. La función bind_rows() permite juntar dos conjuntos de datos de manera vertical. La función bind_cols() permite juntar dos conjuntos de datos de manera horizontal.
En este libro, nos enfocaremos en funciones de la familia *_join(), que permiten la unión de conjuntos de datos en base a una variable de enlace.
Para este ejemplo, usaremos un conjunto de datos covid_reg que contiene observaciones de 25 regiones (filas) y el total de casos (columnas).
Uniremos estos datos con un conjunto de datos adicional (reg_vars) que contiene algunas variables adicionales por región que provienen de otra fuente de información (INEI). Por motivos didácticos, el conjunto de datos no contiene datos de algunas regiones.
reg_vars <- read_csv("data/regions.csv")4.4.1 Verbo inner_join()
La función inner_join() retorna solo las observaciones que se encuentran presentes en ambos conjuntos de datos. El resultado contiene todas las columnas de ambos conjuntos de datos. Usamos el argumento by para definir la variable de enlace (puede ser más de una).
covid_reg %>%
inner_join(reg_vars, by = "REGION")4.4.2 Verbo semi_join()
La función semi_join() retorna solo las observaciones que se encuentran presentes en ambos conjuntos de datos, y solo las columnas del primer conjunto de datos.
4.4.3 Verbo left_join()
La función left_join() retorna todos las observaciones que se encuentran presentes en el primer conjunto de datos, con todas las columnas de ambos conjuntos de datos.
4.4.4 Verbo right_join()
La función right_join() retorna todos las observaciones que se encuentran presentes en el segundo conjunto de datos, con todas las columnas de ambos conjuntos de datos.
covid_reg %>%
right_join(reg_vars, by = "REGION")4.4.5 Verbo anti_join()
La función anti_join() retorna todos las observaciones que se encuentran presentes en el primer conjunto de datos y no están presentes en el segundo conjunto de datos, y solo las columnas del primer conjunto de datos.
4.4.6 Verbo full_join()
La función full_join() retorna todos las observaciones de ambos conjuntos de datos, y todas las columnas de ambos conjuntos de datos.