7 Extensiones gráficas
7.1 Paquetes y Data
Los paquetes que se utilizarán en esta sección son:
Utilizaremos una base de datos ficticia que fue creada usando como referencia los datos abiertos del Gobierno Peruano. Esta base contiene los registros de cada persona diagnosticada de COVID-19 por el Ministerio de Salud (MINSA) hasta el 24-MAY-2021.
Los datos de georeferenciación (coordenadas) de los casos fueron simulados para propósitos didácticos. Puede descargar directamente la base de datos del repositorio de Zenodo
covid <- read_csv("data/covid19-district.csv") 7.2 Estructuras temporales
La característica más importante de los datos temporales es que las observaciones están relacionadas entre ellas. Es decir, las observaciones siguen una secuencia ordenada en función al atributo tiempo. Exploraremos las representaciones gráficas más comunes para este tipo de estructura de datos.
7.2.1 Gráfico de líneas simple
La forma más básica de representar este tipo de estructuras es a través de un gráfico de líneas (geom_line()).
Como nuestra base de datos es a nivel individual, haremos el conteo del número de observaciones para cada fecha y distrito. Construiremos unos datos de tipo
panel, para lo cual completaremos con0todas las fechas de las unidades geográficas que no reportaron casos. Usaremos la funcióncompletedel paquete tidyr que se encuentra dentro del tidyverse.
Para este ejemplo solo usaremos los datos del distrito de Lima.
covid_count %>%
filter(DISTRITO == "LIMA") %>%
ggplot(aes(x = FECHA_RESULTADO, y = casos)) +
geom_line()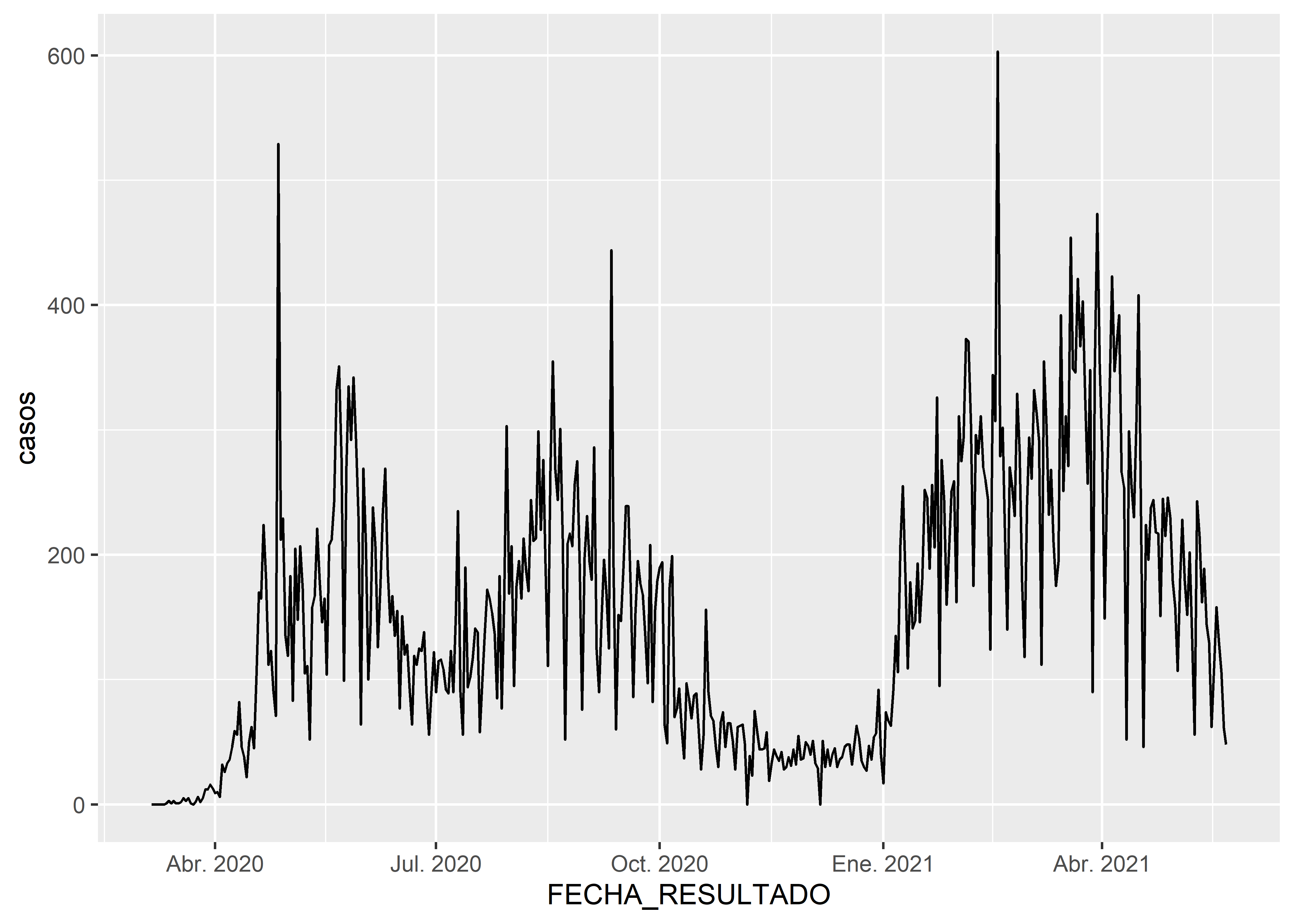
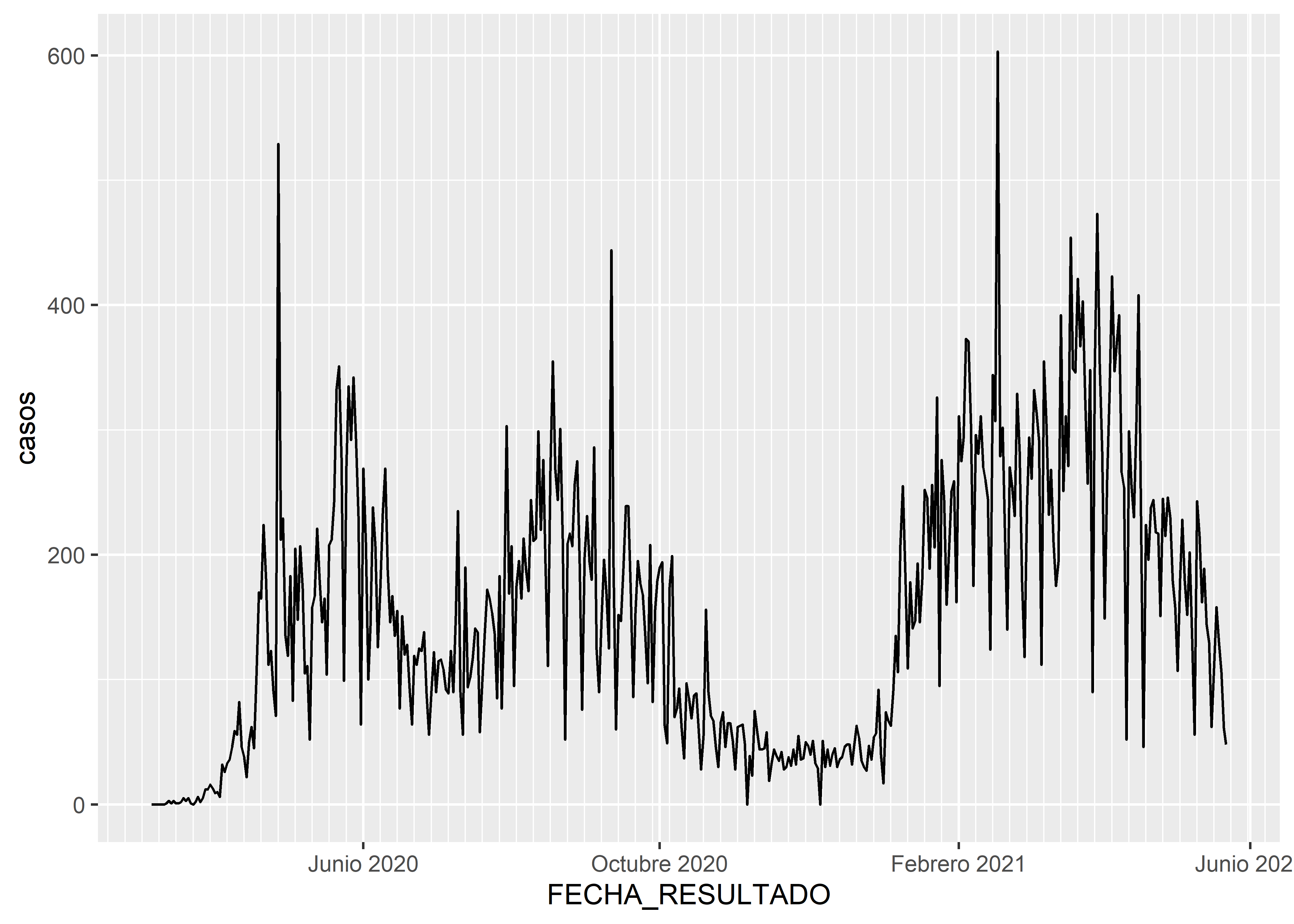
Podemos cambiar la escala temporal (eje x) con la funcion scale_x_date(). Se puede controlar las divisiones mayores (ej. date_breaks = "1 month"), las divisiones menores (ej. date_minor_breaks = "1 week") y el formato de las etiquetas (ej. date_labels = "%B"). Puedes revisar más información sobre los formatos de datos tipo Date o POSIXct en el siguiente link.
covid_count %>%
filter(DISTRITO == "LIMA") %>%
ggplot(aes(x = FECHA_RESULTADO, y = casos)) +
geom_line() +
scale_x_date(
date_breaks = "4 month", date_minor_breaks = "1 week", date_labels = "%B %Y"
)
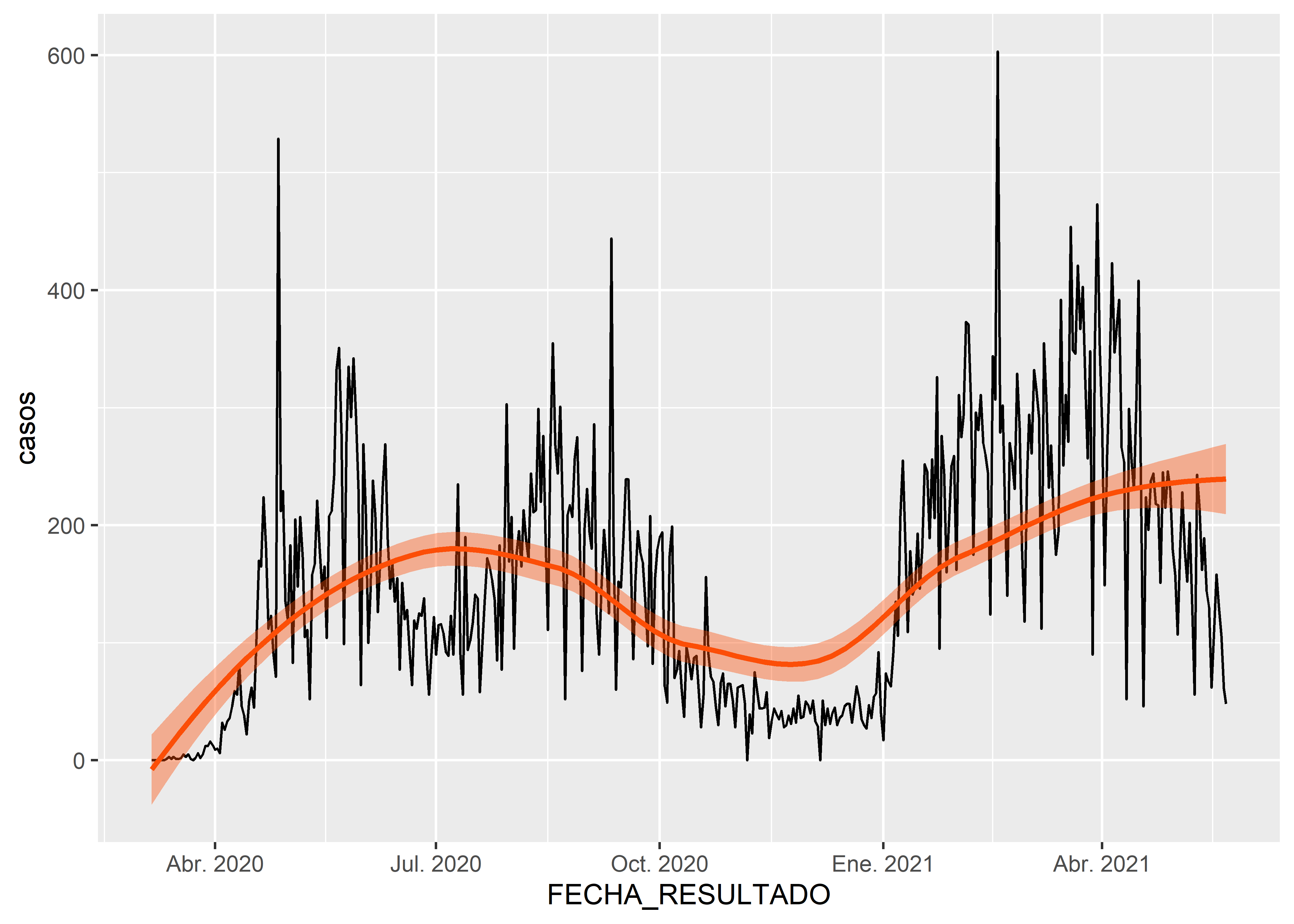
Se puede agregar una línea de tendencia usando la función geom_smooth(). Por defecto, la función geom_smooth() utiliza un metodo de loess; el método se puede ajustar con el argumento method.
7.2.2 Gráfico de líneas estratificado
Cuando tenemos gran cantidad de información en un mismo gráfico se puede perder la narrativa de lo que se quiere mostrar. En estas series temporales de todos los distritos en la base de datos covid hemos cambiado la escala del eje y (scale_y_log10()) para observar con más detalle la series temporales de aquellos distritos con poco número de casos. Sin embargo, la lectura del gráfico es ahora más dificultosa.
covid_count %>%
ggplot(aes(x = FECHA_RESULTADO, y = casos, col=DISTRITO)) +
geom_line() +
scale_y_log10() +
theme(legend.position = "bottom")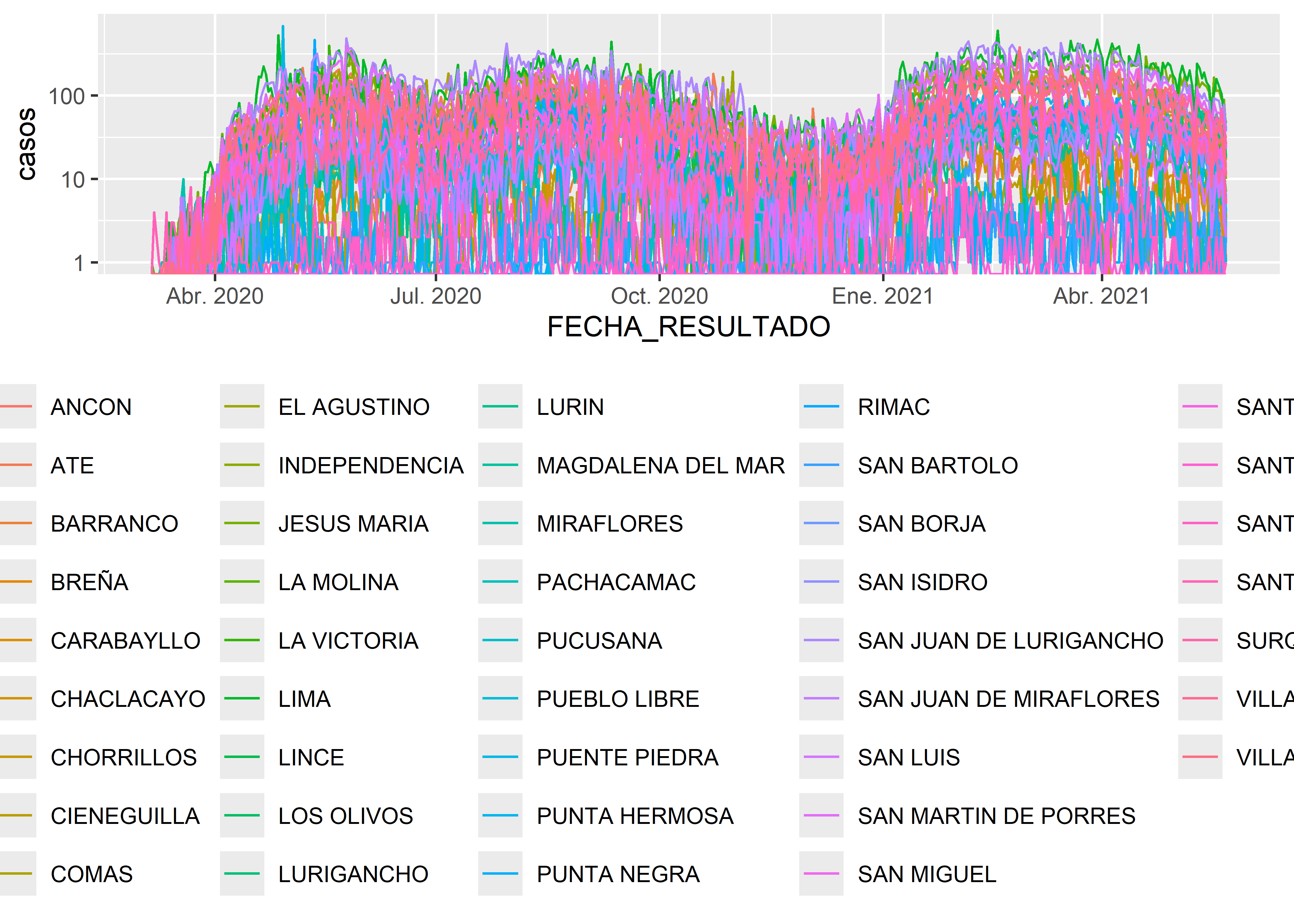
Si queremos mostrar la tendencia de dos distritos, por ejemplo MIRAFLORES y RIMAC, podemos optar por filtrar las series temporales de esos distritos.
covid_count %>%
filter(DISTRITO == "MIRAFLORES" | DISTRITO == "RIMAC") %>%
ggplot(aes(x = FECHA_RESULTADO, y = casos, col=DISTRITO)) +
geom_line() +
scale_y_log10()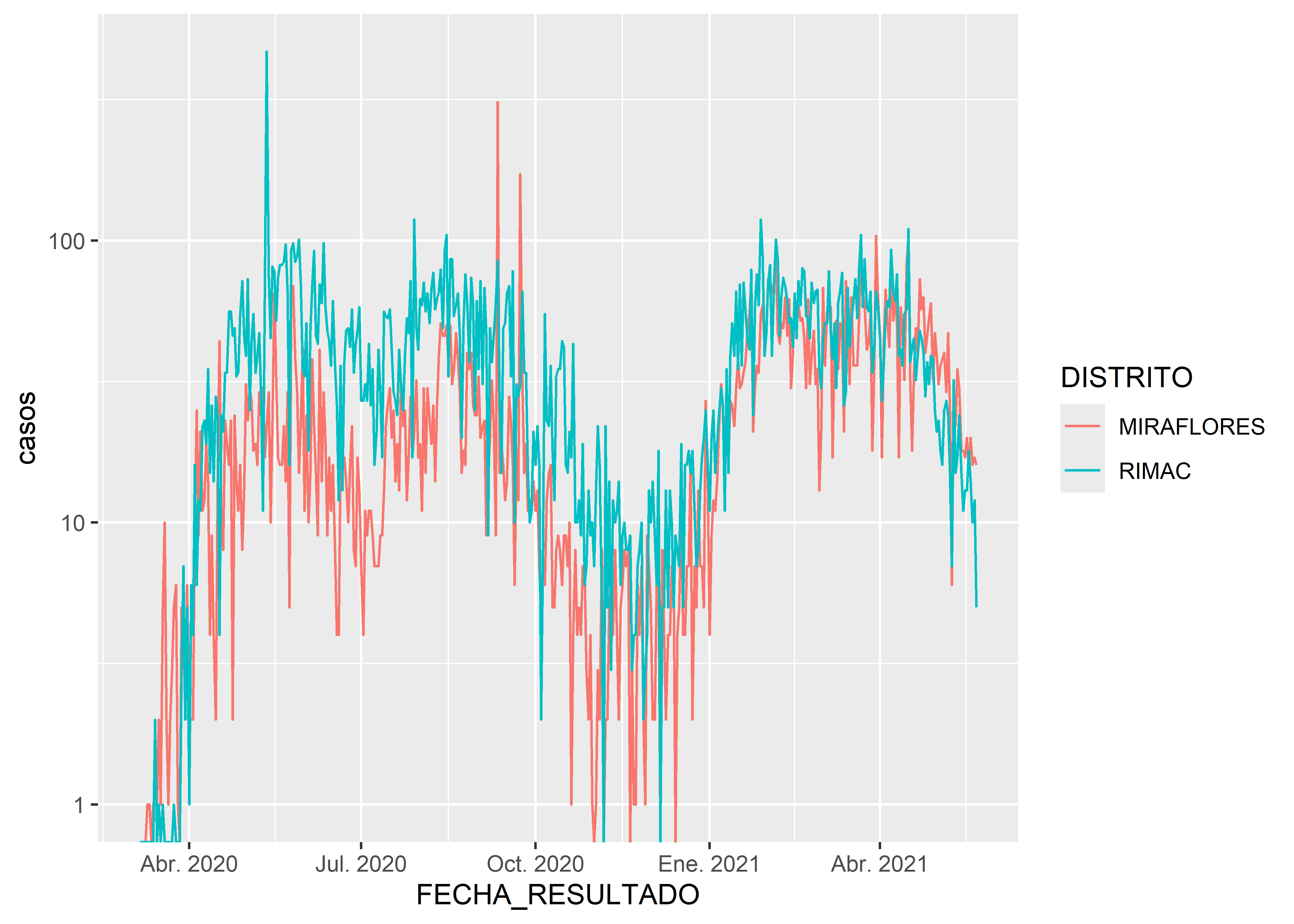
Sin embargo, al hacer esto se pierde el contexto de la evolución de los otros distritos. Podemos utilizar el paquete gghighlight para resaltar aquellos elementos (en este caso DISTRITOS) en los que se quiere hacer énfasis.
library(gghighlight)
covid_count %>%
ggplot(aes(x = FECHA_RESULTADO, y = casos, col=DISTRITO)) +
geom_line() +
scale_y_log10() +
gghighlight(DISTRITO == "MIRAFLORES" | DISTRITO == "RIMAC")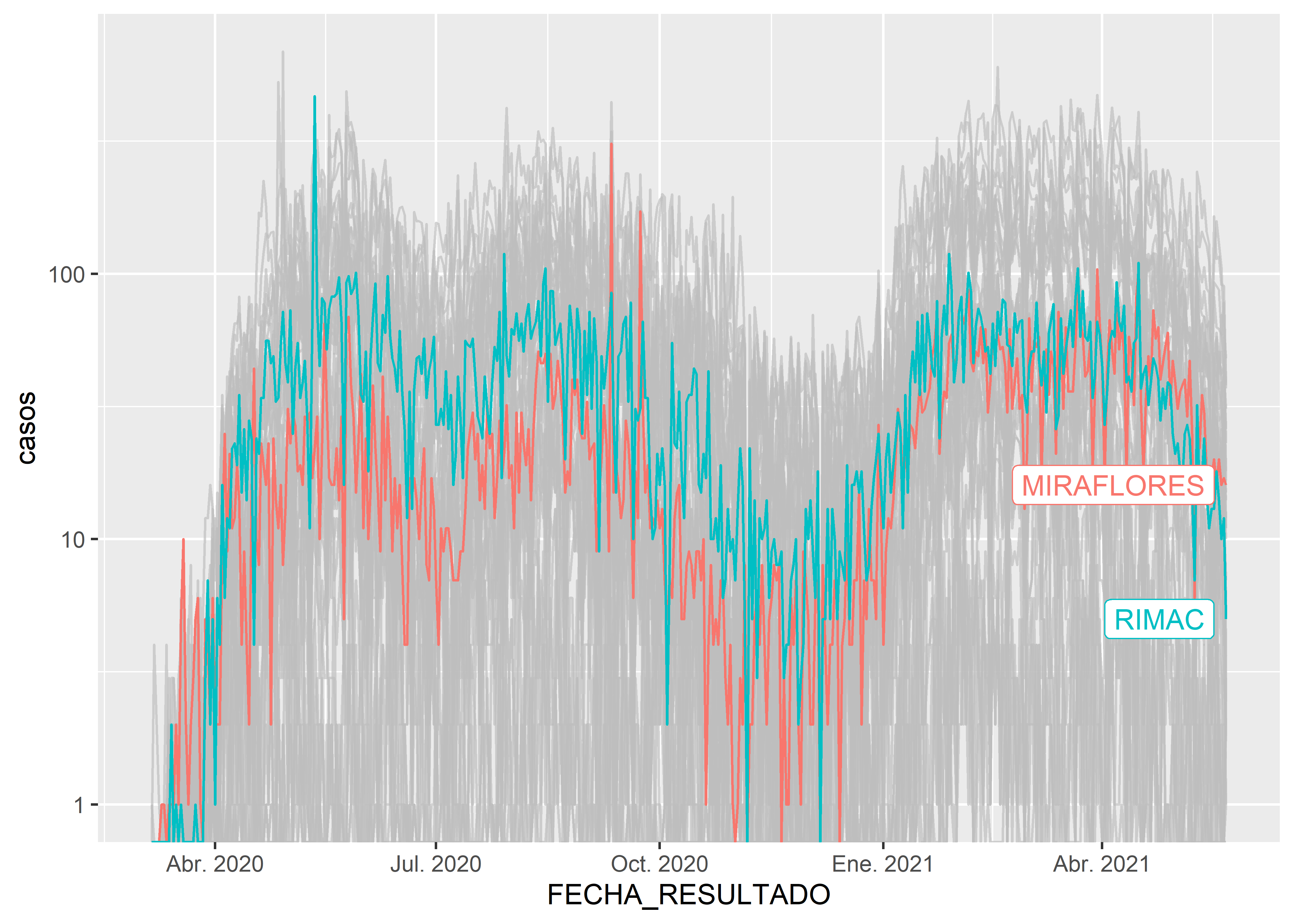
También podemos hacer la selección de las series a destacar en base a alguna estadística de los datos. Por ejemplo, queremos destacar todos los distritos que tienen un total de casos acumulados mayor a 45,000 en lo que va de la pandemia.
covid_count %>%
ggplot(aes(x = FECHA_RESULTADO, y = casos, col=DISTRITO)) +
geom_line() +
scale_y_log10() +
gghighlight(sum(casos)>45000)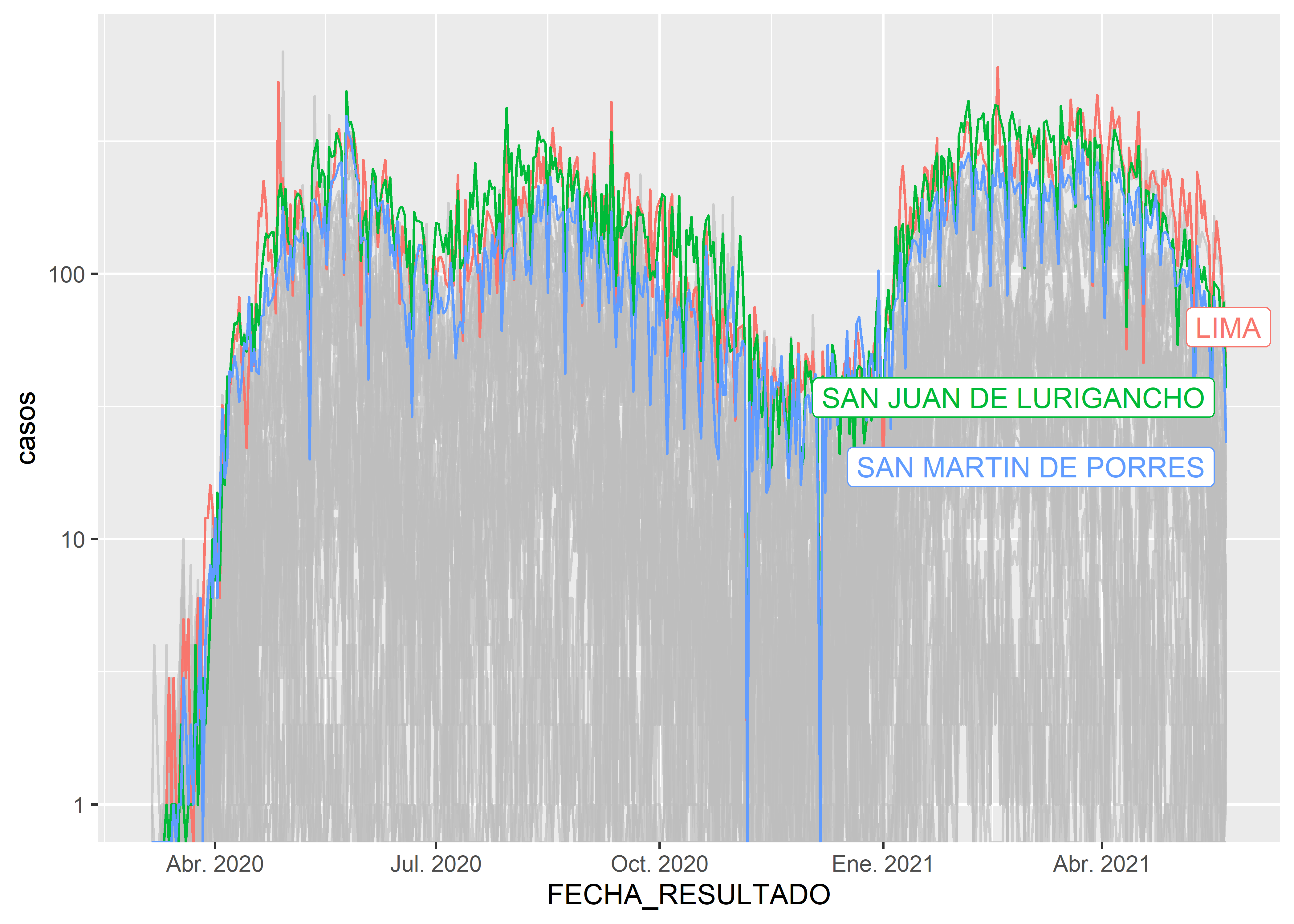
Al estar en el universo
tidyverse, podemos agregar las funciones de composición gráfica (como escalas y temas).
library(ggsci)
covid_count %>%
ggplot(aes(x = FECHA_RESULTADO, y = casos, col = DISTRITO)) +
geom_line() +
scale_y_log10() +
gghighlight(sum(casos) > 45000) +
scale_color_npg() +
labs(
y = "Casos Nuevos", x = "Fecha",
caption = "Datos del Ministerio de Salud (MINSA) \nElaboracióbn:@gabc91"
) +
ggtitle("Tendencia de casos nuevos de COVID-19 en Peru") +
theme_minimal()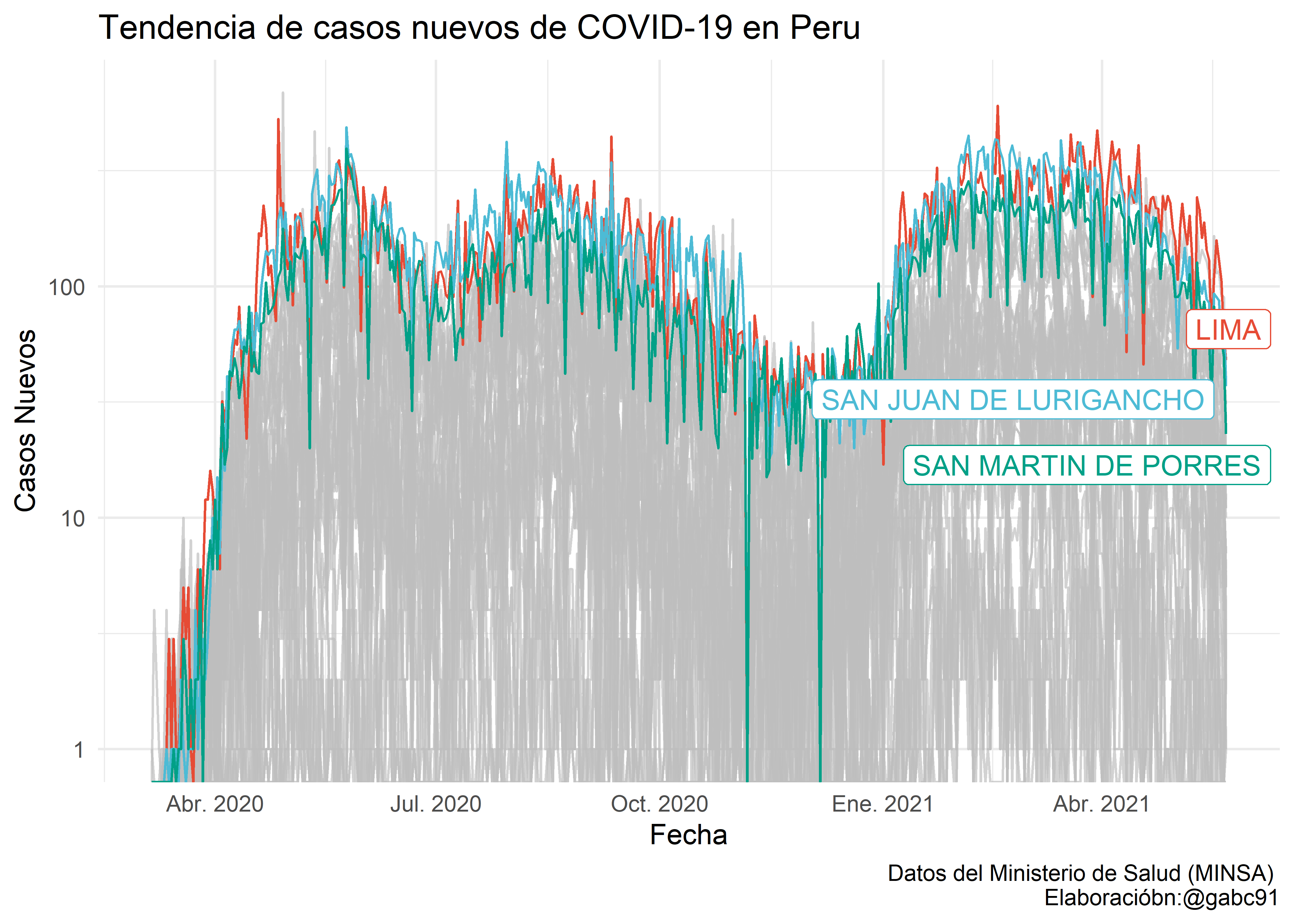
7.2.3 Gráfico de matrices
Otra representación muy utilizada para explorar diferencias entre diferentes subgrupos son las matrices. Su comportamiento es similar a los gráficos de líneas estratificado con la diferencia que los variables en los ejes contienen valores categoricos o numéricos discretos. A este tipo de gráficos también se le conoce como Heatmaps o mapas de calor.
Emepzaremos construyendo una base de datos que agrupe los datos por DISTRITO y semana. Utilizaremos la función
week()del paquetelubridatepara crear una variable que indique la semana del año.
En una matriz, cada dato estará mapeado a un par de coordenadas compuestas por la variable en el
eje xy en eleje y. La variable de interés (en este ejercicio el número de casos de COVID-19 -casos) será mapeada como un atributo que puede ser el color, tamaño, forma, etc.
Estas matrices pueden crearse con diferentes tipos de geometrías. En este ejercicio exploraremos el uso de geom_point() para crear una matríz de puntos y de geom_tile() para crear un mosaico. Al igual que en el gráfico de líneas, represntaremos la tendencia de casos de COVID-19 por DISTRITO.
7.2.3.1 Matrices de Puntos
Empezaremos con una matriz de puntos donde la variable de interés será representada por el atributo color.
covid_week %>%
ggplot(aes(x = year_week, y = DISTRITO, col = casos)) +
geom_point()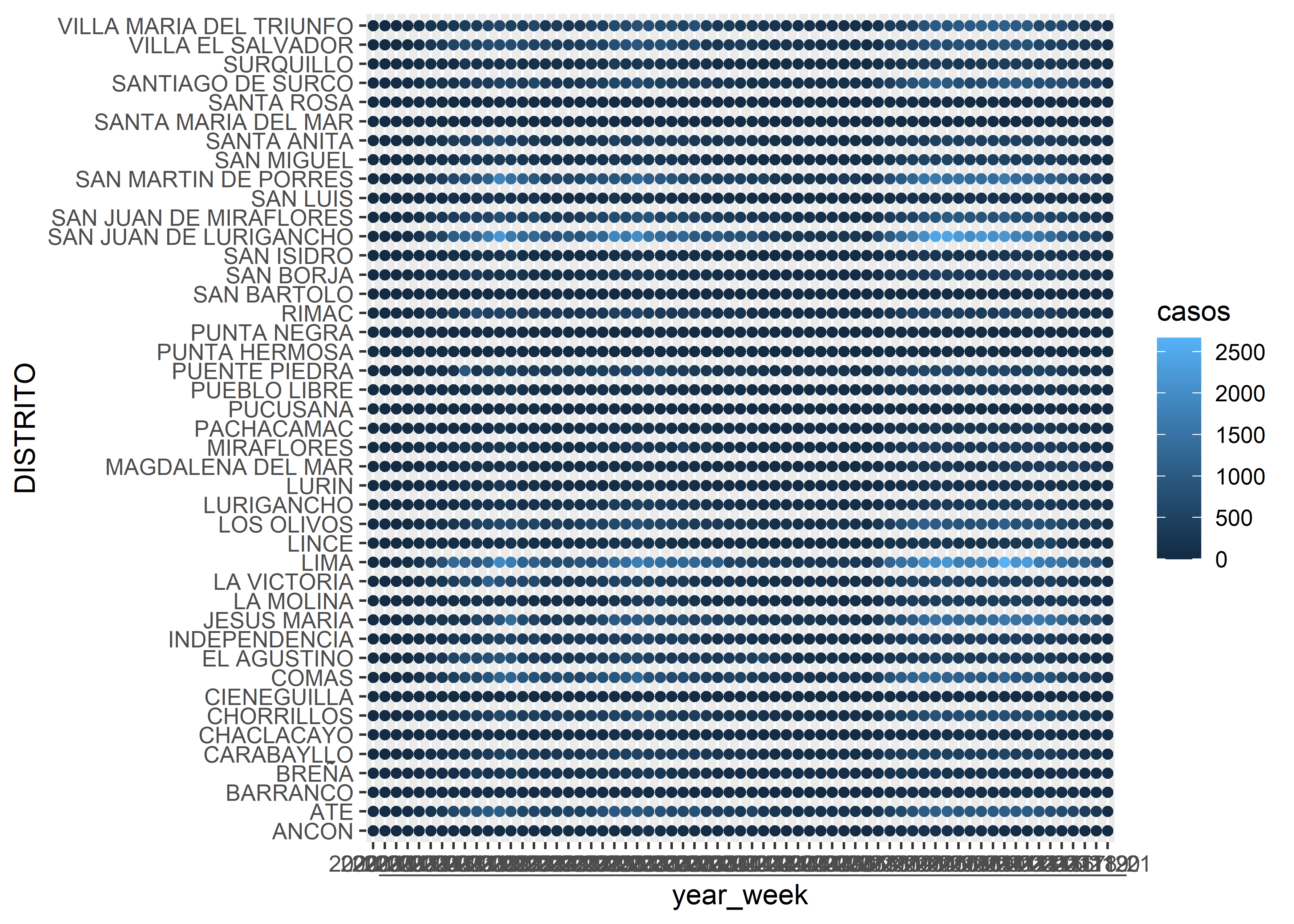
Podemos cambiar el atributo en la estética para representar la variable de interés de acuerdo al tamaño del punto.
covid_week %>%
ggplot(aes(x = year_week, y = DISTRITO, size = casos)) +
geom_point()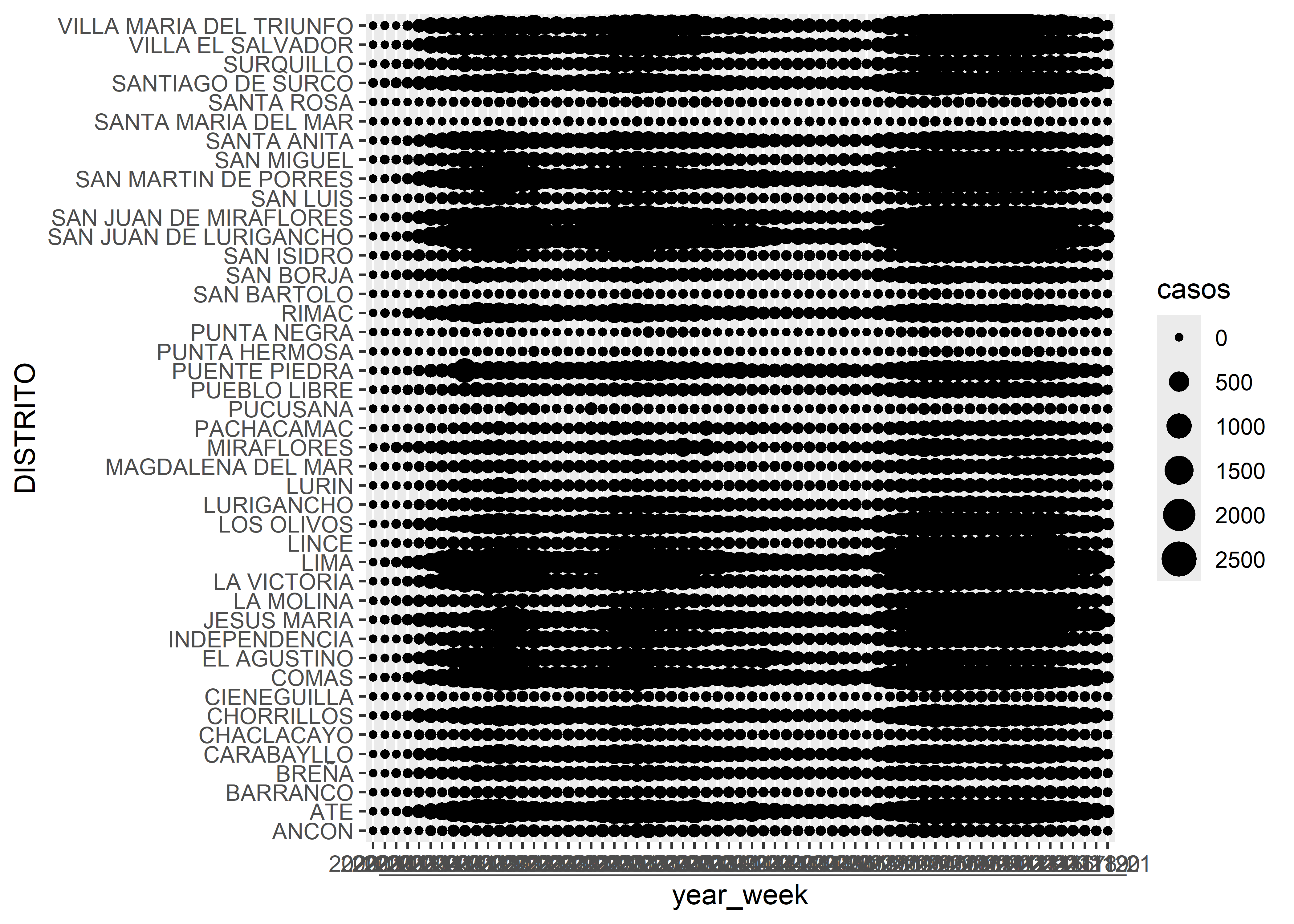
Incluso, podemos combinar los 2 atributos para tener una mejor representación de las tendencias.
covid_week %>%
ggplot(aes(x = year_week, y = DISTRITO, col = casos, size = casos)) +
geom_point()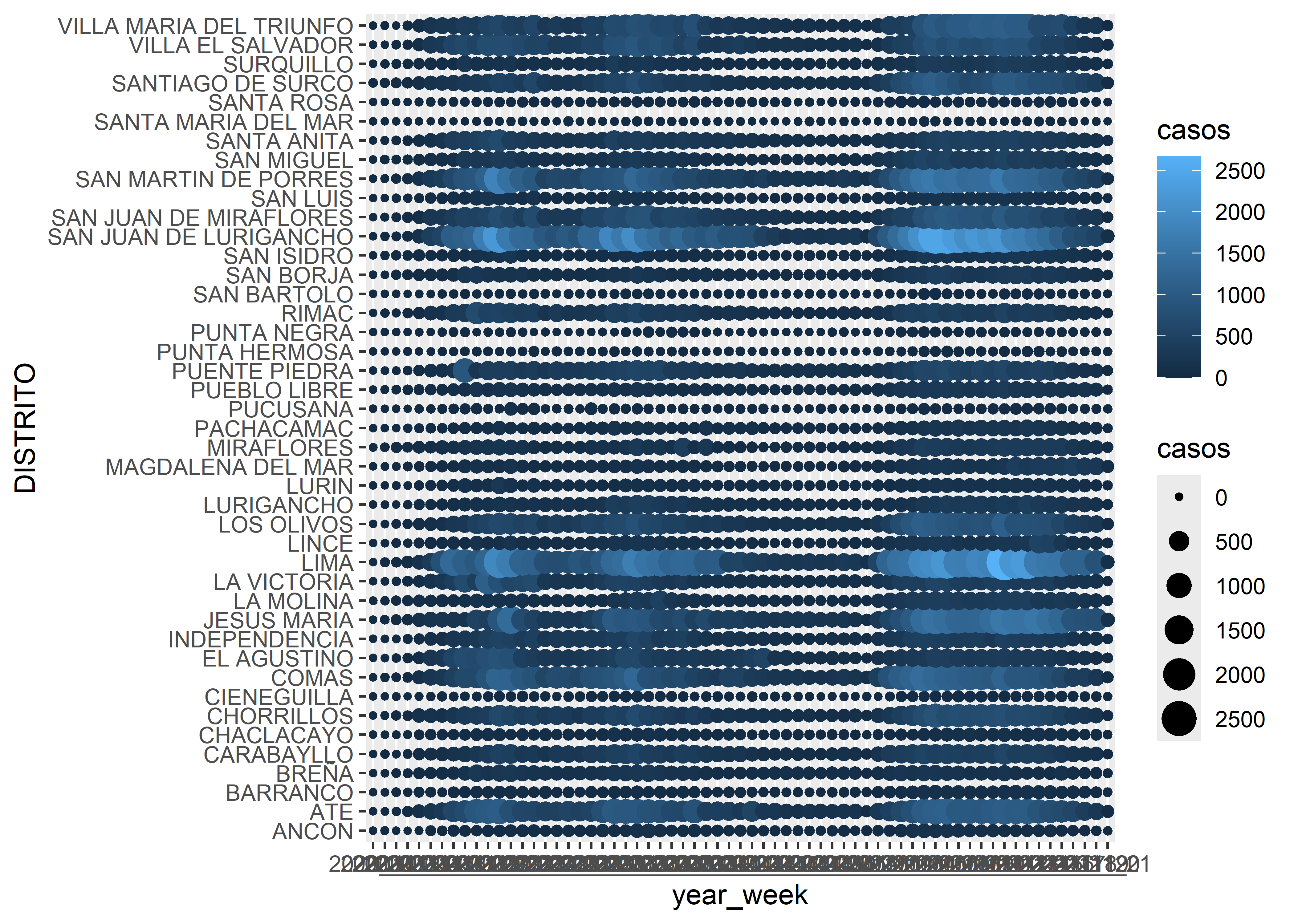
Finalmente, podemos utilizar las escalas, temas y facetas para mejorar la representación de nuestro gráfico.
covid_week %>%
ggplot(aes(x = year_week, y = DISTRITO, col = casos, size = casos)) +
geom_point() +
scale_color_viridis_c(option = "rocket") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 90))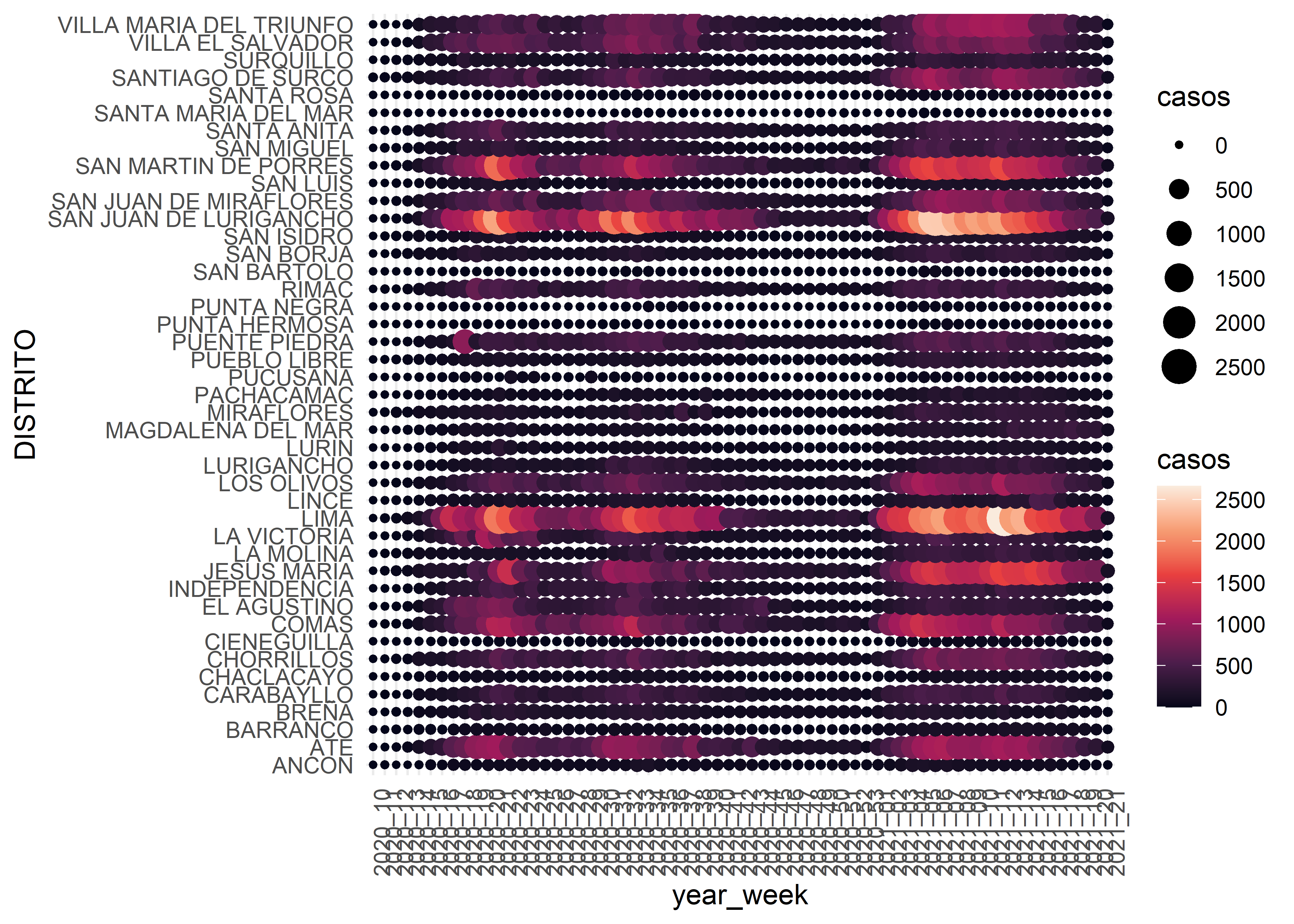
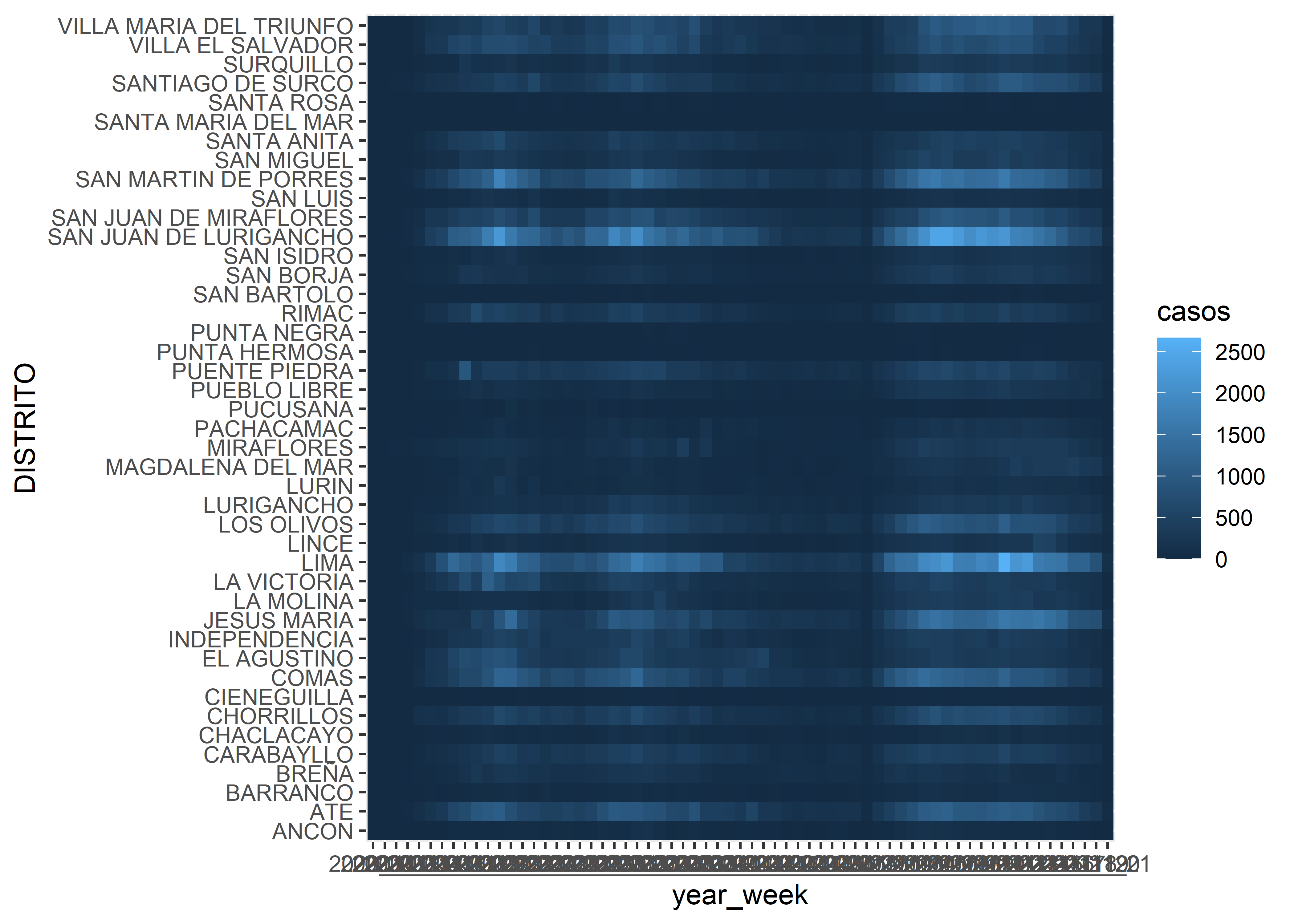
7.2.3.2 Mosaico o Heatmap
Podemos representar los mismos datos en formato mosaico con geom_tile().
Podemos, adicionalmente, ordenar el eje y donde estan los DISTRITOS en base al número total de casos (y otro criterio de ordenamiento).
covid_week %>%
ggplot(aes(x = year_week, y = fct_reorder(DISTRITO, casos, .fun = sum),
fill = casos)) +
geom_tile()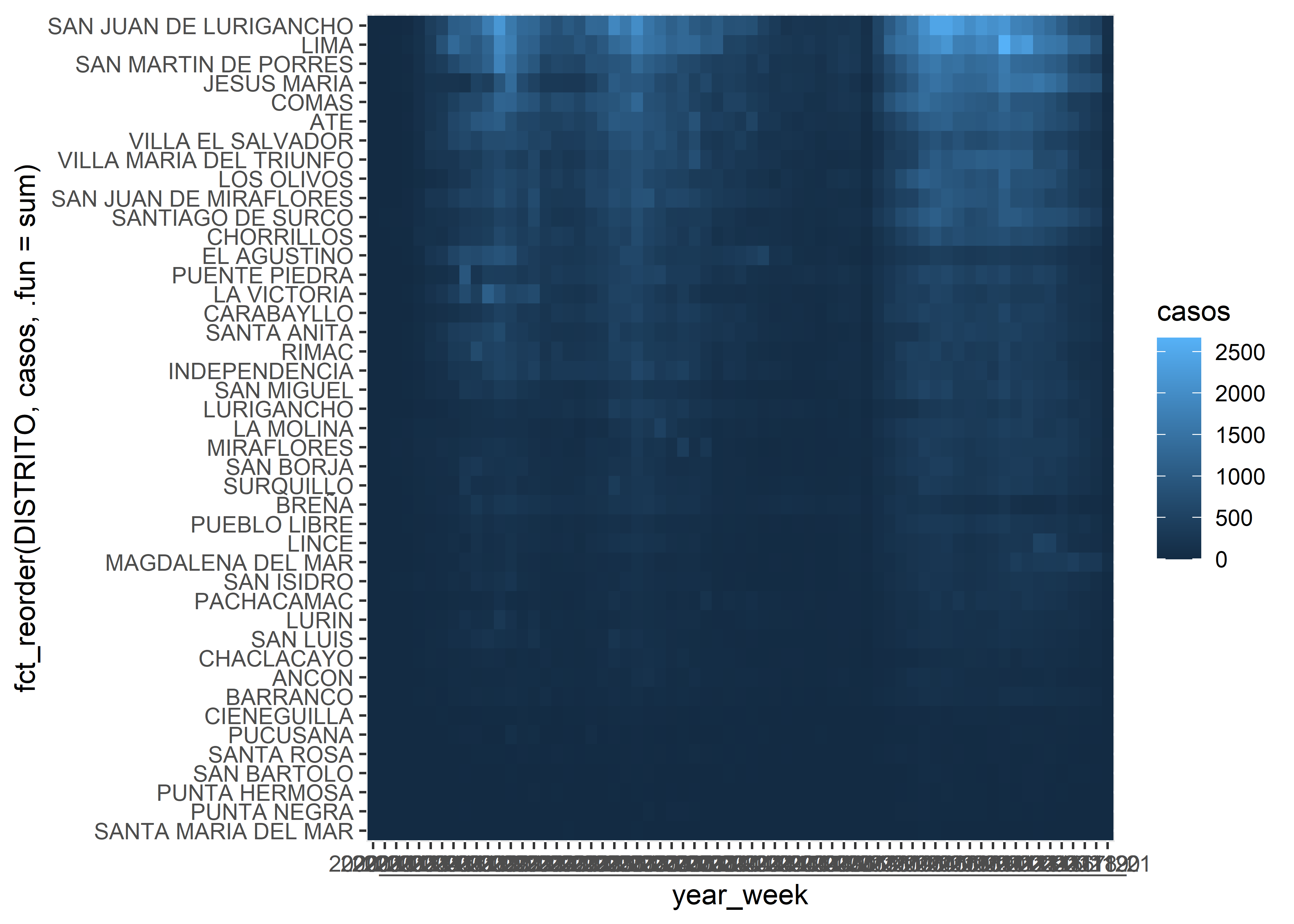
De la misma forma, podemos utilizar las escalas, temas y facetas para mejorar la representación de nuestro gráfico.
covid_week %>%
ggplot(aes(x = year_week, y = fct_reorder(DISTRITO, casos, .fun = sum),
fill = casos)) +
geom_tile() +
scale_fill_viridis_c(option = "rocket") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 90))
En estos gráficos podemos observar las mismas tendencias que en el gráfico de líneas pero de una forma más ordenada y que permite una mejor comparación entre DISTRITOS.
7.3 Graficos dinámicos
Finalmente, revisaremos algunos paquetes de gráficos dinámicos para complementar las representaciones gráficas que hemos mostrado hasta el momento. Al igual que ggplot, plotly es una libreria gráfica muy popular que permite la interacción del usuario con la visualización de datos. plotly es multiplataforma, es decir, tambien puede codificarse en R, python y JavaScript.
Los graficos de plot_ly nos permiten manipularlos de forma interactiva en conjunto a una barra de herramientas

Esta barra nos permite:
- Descargar el plot como .png

- Utilizar puntero de zoom

- Utilizar puntero de movimiento

- Resaltar un area del grafico en un cuadrilatero

- Resaltar un area de forma libre

- Seleccionando las categorias en la seccion derecha
7.3.1 Prueba de motor gráfico
library(plotly)
plot_ly(z = ~volcano) %>%
add_surface()7.3.2 Grafico de dispersion
Los argumentos que utiliza plot_ly para realizar el grafico de dispersion son:
- ‘data’: Indica la base de datos, en este caso ‘who’
- ‘x’: Datos para el eje x
- ‘y’: Datos para el eje y
7.3.3 Funcion layout
Añadimos un color a cada categoria agregando la opcion ‘color’.
Si queremos colores especificos, los podemos especificar en orden de la siguiente forma:
- Primero creamos un vector con los colores que queremos, el cual llamaremos ‘colors’
colors <- pal_npg("nrc")(7)y lo añadimos con la opcion ‘colors’ a plot_ly
Para modificar el nombre de los ejes, empezamos por el tipo de letra con los atributos:
- ‘family’ para el tipo de font
- ‘size’ para el tamaño
- ‘color’ para el color
Lo añadimos en una lista que nombraremos ‘f’.
f <- list(
family = "Arial",
size = 18,
color = "#7f7f7f"
)Ahora, creamos las listas ‘x’ e ‘y’ en las que indicaremos:
- ‘title’ para el nombre de cada eje
- ‘titlefont’ con el que llamaremos a la lista ‘f’, para indicar el estilo de letra.
Lo añadimos en ‘plotly’ en conjunto a la funcion ‘layout’ a traves de un pipe ‘%>%’
7.3.4 Etiquetas
Complementamos el grafico con la informacion de emision de CO2 con ‘size’
Para definir la informacion que aparece al pasar el puntero encima de cada punto, lo definimos con ‘text’ y ‘paste’. En este caso añadiremos la cantidad de CO2 y el nombre del pais para cada observacion.
7.3.5 3D
Para hacer un grafico en 3 dimensiones debemos agregar la variable en el eje z
7.3.6 De ggplot2 a plotly
Utilizaremos la funcion ggplotly para transladar nuestro grafico estático hecho con ggplot2 a uno dinamico con plotly
Utilizaremos nuestro grafico de la
Seccion 7.2.3.1: Matrices de Puntos.
fig <- covid_week %>%
ggplot(aes(x = year_week, y = DISTRITO, col = casos, size = casos)) +
geom_point() +
scale_color_viridis_c(option = "rocket") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 90))
fig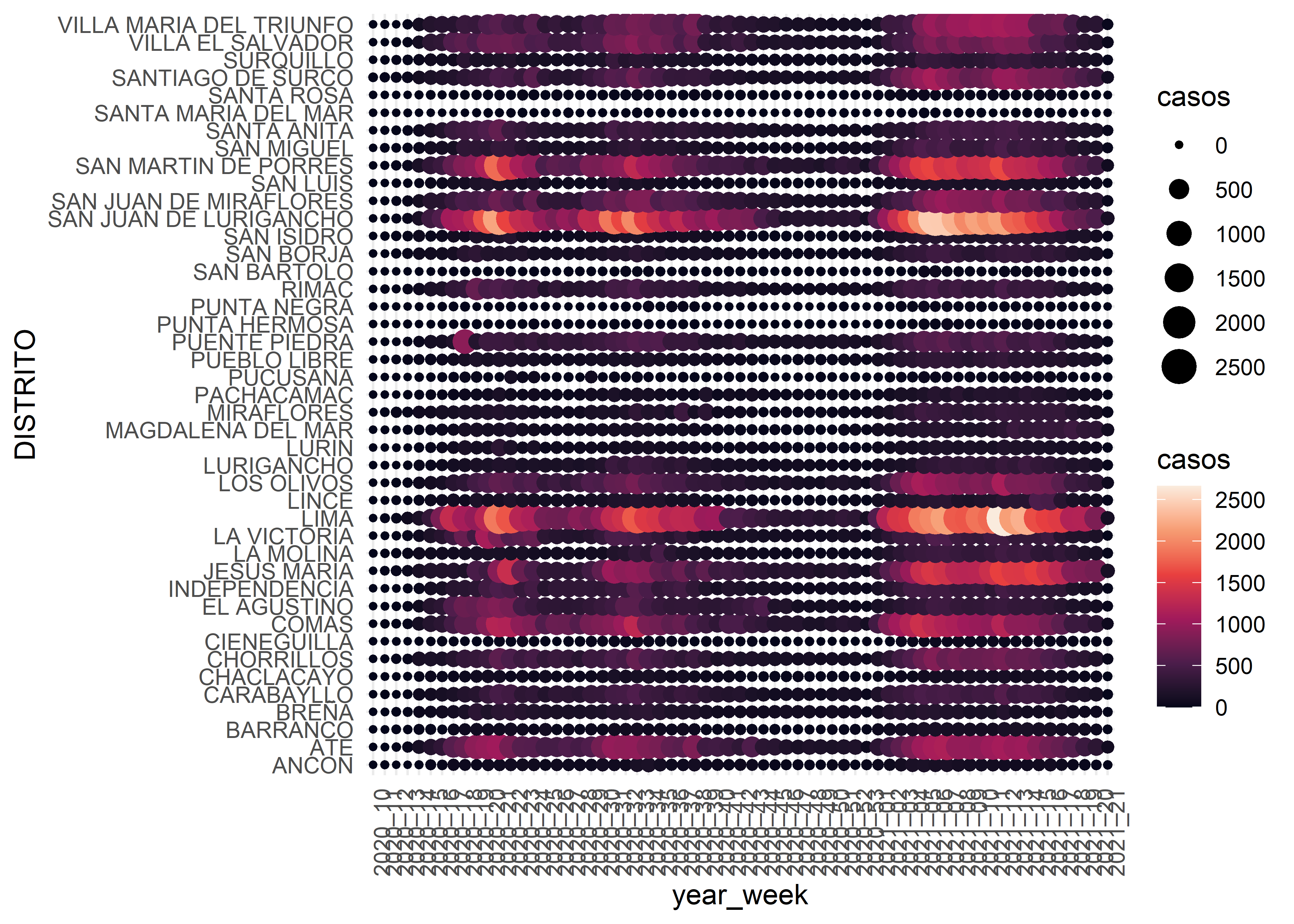
7.4 Ejercicios
Ejercicio 1
Con la base de datos covid_count realizar un gráfico de series de tiempo para los distritos que tienen una mediana de casos (durante todo el periodo de estudio) entre 50 y 80 casos.
Utilizar el formato tradicional filtrando los distritos de interés y usando
gghighlight.
Ejercicio 2
Crear un mapa estático (composición gráfica) y dinámico (usando mapview) de áreas administrativas (distritos) comparando el total de casos del 01 de Mayo del 2020 y el 01 de Mayo del 2021 para los grupos de menores de 50 años y de 50 a más años edad.