
3 tidyverse I: dplyr y pipelines
Hasta el momento hemos utilizado solo comandos de base, es decir, comandos que vienen dentro del funcionamiento básico de R.
La filosofía de tidy data nace del libro “R for Data Science” de Wickham y Grolemund (2016).
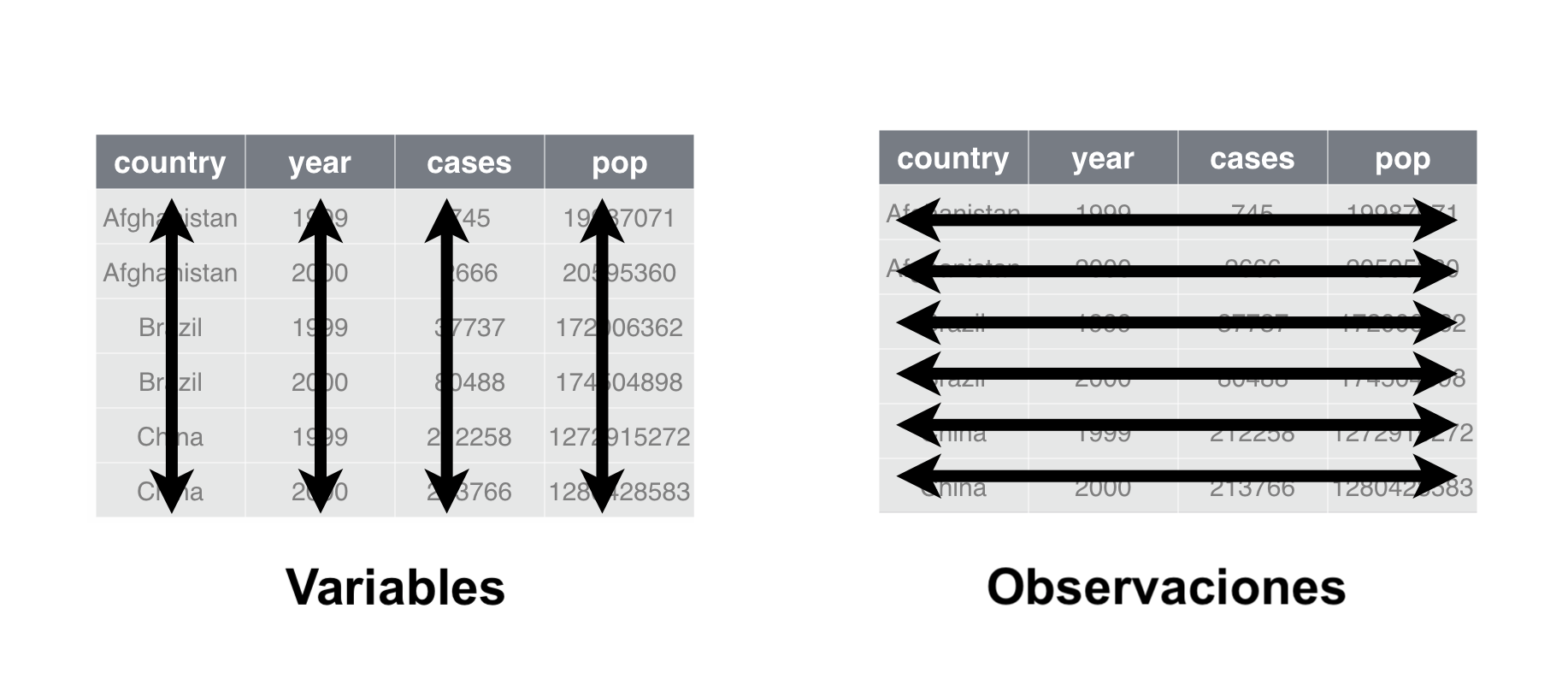
Tidy data es data tabular que está organizada de la siguiente manera:
- Cada columna es una (única) variable
- Cada fila es una (única) observación
tidyverse es una colección de paquetes de R diseñados para la ciencia de datos. Todos estos paquetes comparten la misma filosofía, por lo que aplican las mismas reglas de sintaxis sobre todos estos paquetes.
Los paquetes de tidyverse son:
- Optimizados para correr mas rápido en C++
- Mantenidos por un staff pagado de desarrolladores
- Bien documentados.
Existe más de 25 paquetes dentro del tidyverse y conforman un ecosistema para importar, estructurar, visualizar, modelar y comunicar datos.
Estos paquetes pueden ser cargados de forma individual. Sin embargo, el paquete tidyverse permite cargar 9 paquetes considerados el core de tidyverse:
- dplyr
- ggplot2
- tidyr
- tibble
- purrr
- readr
- stringr
- forcats
- lubridate
Estos paquetes son considerados el core de tidyverse porque son los paquetes más usados y generalmente son usados de forma conjunta. Sin embargo, hay un universo más amplio de paquetes que utilizan esta filosofía:

3.1 Paquetes y data
En esta sección utilizaremos las funciones del paquete tidyverse para el procesamiento de datos. A partir de ahora, para la lectura de los datos usaremos la función read_csv(), la cual es la versión tidy de la función base read.csv(). La principal diferencia entre read_csv() y la función base es que read_csv() devuelve un data frame en formato tibble, el cual es el formato estándar en el universo de tidyverse. Otra diferencia importante es que read_csv() es más rápido para leer conjuntos de datos grandes.
Cargamos el paquete tidyverse con el siguiente código:
Ahora, leeremos el archivo de datos a utilizar:
who <- read_csv("data/who.csv")- El conjunto de datos
whocontiene datos del Global Health Observatory de la Organización Mundial de la Salud (OMS). Contiene 359 variables para 202 países y territorios. El diccionario de este conjunto de datos se encuentra en Anexos.
3.2 Paquete dplyr
Comenzaremos utilizando algunas de las funciones más populares de la paquete dplyr.
3.2.1 Verbo select()
Usaremos la función select() para seleccionar las variables (o columnas) de interés. Seleccionaremos las variables país (country), gasto público en salud (% del PBI) (health_expenditure_gdp) y la expectativa de vida al nacer (life_expectancy_birth)
who_vars <- select(who, country, health_expenditure_gdp,
life_expectancy_birth)Podemos usar el operador : para establecer que queremos seleccionar desde la variable infant_mortality_rate hasta la variable infant_mortality_rate_males como si se tratase de una lista.
who_vars2 <- select(who, infant_mortality_rate:infant_mortality_rate_males)3.2.2 Verbo filter()
La función filter() nos permite filtrar observaciones que cumplen una condición de interés.
La condición puede definirse en base a variables categóricas:
who_africa <- filter(who, continent == "Africa")O en base a variables numéricas:
En este ejemplo filtraremos todos los paises que tienen una población de más de 10 millones
who_10m <- filter(who, population > 10000000)Podemos incluir también un rango de filtración. Por ejemplo tener solo países que tengan entre 5 y 10 millones de habitantes:
Así como también filtrar por múltiples criterios:
who_ame10m <- filter(who, continent == "South America",
population > 10000000)Podemos usar la función drop_na() del paquete tidyr, el cual es un operador lógico que crea un valor de TRUE si la observación no tiene algún valor NA en las variables del conjunto de datos y FALSE en el caso contrario.
En este ejemplo, filtraremos todas las observaciones que tienen datos completos en todas las variables del conjunto de datos.
who_complete <- drop_na(who)También podemos “filtrar” las observaciones que no estén completas en variables seleccionadas (infant_mortality_rate :infant_mortality_rate_males).
who_complete2 <- drop_na(who, infant_mortality_rate:infant_mortality_rate_males)Otros operadores útiles que pueden ser usados junto con la función filter():
3.2.3 Verbo mutate()
La función mutate() nos permite realizar operaciones con las variables y almacenar los resultados en la misma o una nueva variable.
Podemos calcular la población rural como la diferencia entre la población total (population) y la población urbana (urban_population) por país.
who_rural <- mutate(who, rural_population = population - urban_population) En este caso, al realizar este cálculo en esta base de datos algunas poblaciones rurales son negativas. Esto puede deberse, entre otras cosas, a una mala digitación de la base, a una corrupción de los datos durante su almacenamiento o escritura, o a la fuente de los datos (ej. la población total puede tener un origen en un censo y la población urbana puede ser una estimación en base a un modelo).
También podemos utilizar la función if_else() para definir reglas de decisión:
También podemos categorizar una variable continua usando la función ntile() para crear percentiles:
En el caso que se quisiera crear una variable a partir de más de una condición, podemos usar la función case_when(). Por ejemplo, si quisiéramos crear las categorías de alfabetismo Baja, Moderada, Alta y Muy Alta, con case_when() sería de la siguiente manera:
En case_when(), para indicar “en cualquier otro caso”, se usará el argumento .default. Por ejemplo, se podría no haber mencionado la última condición y señalar que cualquier otro caso será “Muy alta”.
Recientemente se añadió la función
case_match()para cuando se necesite hacer múltiples comparaciones, se evite mencionar constantemente el nombre de variable como sucede conliteracy_rateen el anterior ejemplo.
3.2.4 Verbo summarise()
La función summarise() nos permite obtener medidas de resumen de las variables en el conjunto de datos.
Debido a que la variable
populationtiene datos vacíos (NA), se utiliza el argumentona.rm = Tpara excluirlos del cómputo de las medidas de resumen.
Se puede definir más de una medida de resumen.
Otras medidas de resumen del paquete básico de R que pueden ser usadas en conjunto con summarise() son:
Otras funciones que incluye dplyr:
3.2.5 Verbo group_by()
La función group_by() nos permite crear grupos de observaciones los cuales se identifican por una o más variables.
who_group <- group_by(who, continent)A simple vista, podría parecer que no ha ocurrido nada; sin embargo, podemos observar que ahora el dataframe muestra el siguiente atributo (attr): “groups”.
Usualmente, esta función es usada en conjunto con summarise para obtener las medidas de resumen por grupos.
Se puede usar más de una variable para la agrupación. Calcularemos el total de población y promedio de esperanza de vida al nacer por cada continente (continent) y categoría de alfabetismo (literacy_cat) usando el conjunto de datos who_literacy_cat donde hemos creado las categorías.
Para eliminar la agrupación podemos utilizar la función
ungroup()
3.2.6 Verbo arrange()
La función arrange() nos permite ordenar las filas del conjunto de datos con respecto a una o más variables.
Ordenaremos el conjunto de datos who en función a la población total. Utilizamos la función desc() para especificar si queremos que sea de forma descendiente.
3.2.7 Verbo sample_n()
La función sample_n() nos permite crear una muestra aleatoria de nuestro conjunto de datos. Debemos especificar el tamaño (size) de la muestra.
who_sample <- sample_n(who, size = 30)3.3 Inspección de datos
Existen varias formas de inspeccionar de forma rápida nuestro conjunto de datos. Aquí algunos paquetes para realizarlo:
3.3.1 Codebook
Para explorar las variables de nuestro conjunto de datos de forma mas amigable podemos utilizar el paquete codebook (Arslan 2020), lanzado en enero del 2020. Este paquete tiene una interfaz gráfica que permite buscar y ver metadatos y medidas de resumen de las variables.
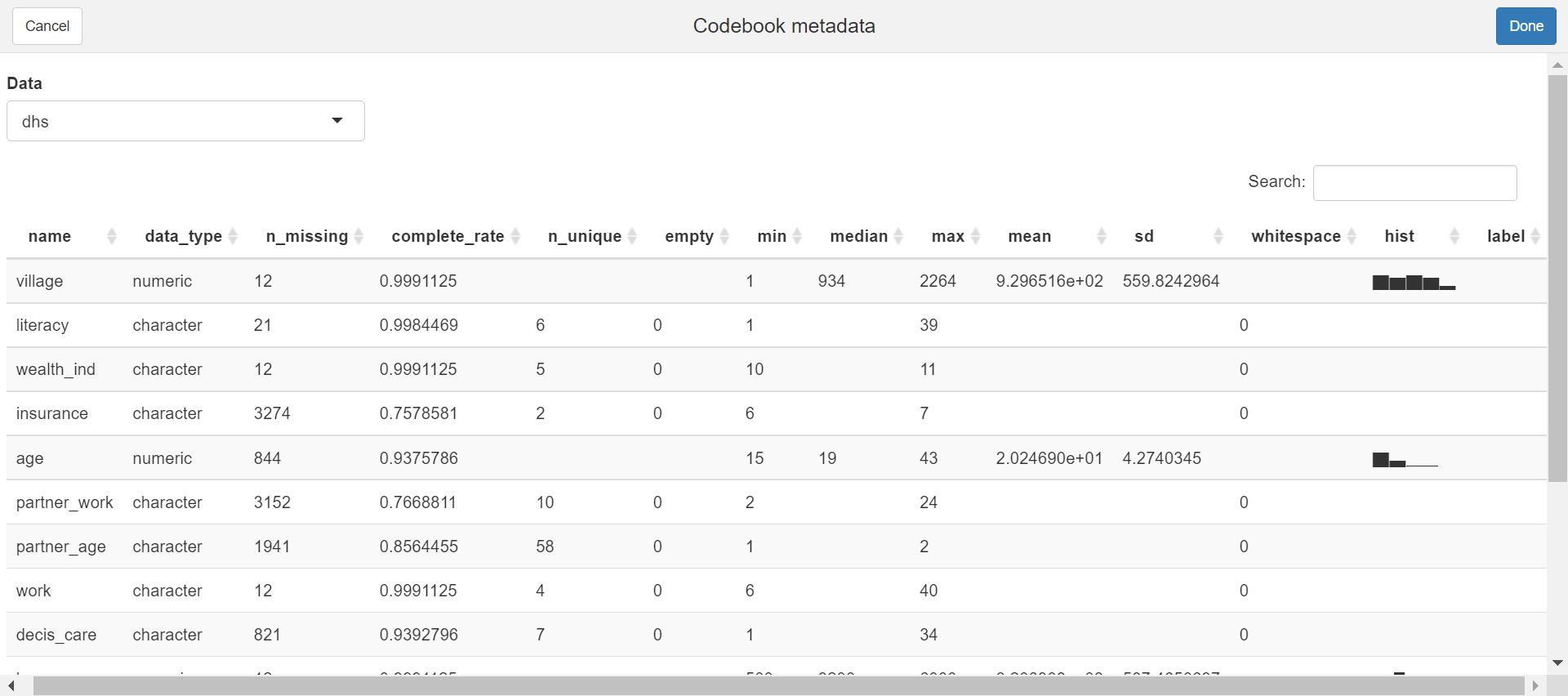
Cuando el archivo de datos es importado de un archivo de STATA, SPSS o SAS y contiene metadatos como nombre de las variables, etiqueta de valores, etc., estos podrán ser visualizados directamente con codebook.
3.3.2 Skimr
Otro paquete útil para una inspección rápida es skimr, que cumple con los Principles of Least Surprise (POLA).
| Name | who |
| Number of rows | 202 |
| Number of columns | 26 |
| _______________________ | |
| Column type frequency: | |
| character | 2 |
| numeric | 24 |
| ________________________ | |
| Group variables | None |
Variable type: character
| skim_variable | n_missing | complete_rate | min | max | empty | n_unique | whitespace |
|---|---|---|---|---|---|---|---|
| continent | 0 | 1 | 6 | 25 | 0 | 7 | 0 |
| country | 0 | 1 | 4 | 41 | 0 | 202 | 0 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| continent_id | 0 | 1.00 | 3.58 | 1.81 | 1.00 | 2.00 | 3.00 | 5.00 | 7.00 | ▇▅▁▃▅ |
| country_id | 0 | 1.00 | 101.50 | 58.46 | 1.00 | 51.25 | 101.50 | 151.75 | 202.00 | ▇▇▇▇▇ |
| population | 28 | 0.86 | 34679451.49 | 131601389.69 | 65365.00 | 2246777.50 | 7309125.50 | 22750000.00 | 1300000000.00 | ▇▁▁▁▁ |
| urban_population | 14 | 0.93 | 16657626.77 | 50948665.82 | 15456.00 | 917162.34 | 3427660.80 | 9837113.25 | 527000000.00 | ▇▁▁▁▁ |
| pop_under_poverty_line | 130 | 0.36 | 16.02 | 18.71 | 2.00 | 2.00 | 7.45 | 23.05 | 70.80 | ▇▂▁▁▁ |
| life_expectancy_birth | 34 | 0.83 | 67.47 | 11.31 | 40.68 | 59.47 | 71.33 | 76.02 | 82.27 | ▂▃▃▇▇ |
| inequality_index | 72 | 0.64 | 40.74 | 9.44 | 24.70 | 34.00 | 39.45 | 47.01 | 74.33 | ▆▇▅▂▁ |
| literacy_rate | 65 | 0.68 | 77.98 | 20.58 | 23.55 | 67.45 | 84.68 | 93.83 | 99.80 | ▁▂▂▃▇ |
| literacy_rate_males | 66 | 0.67 | 82.68 | 17.08 | 31.44 | 74.00 | 88.32 | 95.93 | 99.81 | ▁▁▂▃▇ |
| literacy_rate_females | 70 | 0.65 | 83.31 | 21.82 | 16.86 | 71.08 | 95.46 | 98.84 | 99.95 | ▁▁▁▂▇ |
| co2_economic_output | 29 | 0.86 | 0.42 | 0.35 | 0.01 | 0.19 | 0.31 | 0.50 | 2.81 | ▇▂▁▁▁ |
| gni_per_capita | 24 | 0.88 | 11250.11 | 12586.75 | 260.00 | 2112.50 | 6175.00 | 14502.50 | 60870.00 | ▇▂▁▁▁ |
| health_expenditure_gdp | 22 | 0.89 | 3.63 | 2.16 | 0.26 | 1.96 | 3.19 | 4.68 | 11.73 | ▇▇▃▂▁ |
| health_expenditure_person | 22 | 0.89 | 693.04 | 1312.98 | 0.23 | 33.25 | 157.00 | 486.75 | 6657.00 | ▇▁▁▁▁ |
| hospital_beds | 22 | 0.89 | 32.17 | 25.09 | 1.00 | 12.00 | 26.00 | 48.25 | 141.00 | ▇▅▂▁▁ |
| children_out_school_primary | 38 | 0.81 | 360414.70 | 1109033.85 | 37.00 | 8656.50 | 34216.50 | 223811.00 | 8096824.00 | ▇▁▁▁▁ |
| expenditure_student_primary | 55 | 0.73 | 14.59 | 6.90 | 0.91 | 9.37 | 14.07 | 19.19 | 37.26 | ▃▇▅▂▁ |
| malnutrition_weight_age | 101 | 0.50 | 16.79 | 11.77 | 1.10 | 6.10 | 15.30 | 24.40 | 47.60 | ▇▅▃▂▂ |
| infant_mortality_rate | 9 | 0.96 | 38.04 | 38.15 | 2.00 | 9.00 | 23.00 | 59.00 | 165.00 | ▇▂▂▁▁ |
| infant_mortality_rate_females | 9 | 0.96 | 34.90 | 35.62 | 2.00 | 9.00 | 20.00 | 54.00 | 154.00 | ▇▂▂▁▁ |
| infant_mortality_rate_males | 9 | 0.96 | 41.01 | 40.77 | 3.00 | 9.00 | 24.00 | 63.00 | 176.00 | ▇▂▂▁▁ |
| under_5_mortality_rate | 141 | 0.30 | 114.94 | 57.72 | 29.60 | 69.40 | 111.00 | 157.40 | 253.20 | ▇▇▅▅▁ |
| maternal_mortality_rate | 33 | 0.84 | 322.37 | 421.09 | 1.00 | 15.00 | 130.00 | 510.00 | 2100.00 | ▇▂▁▁▁ |
| tb_treatment_success | 25 | 0.88 | 77.59 | 14.76 | 0.00 | 71.00 | 80.00 | 87.00 | 100.00 | ▁▁▁▆▇ |
3.4 Pipelines
Ahora que ya tenemos un mayor conocimiento de las funciones básicas y elementales que necesitamos para el manejo de datos, volveremos a ver las tres alternativas explicadas inicialmente, para observar el impacto que tiene el uso de pipes (%>%) en la escritura y lectura de código. El objetivo de la rutina de análisis será quitar los valores perdidos que se encuentren en las variables population, life_expectancy_birth y literacy_rate de la data who, dividir la tasa de alfabetización en cinco grupos, seleccionar algunas variables de importancia, y configurar análisis por agrupación de continente y grupo de alfabetización, para posteriormente obtener un resumen de la cantidad de casos, total de población y promedio de esperanza de vida. Finalmente, estos resultados serán ordenados de mayor a menor población total y solo se mostrará los casos en que el promedio de la esperanza de vida sea mayor a 70.
3.4.1 Objetos múltiples
Observaremos que ahora, debido a la cantidad de procesos intermedios que tenemos que realizar, la cantidad de objetos a crear es considerablemente mayor.
who_complete <- drop_na(who, population, life_expectancy_birth, literacy_rate)
who_literacy <- mutate(who_complete,
literacy_cat = ntile(literacy_rate, 5))
who_vars <- select(who_literacy, continent, country, population, life_expectancy_birth, literacy_cat)
who_group <- group_by(who_vars, continent, literacy_cat)
who_summ <- summarise(who_group,
n = n(),
pop_tot = sum(population),
m_life_exp = mean(life_expectancy_birth))
who_ord <- arrange(who_summ, desc(pop_tot))
who_final <- filter(who_ord, m_life_exp > 70)En un proyecto real es probable que las manipulaciones requeridas sean incluso mayores y en múltiples conjuntos de datos, por lo que esta aproximación no es ideal.
3.4.2 Funciones anidadas
Otra opción es el uso de funciones anidadas, es decir, usar funciones dentro de otras funciones, tal y como se muestra en el ejemplo:
who_nested <- filter(
arrange(
summarise(
group_by(
select(
mutate(
drop_na(who,
population, life_expectancy_birth, literacy_rate),
literacy_cat = ntile(literacy_rate, 5)
),
continent, country, population, life_expectancy_birth, literacy_cat
),
continent, literacy_cat
),
n = n(),
pop_tot = sum(population),
m_life_exp = mean(life_expectancy_birth)
),
desc(pop_tot)
),
m_life_exp > 70
)Aunque ya no se crean múltiples objetos, su lectura, escritura y depuración siguen siendo un problema importante al momento de codificar.
3.4.3 Operador pipe (%>%)
Como ya lo mencionamos, el uso del operador pipe (%>%) nos permite realizar análisis con una secuencia que se siente más “natural”.
who_pipe <- who %>%
drop_na(population, life_expectancy_birth, literacy_rate) %>%
mutate(literacy_cat = ntile(literacy_rate, 5)) %>%
select(continent, country, population,
life_expectancy_birth, literacy_cat) %>%
group_by(continent, literacy_cat) %>%
summarise(
n = n(),
pop_tot = sum(population),
m_life_exp = mean(life_expectancy_birth)
) %>%
arrange(desc(pop_tot)) %>%
filter(m_life_exp > 70)En los tres casos, los conjuntos de datos que se generan son idénticos:
identical(who_pipe, who_nested)[1] TRUEidentical(who_pipe, who_final)[1] TRUEidentical(who_nested, who_final)[1] TRUERecordemos que el operador (%>%) puede leerse como “y luego… / entonces”. En nuestro ejemplo, estaríamos diciendo:
- “Del conjunto de datos
who, eliminar las observaciones con datos incompletos de las variablespopulation,life_expectancy_birthyliteracy_rate, luego crear una variable con los quintiles de tasa de alfabetismo (literacy_rate), luego seleccionar las variables de interés, luego agrupar las observaciones (paises) de acuerdo a los continentes y quintiles de alfabetismo, luego calcular estas medidas de resumen (total de paises, suma de población y promedio de esperanza de vida) para cada grupo, luego ordenar las observaciones con respecto a la suma de la población en orden decreciente y luego filtrar aquellos grupos con un promedio de esperanza de vida mayor a 70%. ”
También es importante mencionar que, al usar el operador pipe (%>%), solo se necesita mencionar el dataframe del cual se parte en el inicio del pipe.
3.5 Ejercicios
3.5.1 Parte 1
Ejercicio 1
Realizar las siguientes operaciones secuenciales sobre la data who:
- Seleccionar las variables continente (
continent), país (country), población (population) y la tasa de mortalidad materna por cada 100000 nacimientos vivos (maternal_mortality_rate). - Luego, filtrar los países que tienen una población mayor a 30 millones.
- Por último, ordenar el conjunto de datos según la tasa de mortalidad materna de menor a mayor.
Una vez que se identifique las funciones necesarias para resolver los 3 puntos, el código debería ser:
# A tibble: 34 × 3
country population maternal_mortality_rate
<chr> <dbl> <dbl>
1 Democratic Republic of the Congo 60800000 1100
2 Nigeria 129000000 1100
3 United Republic of Tanzania 36800000 950
4 Ethiopia 73100000 720
5 Bangladesh 144000000 570
6 Kenya 33800000 560
7 India 1080000000 450
8 Sudan 40200000 450
9 Indonesia 218000000 420
10 South Africa 44300000 400
# ℹ 24 more rowsEjercicio 2
Usar el conjunto de datos resultante del ejercicio anterior para crear una nueva variable nivel con la función case_when() definiendo las siguientes categorías:
- Los países con una tasa de mortalidad materna menor a 12 tienen una “Tasa de mortalidad materna baja”,
- los países con una tasa de mortalidad materna mayor a 415 tienen una “Tasa de mortalidad materna alta”.
- En otro caso, asignar una “Tasa de mortalidad materna media”.
Por último, seleccionar el país, la población y el nivel de la tasa de mortalidad. Las 3 primeras filas resultantes deberían ser las siguientes:
# A tibble: 34 × 3
country population nivel
<chr> <dbl> <chr>
1 Democratic Republic of the Congo 60800000 Tasa de mortalidad materna alta
2 Nigeria 129000000 Tasa de mortalidad materna alta
3 United Republic of Tanzania 36800000 Tasa de mortalidad materna alta
4 Ethiopia 73100000 Tasa de mortalidad materna alta
5 Bangladesh 144000000 Tasa de mortalidad materna alta
6 Kenya 33800000 Tasa de mortalidad materna alta
7 India 1080000000 Tasa de mortalidad materna alta
8 Sudan 40200000 Tasa de mortalidad materna alta
9 Indonesia 218000000 Tasa de mortalidad materna alta
10 South Africa 44300000 Tasa de mortalidad materna media
# ℹ 24 more rows3.5.2 Parte 2
Exploraremos ahora un nuevo conjunto de datos. El archivo de datos dhs.csv contiene información relacionada a la salud materno-infantil y condiciones sociodemográficas en Peru. En este conjunto de datos, cada observación (una madre que tiene un hijo dentro del periodo de estudio) esta agrupada en comunidades. Este conjunto de datos es una submuestra de la Encuesta Demográfica y de Salud Familiar (ENDES).
dhs <- read_csv("data/dhs.csv")Ejercicio 1
A) Describir en lenguaje natural qué procedimientos se están llevando a cabo en el siguiente código.
B) ¿Qué conclusiones podría sacar de este pipeline?
Ejercicio 2
A) Escribir el pipeline para la siguiente tarea de manejo de datos.
Del conjunto de datos dhs, filtrar las observaciones de las madres menores de 20 años, luego retirar todas las observaciones con valores ausentes (NA) en las variables edad, edad de pareja y seguro, luego calcular la diferencia entre la edad de la participante y la de su pareja, luego agrupar de acuerdo a si tienen seguro y acceso a agua potable segura, y luego calcular el promedio de hijos de las madres en cada una de estas categorías y la diferencia promedio de edad con sus parejas.
B) ¿Qué conclusiones podría sacar de este pipeline?